2. 济宁医学院附属医院肿瘤放疗科, 济宁 272200;
3. 济宁医学院临床学院, 济宁 272200
2. Department of Oncology Radiotherapy, Affiliated Hospital of Jining Medical University, Jining 272000, China;
3. Department of Clinical Medicine, Jining Medical University, Jining 272000, China
非小细胞肺癌约占所有原发性肺癌的75%~80%,其中肺腺癌是最常见的病理学类型之一[1-2]。随着诊断和手术技术的进步以及靶向治疗的发展,非小细胞肺癌患者的生存率及生活质量得到很大提高[3-6],其中表皮生长因子受体(epidermal growth factor receptor,EGFR)是肺腺癌患者最常见的突变基因,EGFR酪氨酸激酶抑制剂(tyrosine kinase inhibitors,TKIs)靶向治疗可以有效延长EGFR突变型肺腺癌患者的无进展生存期(progression free survival,PFS)[7-9]。目前,EGFR基因检测依赖手术切除或组织活检等侵入性方法,但这些方法耗时、昂贵,存在出血和气胸等风险,且不能实时描述肿瘤内异质性和肿瘤微环境等[10]。因此,开发一种无创的实时预测EGFR基因突变状态的方法至关重要。影像组学可以从医学影像中提取大量人眼难以量化的肿瘤异质性和基因学的信息[11-12]。既往研究表明,影像组学可用于预测非小细胞肺癌的EGFR突变状态[13-14],EGFR突变主要发生在肺腺癌中,鳞状细胞癌较少发生且靶向治疗效果亦不如腺癌[15-16],若鳞癌也纳入研究中,会产生一定的混杂偏倚。另外,一些基于平扫CT影像组学预测EGFR突变状态的研究中,其受试者工作特征(receiver operating characteristic,ROC)曲线下面积(area under the curve,AUC)为0.762~0.816[10, 17]。Yang等[18]、Hong等[19]发现,基于增强CT的影像组学预测EGFR突变能力好于非增强CT,AUC为0.842~0.851。但既往大部分研究为回顾性分析,图像多来自不同的CT设备,扫描参数不一致,在影像组学特征提取之前并未提及对CT图像进行标准化处理,这可能会降低影像组学特征的鲁棒性[19]。本研究探讨基于治疗前标准化胸部增强CT影像组学特征及临床特征开发的联合模型及列线图,预测肺腺癌EGFR基因突变状态的临床价值。
资料与方法1. 研究对象:回顾性收集济宁医学院附属医院2017年7月至2023年6月经病理证实的450例原发性肺腺癌患者的CT影像和临床资料。纳入标准如下:①明确的组织病理学诊断及EGFR基因突变状态(野生型和突变型)。②活检或术前1个月内胸部增强CT影像资料。③病灶直径>1 cm。④临床资料完整。排除标准:①无确诊前胸部增强CT。②确诊前曾行抗肿瘤治疗。③合并其他恶性肿瘤。④感兴趣区(region of interest,ROI)难以勾画,如合并阻塞性肺不张、大量胸腔积液等。⑤缺乏关键临床资料。⑥图像质量较差。本研究EGFR突变状态采用下一代基因测序(NGS)技术进行检测,样本为外周血和/或肿瘤组织,最终纳入262例肺腺癌患者,采用分层随机抽样方法按照7∶3比例分为训练组(n=183)和测试组(n=79)。本研究通过了医院伦理委员会批准(2023-03-C018)。
2. 设备及检查方法:采用美国GE Revolution CT或德国西门子双源CT进行胸部增强扫描。扫描参数:层厚0.5~1.5 mm;管电压100~120 kV;管电流自动;扫描范围自肺尖至肋膈角;采用自动高压注射器以3.0 ml/s的速度将非离子对比剂碘己基(350 mg/ml)注入肘前静脉行增强扫描。影像组学的工作流程包括:图像采集、预处理、ROI勾画、特征提取、特征筛选、模型构建和模型预测。
3. 图像预处理、ROI勾画和影像组学特征提取:CT图像以DICOM格式导入开源软件ITK-SNAP(版本3.8.0,http://www.itksnap.org),由于图像采集来自不同的CT设备,扫描参数不一致,在分割前对CT图像进行标准化预处理。标准化过程包括:①分辨率归一化,通过插值方法将体素重采样到标准尺寸(1 mm × 1 mm × 1 mm),减少不同体素尺寸对影像组学特征值的影响。②对比度归一化,通过高斯拉普拉斯变换(laplacian of gaussian,LOG)对图像进行平滑处理,降低噪声的影响。2名有5年以上胸部影像诊断工作经验且对患者信息不知情的医师,参照胸部增强CT的纵隔窗和肺窗沿肿瘤边界逐层手动勾画ROI并进行分割。通过“Onekey AI”平台(https://www.medai.icu) 分别提取定量标准化及未标准化影像组学特征,包括一阶特征、纹理特征(灰度共生矩阵特征GLCM、灰度依赖矩阵特征GLDM、灰度运行长度矩阵特征GLRLM、灰度大小区域矩阵特征GLSZM和相邻灰度色差矩阵特征NGTDM)、形状特征以及小波变换特征WAVELET。
4. 特征筛选和模型构建:为保证影像组学特征的稳定性和再现性,对每名患者的CT图像进行两次ROI分割和特征提取,使用组内相关系数(intraclass correlation coefficient,ICC)进行观察者间的一致性检验,对ICC > 0.80的特征进一步筛选。服从正态分布的特征使用独立样本t检验。用Pearson标准相关系数来计算特征之间的相关性,通过最小冗余最大相关算法(minimum redundancy maximum relevance,MRMR)删除最大冗余,降低冗余度。采用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)回归对特征进行降维,通过10倍交叉验证调整惩罚系数筛选出最优特征,计算影像组学特征分数(Rad-score)。收集临床基线特征包括性别、年龄、吸烟史、体质量指数(BMI)、临床分期、病灶位置及血清肿瘤标志物[如细胞角蛋白19片段(CYFRA21-1)、神经元特异性烯醇化酶(NSE)、癌胚抗原(CEA)]等。使用Logistic Regression(LR)机器学习方法分别建立临床模型、影像组学模型以及两者相结合的联合模型,并绘制列线图。
5. 模型性能预测及验证:绘制ROC曲线评估不同模型对肺腺癌EGFR基因突变状态的预测效能,并计算AUC、准确度、灵敏度、特异度、阳性预测值(positive predictive value,PPV)和阴性预测值(negative predictive value,NPV)。采用Delong检验比较模型间的差异性,利用决策曲线分析(decision curve analysis,DCA)量化不同阈值概率下的净收益,评估每种模型的临床有效性。
6. 统计学处理:采用“Onekey AI”平台(https://www.medai.icu)和SPSS 22.0软件进行统计分析,其中Onekey AI平台基于Pytorch 1.8.0。连续变量经正态性检验符合正态分布,以x±s表示。采用独立样本t检验对年龄、BMI及影像组学评分进行比较;分类变量以数值和百分数[n(%)] 表示,采用卡方检验对吸烟史、性别、临床分期、CEA、CYFRA21-1片段、NSE及病灶位置进行比较;预测模型组间比较采用Delong检验。P<0.05为差异有统计学意义。
结果1. 基本特征:患者临床基线特征见表 1。训练组中年龄、BMI、吸烟史、性别和临床分期在肺腺癌EGFR突变型和野生型之间差异有统计学意义(t=-2.10、2.36,χ2=35.66、29.03、17.84,P<0.05);测试组中BMI、吸烟史和性别在两者之间差异具有统计学意义(t=2.80,χ2=14.18、6.10,P<0.05);患者血清肿瘤标志物(CEA、CYFRA21-1、NSE)和病灶位置在训练组和测试组差异均无统计学意义(P>0.05)。logistic回归分析表明,年龄、BMI、吸烟史和性别是肺腺癌EGFR基因突变状态的相关预测因素(P<0.05)。
|
|
表 1 患者临床基线特征 Table 1 Clinical baseline characteristics of patients |
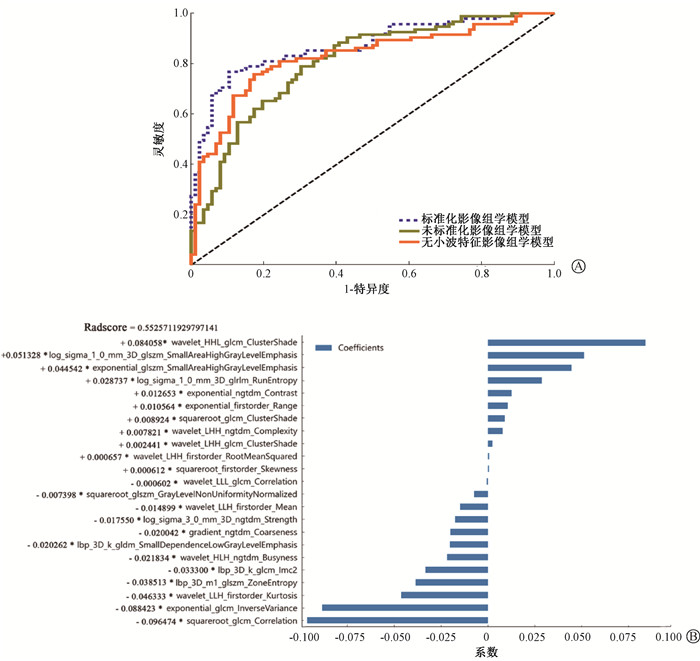
2. 影像组学特征筛选:分别从标准化及未标准化增强CT图像中提取出1 835个影像组学特征,经过LASSO算法和10倍交叉验证,最终筛选出23个影像组学特征用于构建影像组学模型,其中包括8个小波特征。通过Delong检验,标准化影像组学模型的预测性能好于未标准化的影像组学模型[AUC:0.863(95%CI:0.810~0.916) vs. 0.805(95%CI:0.739~0.860),t=2.19,P=0.028]及不含小波特征的标准化影像组学模型[AUC:0.863(95%CI:0.810~0.916) vs. 0.820(95%CI:0.757~0.873),t=2.23,P=0.026](图 1A)。根据筛选的标准化影像组学特征的权重系数计算影像组学评分(Rad-score)(图 1B),其在训练组(野生型 vs. 突变型:0.33±0.22 vs. 0.70± 0.24)和测试组(野生型 vs. 突变型:0.37±0.22 vs. 0.67±0.21)中差异均有统计学意义(t=10.96、6.00,P<0.05)。
|
图 1 影像组学特征筛选 A.3种影像组学模型ROC曲线;B.标准化影像组学特征权重系数分布图 Figure 1 Radiomics features screening A. ROC curves of three radiomics models; B. Distribution of selected standardized radiomics features weight coefficients |
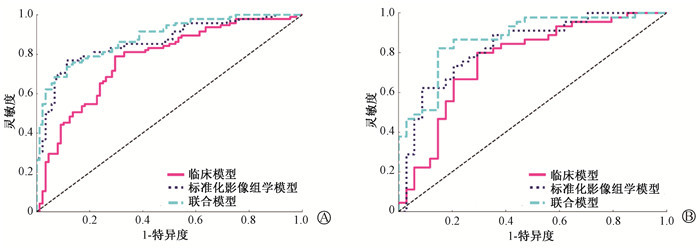
3. 模型建立:采用LR机器学习方法建立临床模型、标准化影像组学模型和联合模型。临床模型由BMI、吸烟史、性别和年龄4个特征建立,联合模型由4个临床特征和23个标准化影像组学特征建立,并绘制3种模型的ROC曲线。在训练组中,临床模型、标准化影像组学模型及联合模型的AUC分别为0.774(95%CI:0.706~0.842)、0.863(95%CI:0.810~0.916)和0.885(95%CI:0.839~0.932),测试组中,3种模型的AUC分别为0.763(95%CI:0.651~0.875)、0.829(95%CI:0.735~0.923)和0.873(95%CI:0.794~0.951)(图 3)。3种模型在训练组及测试组中的准确度、灵敏度、特异度、PPV和NPV见表 2。
|
图 3 训练组(A)和测试组(B)中3种预测模型的ROC曲线 Figure 3 ROC curves of three prediction models in the training(A) and testing(B) groups |
|
|
表 2 不同模型对肺腺癌患者EGFR基因突变状态的预测性能 Table 2 Prediction performance of different models for EGFR gene mutation status in lung adenocarcinoma patients |
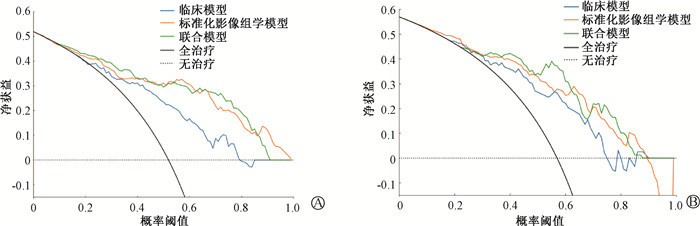
4. 模型预测效能评估和列线图绘制:采用Delong检验对3种预测模型进行比较见表 2,联合模型、标准化影像组学模型对比临床模型的AUC在训练组(0.885、0.863 vs. 0.774,t=3.57、2.17,P<0.05)和测试组(0.873、0.829 vs. 0.763,t=2.19、2.02,P<0.05)差异均具有统计学意义;联合模型与标准化影像组学模型的AUC在训练组(0.885 vs. 0.863)和测试组(0.873 vs. 0.829)差异无统计学意义(P>0.05)。决策曲线(DCA)表明,联合模型及影像组学模型在训练组和测试组中相比于临床模型能获得更高的临床净增益(图 4)。
|
图 4 训练组(A)和测试组(B)中3种模型的DCA曲线 Figure 4 DCA curves of the three models in the training(A) and testing(B) group |
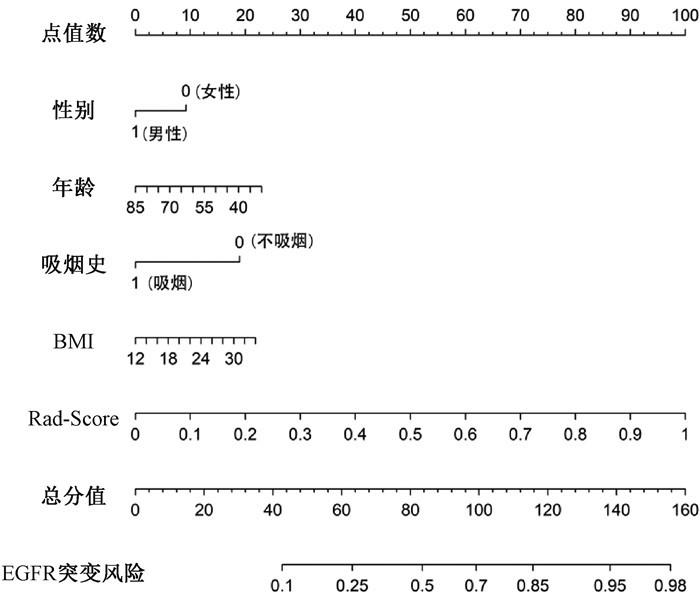
预测肺腺癌EGFR基因突变的影像组学列线图由年龄、BMI、吸烟史和性别4个临床因素及Rad-score共同建立(图 5),该列线图实现了肺腺癌EGFR基因突变的个体化精准预测和可视化效果,通过每一个风险因素对应的点值数来计算出总分值,总分值越高,EGFR突变的可能性就越高。
|
图 5 肺腺癌EGFR突变的列线图 Figure 5 Nomogram of EGFR mutations in lung adenocarcinoma |
讨论
随着靶向治疗的发展,非小细胞肺癌的治疗模式从最初的以铂类为基础的传统化疗转向个体化的分子靶向治疗。研究表明,亚裔人群肺腺癌患者更容易发生EGFR突变[10, 20-21],而肺鳞癌的EGFR突变率低,仅占3.3%~4.6%,且即便是存在EGFR突变,TKI的疗效也远不及肺腺癌[16]。因此本研究只针对肺腺癌EGFR突变状态进行预测。
以往研究表明,女性、未吸烟患者更易发生EGFR突变[22-24],本研究纳入的6个临床特征中,性别、吸烟状况、年龄及BMI在EGFR突变型和野生型之间存在显著差异,并用于构建临床模型,训练组的AUC和准确率分别为0.774 (95%CI:0.706~0.842)和74.9%,测试组的AUC和准确率分别为0.763(95%CI:0.651~0.875)和75.9%,这与以往临床模型预测肺癌患者EGFR基因突变状态的结果相似(AUC:0.610~0.754)[10, 17, 25-26]。
影像组学可从医学图像中挖掘大量肉眼无法识别的多维度数据,通过定量分析探索肿瘤的异质性及基因信息[7, 9]。目前,影像数据采集主要来自CT、磁共振(MRI)、正电子发射断层显像/X射线计算机体层成像仪(PET/CT)等设备,不同的采集设备及参数对图像质量、数据分析均有影响。为提高影像组学特征的鲁棒性,保证模型的可重复性,本研究在图像分割之前对分辨率和对比度进行归一化预处理,并与未标准化的影像组学模型进行对比,结果显示标准化的影像组学模型预测EGFR突变状态的效能好于未标准化的影像组学模型。通过三维小波变换分解原始图像,重新计算纹理特征和一阶特征,描述肿瘤病灶内不同频段的特点[27]。本研究共提取728个小波特征,筛选出8个小波特征,含小波的影像组学模型预测EGFR突变状态的效能好于无小波特征的影像组学模型,特征数量的增加提供了更多有价值的信息,提高了模型的性能。
目前已有研究探讨基于CT影像组学特征与肺癌患者EGFR表达之间的相关性,但多数研究是基于平扫CT进行分析。李淑华等[17]提取183例肺腺癌患者胸部平扫CT影像组学特征并建立影像组学模型,其AUC在训练组及验证组中分别为0.797、0.783。张国晋等[10]对286例肺腺癌患者影像组学模型进行分析,AUC在训练组和验证组中分别为0.762、0.779。本研究基于标准化增强CT影像组学特征建立影像组学模型,AUC在训练组和验证组中分别为0.863、0.829,高于既往基于平扫CT建立的影像组学模型。其可能原因:一是平扫主要反映肿瘤内组织成分及细胞密度不同的非均质性以及坏死等系列继发密度上的改变,而增强CT扫描则能反映肿瘤血供上的非均质性特点;二是增强图像可以更准确地描绘病变边缘,减少对影像组学特征的影响[14, 19]。本研究中,GLSZM及GLCM是权重系数较高的特征,反映了一定程度上肿瘤内部纹理的变化,可能与肿瘤内部的异质性相关。
据文献报道,影像组学模型联合临床模型构建的综合模型的预测性能高于单一模型[5, 17]。本研究中联合模型的预测性能明显高于单一临床模型,但联合模型的预测性能与影像组学模型差异无统计学意义(训练组AUC:0.885 vs. 0.863、测试组AUC:0.873 vs. 0.829,P>0.05),可能原因是基于影像组学建立的模型预测EGFR突变的权重高于临床模型,另一方面也表明影像组学特征相比于临床特征更能反映肿瘤内部异质性。本研究利用Rad-score和临床特征(年龄、性别、吸烟史及BMI)绘制了列线图,实现模型的可视化[28]。DCA表明联合模型和影像组学模型在预测EGFR突变状态方面比临床模型可增加更多的净效益。
本研究仍存在一定的局限性。首先,本研究是回顾性的单中心研究,样本量较小,且只进行了内部交叉验证,缺乏外部验证,这可能会影响预测模型的真实诊断性能。其次,本研究是手动逐层勾画ROI,费时费力,半自动或自动绘制ROI的技术有待进一步发展。另外,本研究仅对EGFR突变状态进行了分析,没有进一步分析其突变亚型。综上所述,基于临床危险因素和影像组学特征建立的联合模型及列线图,可作为预测EGFR基因突变状态的一种安全、无创、有效、经济的临床工具,帮助临床医生优化相关治疗策略。
利益冲突 无
作者贡献声明 王寻负责实验设计、研究实施及论文撰写;葛双、马俊丽协助实验设计和论文修改;郗会珍、马俊、刘亚茹收集患者影像及临床资料,负责数据处理和统计分析;叶书成指导文章修改
| [1] |
Jia B, Zheng Q, Wang J, et al. A nomogram model to predict death rate among non-small cell lung cancer (NSCLC) patients with surgery in surveillance, epidemiology, and end results (SEER) database[J]. BMC Cancer, 2020, 20(1): 666. DOI:10.1186/s12885-020-07147-y |
| [2] |
Duma N, Santana-Davila R, Molina JR. Non-small cell lung cancer: epidemiology, screening, diagnosis, and treatment[J]. Mayo Clin Proc, 2019, 94(8): 1623-1640. DOI:10.1016/j.mayocp.2019.01.013 |
| [3] |
Siegel RL, Miller KD, Fuchs HE, et al. Cancer statistics, 2022[J]. CA Cancer J Clin, 2022, 72(1): 7-33. DOI:10.3322/caac.21708 |
| [4] |
Molina JR, Yang P, Cassivi SD, et al. Non-small cell lung cancer: epidemiology, risk factors, treatment, and survivorship[J]. Mayo Clin Proc, 2008, 83(5): 584-594. DOI:10.4065/83.5.584 |
| [5] |
Dang Y, Wang R, Qian K, et al. Clinical and radiological predictors of epidermal growth factor receptor mutation in nonsmall cell lung cancer[J]. J Appl Clin Med Phys, 2021, 22(1): 271-280. DOI:10.1002/acm2.13107 |
| [6] |
Nair J, Saeed UA, McDougall CC, et al. Radiogenomic models using machine learning techniques to predict EGFR mutations in non-small cell lung cancer[J]. Can Assoc Radiol J, 2021, 72(1): 109-119. DOI:10.1177/0846537119899526 |
| [7] |
Dong Y, Jiang Z, Li C, et al. Development and validation of novel radiomics-based nomograms for the prediction of EGFR mutations and Ki-67 proliferation index in non-small cell lung cancer[J]. Quant Imaging Med Surg, 2022, 12(5): 2658-2671. DOI:10.21037/qims-21-980 |
| [8] |
Ramalingam SS, Vansteenkiste J, Planchard D, et al. Overall survival with osimertinib in untreated, EGFR-mutated advanced NSCLC[J]. N Engl J Med, 2020, 382(1): 41-50. DOI:10.1056/NEJMoa1913662 |
| [9] |
胡江, 贺睿敏, 程品晶, 等. 治疗前CT影像组学结合机器学习预测非小细胞肺癌患者EGFR突变亚型[J]. 中华放射医学与防护杂志, 2023, 43(5): 386-392. Hu J, He RM, Cheng PJ, et al. Prediction of EGFR mutant subtypes in patients with non-small cell lung cancer by pre-treatment CT radiomics and machine learning[J]. Chin J Radiol Med Prot, 2023, 43(5): 386-392. DOI:10.3760/cma.j.cn112271-20221223-00495 |
| [10] |
张国晋, 孔维芳, 尚兰, 等. 基于术前CT影像组学特征预测肺腺癌患者EGFR突变状态的研究[J]. 临床放射学杂志, 2023, 42(5): 760-764. Zhang GJ, Kong WF, Shang L, et al. Study on predicting EGFR mutations status in patients with lung adenocarcinoma based on preoperative CT radiomics features[J]. J Clin Radiol, 2023, 42(5): 760-764. |
| [11] |
Lambin P, Leijenaar R, Deist TM, et al. Radiomics: the bridge between medical imaging and personalized medicine[J]. Nat Rev Clin Oncol, 2017, 14(12): 749-762. DOI:10.1038/nrclinonc.2017.141 |
| [12] |
Thawani R, McLane M, Beig N, et al. Radiomics and radiogenomics in lung cancer: a review for the clinician[J]. Lung Cancer, 2018, 115: 34-41. DOI:10.1016/j.lungcan.2017.10.015 |
| [13] |
Rossi G, Barabino E, Fedeli A, et al. Radiomic detection of egfr mutations in NSCLC[J]. Cancer Res, 2021, 81(3): 724-731. DOI:10.1158/0008-5472.CAN-20-0999 |
| [14] |
Wu S, Shen G, Mao J, et al. CT radiomics in predicting EGFR mutation in non-small cell lung cancer: a single institutional study[J]. Front Oncol, 2020, 10: 542957. DOI:10.3389/fonc.2020.542957 |
| [15] |
Friedlaender A, Banna G, Malapelle U, et al. Next generation sequencing and genetic alterations in squamous cell lung carcinoma: where are we today?[J]. Front Oncol, 2019, 9: 166. DOI:10.3389/fonc.2019.00166 |
| [16] |
周海英, 韩进伟, 吕铮, 等. 1例晚期肺鳞癌患者EGFR突变病例报道并文献复习[J]. 临床与病理杂志, 2019, 39(11): 2627-2630. Zhou HY, Han JW, Lv Z, et al. EGFR mutations in patients with advanced lung squamous cell carcinoma: A case report and literature review[J]. Int J Pathol Clin Med, 2019, 39(11): 2627-2630. DOI:10.3978/j.issn.2095-6959.2019.11.047 |
| [17] |
李淑华, 杨昭, 王小雷, 等. 基于CT影像组学列线图预测肺腺癌EGFR突变的研究[J]. 临床放射学杂志, 2022, 41(9): 1676-1682. Li SH, Yang Z, Wang XL, et al. CT-based radiomics nomogram to predict EGFR mutation in lung adenocarcinoma[J]. J Clin Radiol, 2022, 41(9): 1676-1682. DOI:10.13437/j.cnki.jcr.2022.09.037 |
| [18] |
Yang X, Liu M, Ren Y, et al. Using contrast-enhanced CT and non-contrast-enhanced CT to predict EGFR mutation status in NSCLC patients - a radiomics nomogram analysis[J]. Eur Radiol, 2022, 32(4): 2693-2703. DOI:10.1007/s00330-021-08366-y |
| [19] |
Hong D, Xu K, Zhang L, et al. Radiomics signature as a predictive factor for EGFR mutations in advanced lung adenocarcinoma[J]. Front Oncol, 2020, 10: 28. DOI:10.3389/fonc.2020.00028 |
| [20] |
Zhang G, Zhang J, Cao Y, et al. Nomogram based on preoperative CT imaging predicts the EGFR mutation status in lung adenocarcinoma[J]. Transl Oncol, 2021, 14(1): 100954. DOI:10.1016/j.tranon.2020.100954 |
| [21] |
Sacco JJ, Al-Akhrass H, Wilson CM. Challenges and strategies in precision medicine for non-small-cell lung cancer[J]. Curr Pharm Des, 2016, 22(28): 4374-4385. DOI:10.2174/1381612822666160603014932 |
| [22] |
朱光胜, 李永文, 施睿峰, 等. 基于TCGA数据库的EGFR突变型与野生型肺腺癌患者免疫微环境的差异性分析[J]. 中国肺癌杂志, 2021, 24(4): 236-244. Zhu GS, Li YW, Shi RF, et al. Immune microenvironment comparation study between EGFR mutant and EGFR wild type lung adenocarcinoma patients based on TCGA database[J]. Chin J Lung Cancer, 2021, 24(4): 236-244. DOI:10.3779/j.issn.1009-3419.2021.102.15 |
| [23] |
Tsai MJ, Hung JY, Lee MH, et al. Better progression-free survival in elderly patients with stage iv lung adenocarcinoma harboring uncommon epidermal growth factor receptor mutations treated with the first-line tyrosine kinase inhibitors[J]. Cancers (Basel), 2018, 10(11): 434. DOI:10.3390/cancers10110434 |
| [24] |
Wang C, Ma J, Shao J, et al. Predicting EGFR and PD-L1 status in NSCLC patients using multitask ai system based on CT images[J]. Front Immunol, 2022, 13: 813072. DOI:10.3389/fimmu.2022.813072 |
| [25] |
Wang S, Shi J, Ye Z, et al. Predicting EGFR mutation status in lung adenocarcinoma on computed tomography image using deep learning[J]. Eur Respir J, 2019, 53(3): 1800986. DOI:10.1183/13993003.00986-2018 |
| [26] |
段亚楠, 阮君, 刘思耘, 等. CT影像组学对非小细胞肺癌ALK和EGFR突变的预测[J]. 临床放射学杂志, 2023, 42(7): 1117-1122. Duan YN, Ruan J, Liu SY, et al. Prediction of ALK and EGFR mutations in non-small cell lung cancer by CT radiomics[J]. J Clin Radiol, 2023, 42(7): 1117-1122. DOI:10.13437/j.cnki.jcr.2023.07.024 |
| [27] |
杨春生. 基于CT影像组学特征预测晚期肺腺癌EGFR突变状态及TKIs治疗敏感性的研究[D]. 济南: 山东大学, 2022. DOI: 10.27272/d.cnki.gshdu.2022.006385. Yang CS. Application of CT radiomics features to predict the EGFR mutation status and therapeutic sensitivity to TKIs of advanced lung adenocarcinoma[D]. Jinan: Shandong University, 2022. DOI: 10.27272/d.cnki.gshdu.2022.006385. |
| [28] |
Balachandran VP, Gonen M, Smith JJ, et al. Nomograms in oncology: more than meets the eye[J]. Lancet Oncol, 2015, 16(4): e173-e180. DOI:10.1016/S1470-2045(14)71116-7 |
 2024, Vol. 44
2024, Vol. 44


