2. 浙江大学信息与电子工程学院, 杭州 310058
2. College of Information Science & Electronic Engineering, Zhejiang University, Hangzhou 310058, China
鼻咽癌(NPC) 是东亚和东南亚最常见的癌症之一[1],而放射治疗是首选治疗手段之一。成功的鼻咽癌放射治疗依赖于精准的靶区勾画。手动靶区勾画耗费医生大量时间精力,并且观察者间差异也是影响勾画一致性的重要因素[2-3]。近年来,随着深度学习的不断发展,研究者们借助深度学习模型实现了肿瘤靶区的自动精准勾画,在减轻医生负担的同时提高了勾画的一致性[4-5]。
鼻咽癌肿瘤靶区(GTV)主要由鼻咽部肿瘤原发灶(GTVnx)和转移的区域淋巴结(GTVnd)组成。仅通过平扫CT往往难以精准确定鼻咽癌靶区,临床实践中医生往往会对比平扫期(CT)、增强期(CTC)、延迟期(CTD)扫描,综合判断以完成鼻咽癌肿瘤靶区的勾画。先前的许多研究使用单期增强CT来自动勾画鼻咽癌靶区,尽管造影剂强化后的CT成像更有利于区分一些器官和组织,但勾画准确性仍有待提高,尤其是对于GTVnd这样形状多变、边界与正常组织非常接近的靶区[6]。而当注射造影剂后,不同组织器官呈现出不同的强化模式,而仅使用单期扫描进行自动勾画忽略了造影剂强化过程中时间维度的对比信息。
本研究构建了一个基于3D U-Net结合三个期相CT图像的分割模型,用于鼻咽癌肿瘤靶区(GTVnx和GTVnd)的自动勾画,探索三期相CT对于鼻咽癌靶区自动勾画效果的影响。此外,本研究还探索了利用期相微调预训练模型,仅使用平扫期图像对GTVnx和GTVnd进行自动勾画时的影响。
资料与方法1. 临床资料:本研究获得了浙江大学医学院附属第一医院伦理委员会的批准(2023研第0123-快),回顾性地选择了2019年1月至2022年6月期间在浙江大学医学院附属第一医院治疗的215名鼻咽癌患者资料。采用随机数字表法,分成训练集172例和测试集43例,其中,男性133例,女性82例,中位年龄58岁,靶区范围根据不同分期包括颅底至锁骨上区。局部晚期癌症患者采用诱导化疗,然后同步放化疗,其余患者接受放疗或同步放化疗方案,处方剂量为7 000 cGy/32~33次。
2. 数据采集:在CT模拟定位中,患者使用头颈肩热塑性面罩仰卧位固定在治疗底板上(Orfit AIO Solution3.0,Wijnegem,比利时),在西门子大孔径CT(Somatom Definition AS 64, Forchheim, 德国)上进行对比增强CT扫描,扫描参数为120 kV,1.5 mm层厚采集并重建,对比剂注射方案为2 ml/kg,流率2~3 ml/s。扫描范围包括头顶至气管隆突水平。首先完成平扫,在造影剂注入后约18~20 s开始进行增强扫描,60 s后进行延迟扫描,得到扫描范围一致的三期图像。鼻咽癌靶区勾画需要参考核磁共振(MR)图像以提高软组织分辨率,将MR图像在医科达TPS(Monaco 6.00,斯德哥尔摩,瑞典)融合到平扫图像中,对于有些MR体位不佳患者采用MIM(MIM 6.8.2,克利夫兰,美国)进行弹性融合。所得图像由两位经验丰富的放疗科主治医师依据国家癌症中心(NCC)发布的鼻咽癌靶区勾画指南[7]在平扫CT图像上手动勾画,所有结果由1位10年以上经验的主任医师进行最终确认,保证数据标签的一致性。
3. 数据预处理
(1) 重采样:将DICOM文件都转换为NIFTI格式,并统一方向为右前脚(RAI)方向,将CTC和CTD按照CT的扫描原点进行重采样,使得同一病例的三期扫描具有同样数目且内容对应的切片。
(2) 裁剪:为了减小训练和测试的计算负担,并使得模型关注于人体相关的有效部分,需要对三期CT扫描进行体积裁剪。本研究使用大津法(OTSU)对CT进行二值化[8],获得人体部分(排除空气、固定支架和扫描床)。随后将三期扫描图像裁剪到能够刚好包含人体部分的最小长方体。
(3) 归一化:临床上,医生通常使用350 HU的窗宽和40 HU的窗位来勾画头颈部靶区,本研究使用相同的归一条件:强度在[-310, 390] 之外的体素被设置为-310和390,随后将[-310, 390]范围的体素强度缩放到[0, 1]。针对性的归一化给予鼻咽癌靶区相关的体素更大的表示范围,使模型能够更好地关注靶区相关的特征。归一化步骤如式1所示:
| $ X_{\text {norm }}= \begin{cases}1 & X>390 \\ \frac{X-X_{\min }}{X_{\max }-X_{\min }} & -310 \leqslant X \leqslant 390 \\ 0 & X \leqslant-310\end{cases} $ | (1) |
4. 网络结构:如图 1所示,本研究使用由5层3D编码器-解码器组成的3D Unet。在每个编码器中使用了两组由3 × 3 × 3卷积核的3D卷积、批归一化和ReLU激活层组成的特征提取组合,随后使用2 × 2 × 2的3D最大池化层在xyz 3个维度进行2倍下采样,以扩大后续编码器的感受野。3D编码器5的结构与其他的编码器有所不同,使用了Dropout随机失活层来抑制模型的过拟合现象。4个解码器模块的结构相同,模块首先使用2 × 2 × 2卷积核的3D转置卷积对下层编码器输入的特征进行上采样,与跳跃连接输入的特征图层叠后再使用“3D卷积+批归一化+ReLU激活层“提取特征,完成自动勾画并输出结果。
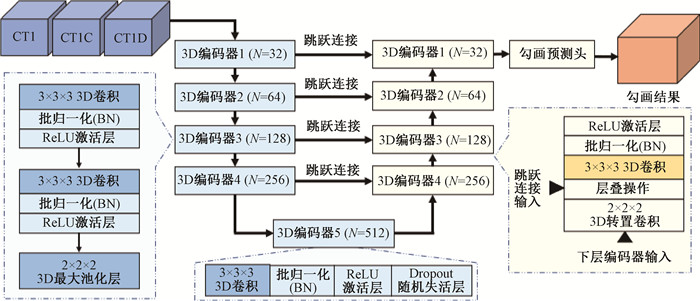
|
图 1 自动分割模型工作流概览 Figure 1 Overview of workflow for an automatic segmentation model |
5. 训练目标:本研究中,在训练网络勾画GTVnx时所使用的损失函数(loss function)为交叉熵和Dice Loss之和(公式2)。在训练网络勾画GTVnd时,由于转移淋巴结靶区位置分布多样,在CT成像中的边界不清晰,神经网络容易将正常的组织误判为靶区。因此,本研究修改了Dice Loss,在损失函数中增加了网络将正常组织误判为靶区情况的惩罚权重,从而抑制上述情况的发生(公式3)。
| $ L_{\mathrm{GTVnx}}=L_{\text {dice }}+L_{\mathrm{CE}} $ | (2) |
| $ L_{\text {GTVnd }}=L_{\text {modified }_{\text {dice }}}+L_{\mathrm{CE}} $ | (3) |
式中,Ldice、Lmodified_dice、LCE分别如下式所示:
| $ L_{\text {dice }}=-\frac{2 \sum _{i=1}^N y_i \hat{y}_i}{\sum _{i=1}^N y_i+\sum _{i=1}^N \hat{y}_i} $ | (4) |
| $ \begin{aligned} & L_{\text {modified }_{\text {dice }}}= \\ & -\frac{2 \sum _{i=1}^N y_i \hat{y}_i}{\sum _{i=1}^N y_i+\sum _{i=1}^N \hat{y}_i+\sum _{i=1}^N \hat{y}_i\left(1-y_i\right)} \\ & \end{aligned} $ | (5) |
| $ L_{\mathrm{CE}}=-\frac{1}{N} \sum\limits_{n=1}^N \hat{y}_i \log y_i $ | (6) |
式中,yi和
6. 期相微调模型:在期相微调实验中设置了3个实验分组,对比了直接在平扫期上训练模型与经过增强期/延迟期训练后在平扫期上进行期相微调后模型的勾画效果。首先输入经过预处理的训练集病例的增强期或延迟期图像,batch size设置为4,切分32 × 64 × 128图像块作为3D U-Net的输入进行训练,损失函数设置和上文一致。模型训练过程中使用动量(momentum)为0.9的随机梯度下降(SGD)作为优化器,初始学习率设置为0.01,训练100轮次(epoch)后观察到训练集loss不再下降后,即停止训练。随后将训练集病例的平扫期图像输入模型进行期相微调训练,设置初始学习率为0.01,由于模型已经学习了增强期/延迟期CT上的靶区特征,微调50轮次(epoch)后观察到训练集loss不再下降即停止训练,并使用测试集的平扫期数据对模型进行测试。期相微调实验的训练和测试流程如下图 2所示。
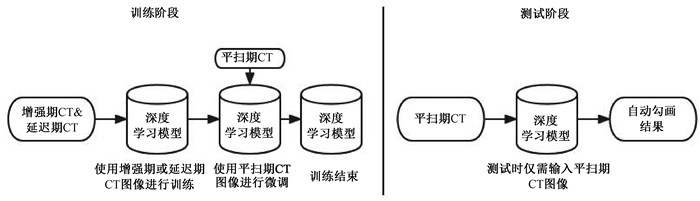
|
图 2 期相微调实验的训练及测试流程 Figure 2 Training and testing process of a phase fine-tuning CTC experiment |
7. 评估指标:本研究使用的数据集包含215例鼻咽癌病例,被随机分为训练集和测试集两组:其中训练集包含172例患者,用于训练网络学习靶区特征;测试集包含43例患者,用于评估模型勾画效果。由于显卡显存的限制,训练时随机裁剪CT图像中的32 × 64 × 128图像块作为3D U-Net的输入,网络输入的batch size为4。模型训练过程中使用动量(momentum)为0.9的随机梯度下降(SGD)作为优化器,初始学习率设置为0.01。所有实验分组均在训练100轮次(epoch)后进行测试,测试时使用滑动窗口法将CT图像块(patch)输入模型,窗口之间的重叠比例为0.5,并对各个patch进行高斯融合得到完整尺寸的勾画结果。将模型勾画结果与专家勾画对比,得到各个评价指标。为了探究三期CT扫描对于勾画鼻咽癌靶区的提升,本研究设置了4个实验分组,对比仅使用平扫期A1组(CT)、仅使用增强期A2组(CTC)、仅使用延迟期A3组(CTD)和同时使用三期扫描A4组(All)条件下的自动勾画效果,并在GTVnd和GTVnx两个勾画对象上评估了方法的有效性。
为了定量评估模型的勾画准确性,采用了Dice相似系数(Dice similarity coefficient,DSC)和95%豪斯多夫距离(95%Hausdorff distance, HD95),作为评价指标以评估模型的靶区勾画结果(GTVnx和GTVnd)与专家勾画之间的差异[9]。
8. 统计学处理:读取模型对于各个测试集病例的勾画结果,对比这些病例的专家勾画轮廓分别计算DSC和HD95指标。本研究使用统计软件SPSS 21.0对实验结果进行分析,采用配对t检验对比使用三期CT扫描和仅使用各个单期扫描的结果差异。P < 0.05为差异有统计学意义。
结果1. 深度学习模型勾画性能评估:表 2为仅使用CT、仅使用CTC、仅使用CTD和同时使用3种期相的情况下,3D U-Net对于GTVnd和GTVnx在测试集病例的勾画结果,表明使用三期相CT(A4)进行GTVnd靶区自动勾画相比于仅使用单期相CT(A1、A2、A3)获得更好的勾画效果(DSC:0.67 vs. 0.61、0.64、0.64,t=7.48、3.27、4.84,P < 0.01;HD95: 36.45 mm vs. 79.23、59.55、65.17 mm,t=5.24、2.99、3.89,P < 0.01),差异有统计学意义。使用三期相CT(A4)对于GTVnx的自动勾画效果相比于仅使用单期相(A1、A2、A3)无明显提升(DSC: 0.73 vs. 0.74、0.74、0.74;HD95: 14.17 mm vs. 8.06、8.11、8.10 mm),差异无统计学意义(P>0.05)。结果提示,对于GTVnd使用三期图像可以提升自动勾画准确性,见图 3中的A所示,而GTVnx则没有明显受益。
|
|
表 2 使用不同期相勾画GTVnd和GTVnx的结果 Table 2 Automatic segmentation results of GTVnd and GTVnx using different phases of CT |
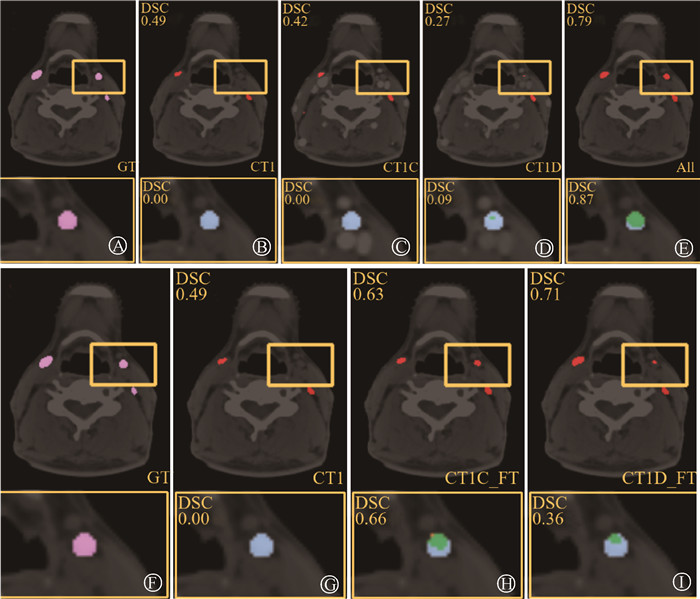
|
注:绿色表示正确勾画,橙色表示模型将正常组织误判为靶区,蓝色表示模型将靶区误判为正常组织。图中上下标注的两个DSC分别表示当前扫描层和局部放大图中模型自动勾画的DSC指标;图A~E为GTVnd的自动勾画结果;图F~I为期相微调GTVnd的自动勾画结果 图 3 使用两种训练模型进行GTVnd自动勾画的结果 A.专家标注(GT); B. 仅使用CT(CT1); C. 仅使用CTC(CT1C); D. 仅使用CTD(CT1D); E. 同时使用三期相CT图像(All);F. 专家标注(GT); G. 仅使用平扫期训练(CT); H. 增强期训练后在平扫期上进行期相微调(CTC_FT); I. 延迟期训练后在平扫期上进行期相微调(CTD_FT) Figure 3 Automatic segmentation results of GTVnd using two training models A. Expert annotation (GT); B. Only CT (CT1); C. Only CTC (CT1C); D. Only CTD (CT1D); E. Triphasic CT; F. Expert annotation (GT); G. Only CT; H. Phase fine-tuning based on CT after CTC; I. Phase fine-tuning based on CT after CTD |
2. 期相微调性能评估:表 2中GTVnd(期相微调)中对比了直接使用CT进行训练,以及模型在其他期相上训练后再在CT上进行微调的情况下的不同勾画性能。由于在前一个实验中使用CT、CTC和CTD对GTVnx的自动勾画效果差异无统计学意义(P>0.05)。因此,本研究仅对于GTVnd进行了期相微调影响的研究。相对于直接在CT(DSC: 0.61±0.13,HD95: 79.23±57.33)上进行训练,预先CTC和CTD上训练后再进行微调的模型取得了更高的DSC(CTC: 0.63±0.13; CTD: 0.63±0.12)和HD95(CTC: 58.11±50.32; CTD: 50.31±46.17),并且结果在DSC和HD95上差异均有统计学意义(t=4.10、3.03、2.75、3.10,P < 0.01)。结果表明,先学习CTC或者CTD的纹理特征,然后在平扫上训练,得到的模型后期只使用平扫图像,自动勾画GTVnd效果也会有提升,见图 3。
讨论鼻咽癌放射治疗的成功取决于GTV的精准勾画,手动勾画耗时较长且耗费医生大量精力。随着深度学习的不断发展,研究者们借助深度学习模型实现了肿瘤靶区的自动精准勾画,在减轻医生负担、提高工作效率的同时,提高了勾画的一致性。
Men等[10]将卷积神经网络(CNN)首次应用于鼻咽癌放疗靶区的自动勾画,提出了由编码器和解码器组成的深度反卷积神经网络(DDNN),并基于2D的增强CT图像输入,在GTVnd的勾画任务上得到了DSC为0.623的结果。随着深度学习的发展,U-Net及其变体在医学图像分割领域被提出并在大量器官分割及病变分割研究中取得优异表现[11-14]。相比于之前一些工作使用的2D U-Net模型及其变形[15],本研究采用的3D U-Net模型使用3D图像块作为输入,能够更好地捕捉相邻扫描切片之间靶区的空间联系。因此,GTVnd(DSC=0.66)也得到了更好的结果[16-17]。此外,也有一些研究使用其他模态图像,如Ma等[18]和Liao等[19]的研究由于MRI对于软组织的分辨率更高,提高了鼻咽癌靶区勾画的准确性,展现了更好的DSC。Li等[20]使用CNN在MRI图像上完成了对GTVnx的自动勾画,并且达到了0.79的DSC。然而,因MR图像的采集时间及体位和真正用于放疗的定位CT存在差异,且目前放射治疗中的剂量计算仍然依赖于定位CT图像,需要将其他模态图像与定位CT图像进行融合配准,这将不可避免地带来误差,直接影响勾画精度。因此,本研究希望寻求一种直接采用多期相定位CT图像的方法来实现鼻咽癌GVT的自动勾画。相比于仅使用单个期相或MR图像,3个期相的CT扫描图像能够更好地表现各个器官组织的不同强化的时间维度特征,更有利于对深度学习模型进行区分。
以往的鼻咽癌GTV仅仅通过人工参考平扫CT图像难以精准勾画,特别是对于GTVnd,往往和血管难以区分,多期的CT增强图像能通过造影剂的不同程度强化的动态变化来对其进行区分。本研究希望通过引入深度学习强大的分析和推理能力,通过分析多期相CT图像中的造影剂带来的时间维度的信息,识别多期相间的差异特征来提升自动勾画的能力,相应的研究结果也证明了方法的可行性。本研究采集了病例平扫期、增强期、延迟期3个期相的CT扫描,分别对应强化前、强化中和强化后3个时间点,使用3D U-Net模型,对比了仅使用平扫期、增强期、延迟期和同时使用3个CT扫描期相进行自动勾画的效果。GTVnd勾画任务的实验结果表明,相比于仅使用CT,造影剂的强化后的CTC和CTD有助于模型更好地勾画GTVnd靶区,即同时使用三期相CT图像作为输入时,模型能够捕捉GTVnd和邻近组织器官在时间维度上不同的强化过程特征,因而呈现出最佳的勾画结果。在GTVnx勾画任务中,仅使用CT、CTC和CTD进行单期相勾画的结果相近,而使用3个期相进行GTVnx勾画的结果与仅使用单期相的结果相近,差异无统计学意义。上述现象可能是由于造影剂对于GTVnx邻近组织的强化作用不明显造成的。
基于上述实验结果,使用3个期相能够有效提高模型对于GTVnd的勾画精度,而一些病例可能存在造影剂过敏等不适用于增强扫描的情况,因此本研究还尝试了提高模型在仅使用平扫期进行GTVnd靶区自动勾画的实验。实验结果表明,模型使用训练集的CTC或CTD期相进行训练,随后在训练集CT期相上进行微调的效果相比直接在平扫CT上进行训练有明显提高,且DSC和HD95的精度提升有统计学意义。
在期相微调方法中,模型首先学习增强后GTVnd靶区的特征,随后在CT上根据平扫期靶区的特征继续学习以进行参数微调,这一过程中模型隐式地学习了GTVnd靶区和周围组织的造影剂强化后的差异特征。经过期相微调方法训练后的模型,在仅使用平扫期进行测试时,其在测试集病例上相比于使用平扫CT直接训练的模型依然具有更高的自动勾画精度,这证明了期相微调方法提高了模型的泛化能力。
在深度学习中,数据集的大小和质量起到至关重要的作用。本研究在具有215例病例的鼻咽癌数据集上取得了不错的效果,在数据集大小进一步扩充的情况下,深度学习模型对于鼻咽癌肿瘤靶区(GTVnx和GTVnd)的自动勾画仍有进一步提高的空间。本研究为单一中心研究,图像及勾画一致性较好,并且由于数据集的数量有限,为了有更多的数据用于训练,本文没有划分验证集,而是在模型训练时对训练集使用“早停”策略,在100轮次(epoch)后观察到训练集损失函数不再下降,即停止模型训练,随后进行模型测试,从而保证测试集的独立性。未来将在此基础上开展多中心的研究,在扩充数据集病例数量的同时增加验证集的划分。后期计划与其他研究机构合作,使用来自不同CT扫描设备、不同医疗组的鼻咽癌病例CT扫描及勾画结果进行训练,进一步验证和优化利用三期相CT扫描进行鼻咽癌肿瘤靶区实现自动勾画的泛化性和鲁棒性。鼻咽癌的GTV自动勾画是业界的一个难题,在单一模态图像中很难得到理想的DSC值,同类研究基本平均都在0.58~0.72之间[21]与本研究的0.73类似。本研究希望借助深度学习的方法依靠多期相增强定位CT图像,提升自动勾画精度,简化定位工作流程,为相关研究提供一个新的思路。
成功的鼻咽癌放射治疗依赖于精准的GTV勾画,手动勾画靶区耗时费力并且一致性不足,利用深度学习模型进行自动勾画能够大大提升医生的工作效率并提高诊疗一致性。基于深度学习模型,使用三期相CT扫描进行GTVnd靶区自动勾画相比于仅使用单期,在勾画准确性上具有显著性提升,并且相比于使用MRI进行自动勾画的方法,具有无需配准的优势。由于GTVnd位置分布的多样性及其与周围组织的相似性,仅凭借CT扫描信息仍然无法充分加以区分,未来将尝试融合患者病历文本、诊断时拍摄的MRI扫描等多来源、多模态信息,进一步提升GTVnd和GTVnx的勾画精度。
利益冲突 所有作者声明没有相关利益冲突
作者贡献声明 姚国荣负责论文撰写及修改;沈恺负责算法实现,协助撰写论文;赵峰、王思源负责数据准备;陆中杰负责数据核对;黄科杰负责算法的科学审核;严森祥负责整体方案设计与论文修改
| [1] |
Chen YP, Chan A, Le QT, et al. Nasopharyngeal carcinoma[J]. Lancet, 2019, 394(10192): 64-80. DOI:10.1016/S0140-6736(19)30956-0 |
| [2] |
Wang J, Chen Z, Yang C, et al. Evaluation exploration of Atlas-based and deep learning-based automatic contouring for nasopharyngeal carcinoma[J]. Front Oncol, 2022, 12: 833816. DOI:10.3389/fonc.2022.833816 |
| [3] |
Teguh DN, Levendag PC, Voet PW, et al. Clinical validation of atlas-based auto-segmentation of multiple target volumes and normal tissue (swallowing/mastication) structures in the head and neck[J]. Int J Radiat Oncol Biol Phys, 2011, 81(4): 950-957. DOI:10.1016/j.ijrobp.2010.07.009 |
| [4] |
Men K, Zhang T, Chen X, et al. Fully automatic and robust segmentation of the clinical target volume for radiotherapy of breast cancer using big data and deep learning[J]. Phys Med, 2018, 50: 13-19. DOI:10.1016/j.ejmp.2018.05.006 |
| [5] |
田素青, 许昕, 姜玉良, 等. 基于深度学习的多模态影像脑胶质母细胞瘤放疗靶区的自动勾画研究[J]. 中华放射医学与防护杂志, 2022, 42(9): 697-703. Tian SQ, Xu X, Jiang YL, et al. Application of deep learning-based multimodal imaging to the automatic segmentation of glioblastoma targets for radiotherapy[J]. Chin J Radiol Med Prot, 2022, 42(9): 697-703. DOI:10.3760/cma.j.cn112271-20220331-00133 |
| [6] |
Groendahl AR, Huynh BN, Tomic O, et al. Automatic gross tumor segmentation of canine head and neck cancer using deep learning and cross-species transfer learning[J]. Front Vet Sci, 2023, 10: 1143986. DOI:10.3389/fvets.2023.1143986 |
| [7] |
国家肿瘤质控中心, 国家癌症中心. 鼻咽癌靶区勾画和计划设计指南[J]. 中华放射肿瘤学杂志, 2022, 31(11): 971-991. National Cancer Center, National Cancer Quality Control Center. Guideline of target delineation and treatment planning for nasopharyngeal carcinoma[J]. Chin J Radiat Oncol, 2022, 31(11): 971-991. DOI:10.3760/cma.j.cn113030-20220110-00011 |
| [8] |
Malik YS, Tamoor M, Naseer A, et al. Applying an adaptive Otsu-based initialization algorithm to optimize active contour models for skin lesion segmentation[J]. J Xray Sci Technol, 2022, 30(6): 1169-1184. DOI:10.3233/XST-221245 |
| [9] |
Liu X, Huang H, Zhu C, et al. Interobserver variations in target delineation in intensity-modulated radiation therapy for nasopharyngeal carcinoma and its impact on target dose coverage[J]. Technol Cancer Res Treat, 2023, 22: 15330338231169592. DOI:10.1177/15330338231169592 |
| [10] |
Men K, Chen X, Zhang Y, et al. Deep deconvolutional neural network for target segmentation of nasopharyngeal cancer in planning computed tomography images[J]. Front Oncol, 2017, 7: 315. DOI:10.3389/fonc.2017.00315 |
| [11] |
Cheng J, Tian S, Yu L, et al. Fully convolutional attention network for biomedical image segmentation[J]. Artif Intell Med, 2020, 107: 101899. DOI:10.1016/j.artmed.2020.101899 |
| [12] |
Bose S, Sur CR, Das R, et al. Dense dilated deep multiscale supervised U-Network for biomedical image segmentation[J]. Comput Biol Med, 2022, 143: 105274. DOI:10.1016/j.compbiomed.2022.105274 |
| [13] |
Punn NS, Agarwal S. Modality specific U-Net variants for biomedical image segmentation: a survey[J]. Artif Intell Rev, 2022, 55(7): 5845-5889. DOI:10.1007/s10462-022-10152-1 |
| [14] |
Li C, Yao G, Xu X, et al. DCSegNet: deep learning framework based on divide-and-conquer method for liver segmentation[J]. IEEE Access, 2020, 8: 1. DOI:10.1109/ACCESS.2020.3012990 |
| [15] |
Nemoto T, Futakami N, Yagi M, et al. Efficacy evaluation of 2D, 3D U-Net semantic segmentation and atlas-based segmentation of normal lungs excluding the trachea and main bronchi[J]. J Radiat Res, 2020, 61(2): 257-264. DOI:10.1093/jrr/rrz086 |
| [16] |
Nodirov J, Abdusalomov AB, Whangbo TK. Attention 3D U-Net with multiple skip connections for segmentation of brain tumor images[J]. Sensors (Basel), 2022, 22(17): 6501. DOI:10.3390/s22176501 |
| [17] |
许卓华, 杨慧, 江舟, 等. 基于3D ResSE-Unet的智能靶区勾画在乳腺癌术后放疗中的应用研究[J]. 中华放射医学与防护杂志, 2023, 43(4): 269-275. Xu ZH, Yang H, Jiang Z, et al. Application of 3D ResSE-Unet-based intelligent delineation of clinical target volume in postoperative radiotherapy for breast cancer[J]. Chin J Radiol Med Prot, 2023, 43(4): 269-275. DOI:10.3760/cma.j.cn112271-20221212-00486 |
| [18] |
Ma Z, Zhou S, Wu X, et al. Nasopharyngeal carcinoma segmentation based on enhanced convolutional neural networks using multi-modal metric learning[J]. Phys Med Biol, 2019, 64(2): 25005. DOI:10.1088/1361-6560/aaf5da |
| [19] |
Liao W, He J, Luo X, et al. Automatic delineation of gross tumor volume based on magnetic resonance tmaging by performing a novel semisupervised learning framework in nasopharyngeal carcinoma[J]. Int J Radiat Oncol Biol Phys, 2022, 113(4): 893-902. DOI:10.1016/j.ijrobp.2022.03.031 |
| [20] |
Li Y, Han G, Liu X. DCNet: Densely connected deep convolutional encoder-decoder network for nasopharyngeal carcinoma segmentation[J]. Sensors (Basel), 2021, 21(23): 7877. DOI:10.3390/s21237877 |
| [21] |
Domoguen J, Manuel JA, Cañal J, et al. Automatic segmentation of nasopharyngeal carcinoma on CT images using efficient UNet-2.5D ensemble with semi-supervised pretext task pretraining[J]. Front Oncol, 2022, 12: 980312. DOI:10.3389/fonc.2022.980312 |
 2024, Vol. 44
2024, Vol. 44


