放疗临床效果取决于对危及器官(OAR)的准确勾画,基于图谱或深度学习的自动分割方法在节省医疗时间和降低医疗资源占用方面被广泛应用[1-2]。然而,在临床实践中应用自动分割软件之前,必须进行彻底的验证和质量保证,因为不准确的分割可能导致剂量分布错误,进而影响临床结果[3-4]。通常通过几何指标来评价图像自动分割的准确性,而计划质量的评价主要依据剂量指标,两者之间存在缺乏相关性的问题[5-9]。要在临床中使用OAR自动分割结果,必须由专家进行校正。但由于几何分割与剂量分布之间缺乏相关性,放疗专家在没有潜在校正如何影响剂量分布的信息下进行校正,可能导致低效或次优的分割校正工作流程。
通过评估专家对自动分割结果的校正范围和类型,以及探讨是否存在系统性偏差和OAR的高变异性区域,可以更深入地了解算法模型在现实应用中的性能,这对于提高自动分割的质量保证至关重要。由于不同的算法(如基于图谱库和深度学习的方法)提供了不同的自动分割结果,临床专家面临着不一致的校正起点。目前,缺乏研究探讨这种不一致性及其与专家勾画差异在放疗计划的剂量、优化和评估方面的显著性影响。本研究旨在定量评估鼻咽癌OAR自动分割轮廓的校正范围和类型,并探究不同源自动分割轮廓对放疗计划剂量分布的影响。
资料与方法1. 患者数据:回顾性选择广州中医药大学金沙洲医院2023年1月1日至2023年6月30日确诊鼻咽癌共50例患者的临床资料。纳入标准:①TNM分期T1-3N0-2M0,分期标准依据中国鼻咽癌临床分期工作委员会,中国鼻咽癌分期2017版[10]。②CT及磁共振(MR)影像资料完整,临床专家完整勾画了靶区及OAR。③患者放疗计划已执行完成。排除标准:①靶区侵犯脑干、脊髓或视通路,导致放疗计划需要在保靶区或保OAR之间权衡。②患者年龄<18岁。③CT影像质量不佳,如口腔伪影过多等。通过纳入及排除筛选,最终选择25例患者,其中男15例,女10例,年龄38~74岁,平均年龄53.8岁。本研究经广州中医药大学金沙洲医院伦理委员会审查批准(JSZ-IEC-SL-KT-20240603),患者均豁免知情同意。
德国西门子SOMATOM CT 2.5 mm层厚扫描,扫描图像分别传送美国瓦里安Eclipse治疗计划系统、Manteia公司深度学习勾画软件AccuContour 3.1及MIM软件公司图谱库软MIM-6.8中。AccuContour和MIM自动分割完成后将结构回传至Eclipse,分别定义为深度勾画(CDL)和图谱勾画(CAT)。一名经过确认能够重复高质量勾画的专家在Eclipse融合CT和磁共振成像(MRI)勾画靶区及OAR[11]。选择脊髓、脑干、视神经、视交叉、腮腺、口腔、下咽和下颌骨8个OAR参与研究,专家勾画OAR结构定义为CEC。该名专家对AccuContour勾画结果予以校正使其临床可接受定义为专家校正深度勾画(CEDL),对MIM勾画结果也进行了相应的校正定义为专家校正图谱勾画(CEAT)。靶区及其他OAR均选择CEC结构复制到上述5套图像中参与后续研究。
2. 计划优化:处方为计划肿瘤靶区体积(PGTV)69.96 Gy,计划靶区体积(PTV)60.06 Gy共33次,基于TrueBeam平台容积旋转调强放疗(VMAT)技术2个共面全弧,6 MV X射线,光子剂量算法(AXB),2.5 mm计算优化网格。优化保证靶区剂量覆盖同时最小化OAR剂量,以PTV处方覆盖95%体积归一。以专家勾画(CEC)优化的专家计划(PEC)为模板,计划参数保持不变,深度勾画(CDL)、专家校正深度勾画(CEDL)、图谱勾画(CAT)和专家校正图谱勾画(CEAT)更换专家勾画(CEC)结构重新优化得到深度计划(PDL)、专家校正深度计划(PEDL)、图谱计划(PAT)和专家校正图谱计划(PEAT),流程见图 1。脊髓、脑干、视神经和视交叉评价最大剂量(Dmax),腮腺、口腔和下咽评价平均剂量(Dmean),下颌骨评价1 cm3最大剂量(D1 cm3)[12]。
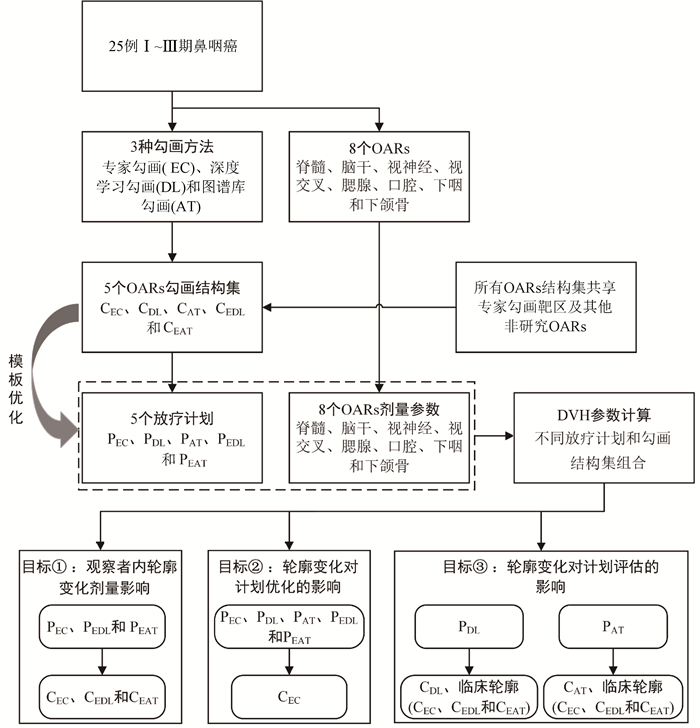
|
注:专家勾画、自动勾画和专家校正的自动勾画5种结构集,生成5种剂量分布。在不同结构集上分析剂量参数,形成3个研究目标,即①观察者内轮廓变化剂量影响。②轮廓变化对计划优化的影响。③轮廓变化对计划评估的影响。CEC、CDL和CAT分别为专家勾画、深度学习和图谱勾画,CEDL、CEAT分别为经专家校正的深度学习和图谱勾画,PEC、PDL、PAT、PEDL、PEAT分别为相应的放疗计划剂量分布 图 1 轮廓可变性剂量学影响工作流程图 Figure 1 Work flow showing of the dosimetric impact of contour variability |
3. 计算轮廓间戴斯相似性系数(DSC),反映轮廓几何指标差异[13]。评估几何指标与剂量体积参数(DVH)相关性。包括以下3个步骤:①专家勾画(CEC)为参考轮廓。②自动勾画(CDL、CAT)和专家校正自动勾画(CEDL和CEAT)为替代轮廓反映参考轮廓几何变化。③计算参考轮廓与替代轮廓几何指标DSC,其计算方法见式(1):
| $ \begin{gathered} \mathrm{D}SC=\frac{2 \times\left|\mathrm{C}_{\mathrm{EC}} \cap \mathrm{C}_i\right|}{\left|\mathrm{C}_{\mathrm{EC}}\right|+\left|\mathrm{C}_i\right|}, \\ \mathrm{i} \in[\mathrm{DL}, \quad \mathrm{EDL}, \quad \mathrm{AT},\quad \mathrm{EAT}] \end{gathered} $ | (1) |
式中,i为对应结构集编号;| |为集合中的元素数量;∩为2个集合的交集,即2个集合共有的元素。
4. 研究目标:本研究设定3个研究目标(图 1)。目标1为考虑观察者内轮廓变化对剂量影响。专家勾画(CEC)、专家校正深度勾画(CEDL)和专家校正图谱勾画(CEAT)为不同时间节点上同一专家勾画或校正的临床可接受结构集,PEC、PEDL和PEAT为对应剂量分布。剂量分析除考虑本身结构集DVH参数外,还考虑其他两个结构集DVH参数,每个病例形成了9个剂量分布和结构集组合,组合波动范围(ΔR)定义为该患者观察者内轮廓变化导致的剂量变化,见式(2),计算患者ΔR平均值和标准差。
| $ \begin{gathered} \varDelta R=\left[S_2-S_1, S_9-S_1\right], S={sort}\left(D\left(C_i, \mathrm{P}_j\right)\right), \\ i, j \in[\mathrm{EC}, \mathrm{EDL}, \mathrm{EAT}] \end{gathered} $ | (2) |
式中,D(Ci, Pj)为Pj计划在Ci结构集上的剂量分布;sort对9种组合升序排序。
目标2为考虑轮廓变化对计划优化影响。5个剂量分布均在专家勾画(CEC)上评价。对每个DVH参数,计算自动计划(PDL、PAT)和专家校正自动计划(PEDL、PEAT)与专家计划PEC剂量之差(ΔD),详见式(3)。计算OAR与PTV的重叠体积直方图(OVH),判断OVH参数与ΔD是否存在线性相关性[12]。为了研究专家校正对自动分割结构剂量学影响,分析ΔD之间是否有统计差异。
| $ \begin{gathered} \Delta D=D\left(\mathrm{C}_{\mathrm{EC}}, \mathrm{P}_i\right)-D\left(\mathrm{C}_{\mathrm{EC}}, \mathrm{P}_{\mathrm{EC}}\right), \\ i \in[\mathrm{DL}, \mathrm{AT}, \mathrm{EDL}, \mathrm{EAT}] \end{gathered} $ | (3) |
目标3为考虑轮廓变化对计划评估影响。判断PDL或PAT在自动分割结构集CDL或CAT上DVH参数是否落在临床可接受结构集(CEC、CEDL和CEAT)的DVH范围内,见公式(4),旨在研究自动分割轮廓放疗计划是否存在与临床可接受轮廓类似DVH参数值,探讨图像自动分割到计划自动优化,再到自动评估的放疗全自动化工作流程的潜力。
| $ \begin{array}{l} \left\{\begin{array}{l} 1, \min \left(D\left(\mathrm{C}_i, \mathrm{P}_j\right)\right) \leqslant D\left(\mathrm{C}_j, \mathrm{P}_j\right) \leqslant \max \left(D\left(\mathrm{C}_i, \mathrm{P}_j\right)\right) \\ 0, \text { 其他情况 } \end{array}\right.\\ i \in[\mathrm{EC}, \mathrm{EDL}, \mathrm{EAT}], j \in[\mathrm{DL}, \mathrm{AT}] \end{array} $ | (4) |
5. 统计学处理:使用EvaluateSegmentation软件计算几何指标DSC[13]。DSC及剂量数据(ΔR,ΔD)采用夏皮洛—威尔克检验(Shapiro-Wilk,SW)方法进行正态分布检验,符合正态分布的计量资料以x±s表示,不符合正态分布的计量资料以中位数四分位间距M(P25,P75)表示。对几何和剂量差异予以统计学检验,2组数据符合正态分布采用Student′s t检验,不符合正态分布则采用威尔科克森(Wilcoxon)秩和检验。统计分析采用SPSS 25软件,P<0.05为差异具有统计学意义。
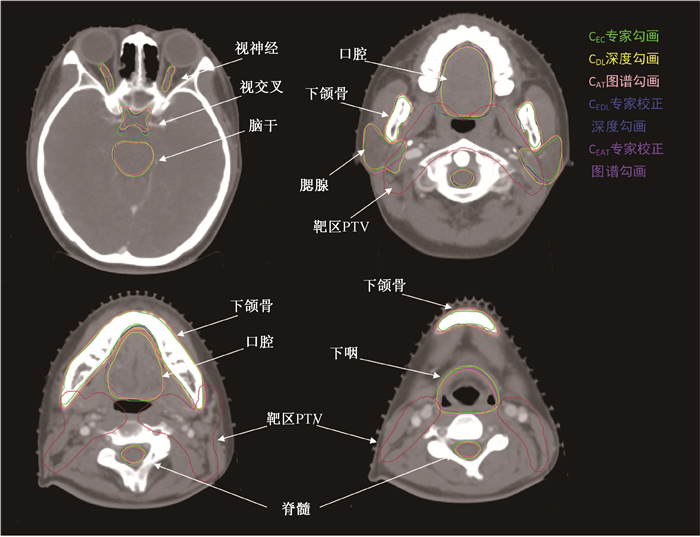
结果1. 几何分割指标:图 2举例说明了自动勾画轮廓CDL和CAT,专家校正自动勾画轮廓CEDL和CEAT及与专家勾画轮廓CEC的对比。对纳入研究的8个OAR,专家逐层进行校正。简单几何结构(如脊髓或下颌骨),自动勾画和专家勾画观察到良好的一致性。在某些结构(如视交叉、或下咽)的头尾部断层中,与专家勾画参考相比,轮廓差异较大。为了说明OAR相对于靶区的位置,还显示了PTV轮廓。专家对CDL校正速度快于CAT,脊髓、脑干和下颌骨校正时间一般<2 min,而视交叉、腮腺和下咽的校正时间则>3 min。
|
图 2 自动分割与专家勾画的不确定性示例 Figure 2 Example showing the uncertainties of automatic segmentation and expert delineation |
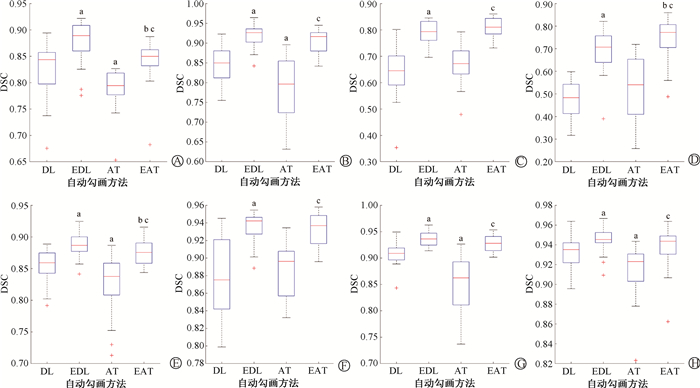
图 3为OAR勾画组合与专家勾画DSC箱图,视交叉最小为0.51±0.12,视神经为0.65±0.08,低于其他OAR的0.86±0.04。视神经、视交叉和口腔两种算法DSC相当,其他OAR深度学习DSC优于图谱库(t=4.87,P<0.05),表明深度学习在当前OAR勾画性能优于图谱库。两种自动勾画(CDL和CAT)方法校正前后,其与专家勾画DSC差异均具有统计学意义(t=-6.16、-7.51,P<0.05),平均DSC分别提高了7.61%和10.69%。校正后,脊髓、视交叉和腮腺两种算法DSC差异具有统计学意义(t=3.11、-2.29、2.41,P<0.05),而其他OAR两种算法DSC相当,均为0.87±0.03。表明专家对图谱库勾画校正较多而对深度学习勾画校正较少,提高了校正后图谱库与专家勾画几何相似度。视交叉经校正后的DSC仍<0.75且校准差>0.1,表明对勾画较难小体积OAR,即使同一专家对同一患者图像,参考不同勾画起点(深度学习和图谱库)予以校正,最终的勾画结果仍存在较大几何差异。
|
注:DSC.戴斯相似系数;DL.深度勾画与专家勾画;EDL.专家校正深度勾画与专家勾画;AT.图谱勾画与专家勾画;EAT.专家校正图谱勾画与专家勾画。红色+为离群值。a与DL比较,t=-4.28、2.56、-7.14、3.15、-7.72、-8.83、-5.02、2.56、-6.71、-5.64、4.96、-3.13、3.02,P<0.05;b与EDL比较,t=3.11、-2.29、2.41,P<0.05;c与AT比较,t=-4.75、-7.16、-8.44、-6.54、-4.75、-6.64、-6.99、-3.32,P<0.05 图 3 不同危及器官自动勾画组合与专家勾画戴斯相似系数箱图 A.脊髓;B.脑干;C.视神经;D.视交叉;E.腮腺;F.口腔;G.下咽;H.下颌骨 Figure 3 Boxplots showing the dice similarity coefficients between contours of automatic delineation combinations and expert contours of organs at risk A. Spinal cord; B. Brain stem; C. Optic nerve; D. Optic chiasm; E. Parotid gland; F. Oral cavity; G. Hypopharynx; H. Mandible |
2. 观察者内临床轮廓变化剂量影响:根据式(2),表 1列出25例患者的观察者内临床轮廓(CEC,CEDL和CEAT)剂量组合波动范围ΔR,其中ΔR最大为视交叉和视神经Dmax,分别达到0~35.56 Gy和0~27.3 Gy,平均值(3.96±6.02)和(3.97±4.79)Gy,而ΔR最小为下咽Dmean和下颌骨D1 cm3,平均值分别为(0.81±0.55)和(0.86±0.59)Gy。视神经和视交叉ΔR(0 ~35.56 Gy)显著大于其他5个OAR(0~10 Gy),这与视通路DSC较小且与靶区距离位置差异较大观察一致。此外,评价Dmax器官的剂量变化范围大于评价Dmean器官。
|
|
表 1 观察者中25例患者的临床轮廓剂量变化范围(Gy) Table 1 Dose ranges of inner-observer clinical contours of 25 patients(Gy) |
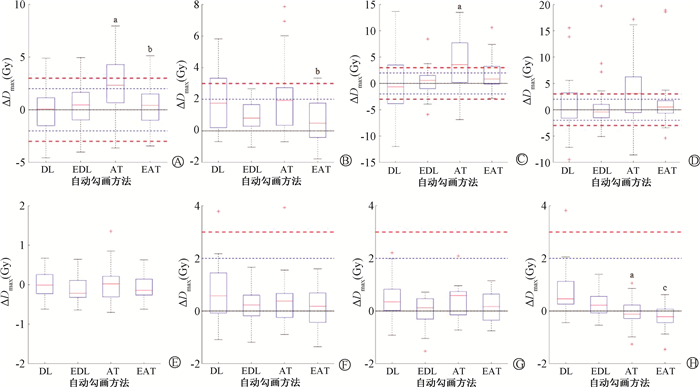
3.轮廓变化对计划优化影响:参考调强剂量验证要求[15],设置ΔD的±2 Gy(图 4中蓝虚线)和±3 Gy阈值(图 4中红虚线)。腮腺ΔDmean均在±2 Gy内,下咽ΔDmean均在±3 Gy内,98%在±2 Gy内。而口腔ΔDmean和下颌骨ΔD1 cm3过±3 Gy的比例分别只占2%和1%。脊髓、脑干、视神经和视交叉的剂量变化范围较大,分别有22%,14%,46%和42%的ΔDmax超过±3 Gy,其中视神经和视交叉波动范围更广。OAR经校正后与专家勾画剂量差值均减小,尤其对于图谱库分割方法,经专家校正后脊髓和脑干ΔD范围缩小有统计学意义(t=2.57、2.41,P<0.05)。视神经和视交叉校正后ΔD范围超过±3 Gy分别下降了16%和14%。脊髓、视神经和下颌骨两种自动分割轮廓(CDL和CAT)ΔD差异有统计学意义(t=-3.17、-2.69、3.69,P<0.05),而下颌骨勾画在经专家校正后,其剂量差异仍有统计学意义(t=3.44,P<0.05),但变化范围较小。
|
注:ΔD表示剂量差值,由公式(3)计算;DL.深度勾画;EDL.专家校正深度勾画;AT.图谱勾画;EAT.专家校正图谱勾画。图中蓝虚线为±2 Gy阈值,红虚线为±3 Gy阈值。红色+为离群值。a与DL比较,t=3.17、-2.69、3.69,P<0.05;b与AT比较,t=2.57、2.41,P<0.05;c与EDL比较,t=3.44,P<0.05 图 4 不同危及器官自动及校正勾画优化的放疗计划剂量分布均在专家勾画轮廓上评价,计算其与专家勾画优化的放疗计划剂量分布的差值(ΔD) A.脊髓;B.脑干;C.视神经;D.视交叉;E.腮腺;F.口腔;G.下咽;H.下颌骨 Figure 4 The dose distributions of the optimized plan with auto-segmented and adjusted contour of different organs at risk were evaluated against the expert contour. The difference in dose (ΔD) between these contours and the expert contour was calculated to evaluate the impact of contour variation on plan optimization A. Spinal cord; B. Brain stem; C. Optic nerve; D. Optic chiasm; E. Parotid gland; F. Oral cavity; G. Larynx; H. Mandible |
对于评价Dmax OAR,当OVH平均距离>2.0 cm时,97.3% ΔDmax在±5 Gy范围内;而当OVH平均距离>3.5 cm时,ΔDmax均在±3 Gy范围内;当OVH平均距离>4.0 cm时,ΔDmax均在±2 Gy范围内,但此种情况数据较少。对于评价Dmean OAR,OVH平均距离所有范围内,99.5% ΔDmean在±3 Gy范围内,而当OVH平均距离>1.5 cm时,ΔDmean均在±2 Gy范围内。
4.轮廓变化对计划评估影响:表 2为25例患者深度计划PDL或图谱计划PAT的DVH参数落在临床勾画(CEC,CEDL和CEAT)剂量范围内的个数。对于PDL,视交叉Dmax最小,只有6例患者的剂量落在临床轮廓剂量范围内;而下颌骨D1 cm3最大,有19例患者剂量在临床轮廓剂量范围内;所有OAR的平均患者数为12 ± 4。对于PAT,腮腺Dmean最少,7例患者剂量落在临床轮廓剂量范围内;而下颌骨D1 cm3最大,有21例患者剂量在临床轮廓剂量范围内;所有OAR的平均患者数为13 ± 5。两种自动轮廓在脊髓、视神经和视交叉Dmax、口腔Dmean及下颌骨D1 cm3的剂量差异具体统计学意义(t=-4.24、-3.99、-3.16、3.51,P<0.05)。
|
|
表 2 自动勾画轮廓的剂量分布DVH参数落在临床勾画剂量范围内的患者数 Table 2 Number of patients whose automatic segmentation contour dose distribution DVH parameters fall within the clinically contour dose ranges |
讨论
小体积OAR的准确分割是图像自动分割难点之一[16],本研究发现,视神经和视交叉深度学习和图谱库勾画DSC分别只有0.65±0.08和0.51±0.15,而其他OAR的DSC达0.86±0.04。经专家校正后,视神经和视交叉的DSC分别提高到0.80±0.05和0.72±0.10,但仍低于其他OAR的0.87±0.03。视神经和视交叉在校正后,CEDL与CEAT的DSC只有0.79±0.06和0.74±0.12,说明小体积OAR勾画存在较大不确定性,即使同一专家对同一患者图像,参考不同勾画起点(深度学习或图谱库)予以校正最终于勾画结果也存在较大几何差别。从本研究勾画组合DSC来看,深度学习算法性能要优于图谱库。专家校正后,两种方法的DSC相差无几,说明专家对深度学习勾画校正较少而对图谱库勾画校正较多,提高了图谱库校正后轮廓几何相似度。两个自动分割结果虽然达到了与专家勾画相当的DSC,但两者间的DSC仍然较小,这可能了反映出用DSC展示结构差异的不足之处。因此在下一步研究可以考虑使用更能体现轮廓一致性的指标来评估几何差异,并研究这些差异与剂量差异之间的关联性。
在观察者内临床轮廓变化中,视通路器官的剂量变化显著大于其他OAR:一方面视通路器官勾画在几何上存在较大差异;其次,鼻咽癌患者靶区长度各异,视通路器官与靶区的最小距离(0.57±0.43)cm标准差显著大于脊髓(0.99±0.20)cm及脑干(0.56±0.12)cm,增加了剂量变化的范围。大多数视通路器官在空间位置上与靶区属于进出方向,相较于其他OAR左右或上下方向,直线加速器在进出方向剂量跌落更为迅速。OAR位置在进出方向差一个CT扫描层,钨门或多叶光栅(MLC)运动可能会形成全遮挡或全打开的形状差异。此外,计划优化过程中有用到其他辅助结构,本研究中将其他辅助结构保持一致,仅危及器官进行替换,是否会放大结构变化对剂量变化的贡献需要进一步探讨。
在探讨轮廓变化对计划优化的影响时,发现对于评价Dmean腮腺、口腔和下咽及D1 cm3下颌骨,98% ΔD在±3 Gy范围内,腮腺ΔD均在±2 Gy范围内,说明这几个OAR当前自动分割方法达到较高的精度,对计划优化的影响较小,因此在审核时,专家只需快速浏览自动分割结果,关注是否有缺省层面。然而对距离靶区较近的脊髓和脑干或自动分割精度欠缺视神经和视交叉,仍然会导致较大ΔD,其中有超过40%的ΔD超过±3 Gy。尽管脊髓和脑干DSC大于视通路,其ΔD却与视通路一样,而下颌骨DSC大于口腔,其ΔD也比腮腺小,说明相同DSC趋势会导致不一样剂量差异(更高或更低),暗示几何指标和剂量指标缺乏一致的相关性。DSC与ΔD皮尔逊相关系数绝对平均值0.21±0.16,意味着简单使用几何指标估计剂量效应不可行,剂量变化还与OAR与靶区相对位置和距离,OAR形状和体积等因素有关[17-20]。找到面向临床具有剂量预测效应的特征,在剂量引导下进行器官分割,提高分割对剂量的稳健性,解决远端离群值,聚焦于高剂量梯度的位置是一个有前景的研究方向。
根据OVH与ΔD关系,建议对于脊髓、脑干、视神经和视交叉评价Dmax的OAR,当OVH平均距离>4.0 cm时,ΔD在±2 Gy内,此时自动分割结果只需快速审核和恰当调整;当OVH最小距离<2.0 cm时ΔD变化超过±5 Gy,自动分割结果需要详尽核查校正。对于腮腺、口腔和下咽评价Dmean OAR及下颌骨,OVH范围内ΔD均在±3 Gy内,OVH平均距离>1.5 cm时,ΔD在±2 Gy内,因此OAR自动分割结果只需快速审核,用于确定轮廓大致正确位置。通过治疗计划系统开放的应用程序接口,建立判定规则,在专家校正之前通过脚本计算OVH等因子会显著提高工作效率。
本研究结果发现,观察者内临床轮廓剂量学效应与轮廓变化对计划评估的剂量影响相当,因此使用自动分割轮廓直接优化,与在实际临床中使用的临床勾画优化相比,不会导致更大剂量差异。使用自动分割结果(CDL和CAT)进行剂量评价时,深度学习和图谱库分别有49.52%±16.96%和52.52%±19.28%患者落在临床勾画剂量范围内。虽然剂量效应可能并不显著,但准确OAR勾画仍然是计划设计重要前提,不能因为自动分割误差不显著影响剂量分布就认为其临床可接受。当前阶段自动分割技术尚不成熟,可以作为辅助工具提高工作效率、节省医疗资源并保证放疗同质化水平,但不能以自动分割直接用于计划优化,特别是小体积器官(如视神经、视交叉等)和距离靶区较近评价Dmax器官(如脊髓、脑干等),仍需要认为检查校正后方可用于计划优化。通过本研究,放疗医生可以获得关于剂量的信息来审查和纠正自动分割的误差范围,从而在临床实践中做出判断。
总之,本研究对深度学习和图谱库的自动分割OAR与专家勾画的结果进行了比较。发现即便经过专家校正,参考不同来源的自动勾画结果最终仍显示出一定的几何差异。专家的校正操作减少了自动勾画轮廓对治疗计划优化的影响。重叠体积直方图(OVH)的平均距离是判定剂量差异的一个重要指标。对于体积较小且靠近靶区的串行器官,在临床应用自动分割轮廓之前需进行细致的校正。
利益冲突 无
作者贡献声明 张丽媛负责设计研究方案,研究实施及论文撰写;胡金炎参与数据收集及论文修改;辜石勇参与放疗计划设计;魏夏平提供技术指导
| [1] |
Seo H, Badiei Khuzani M, Vasudevan V, et al. Machine learning techniques for biomedical image segmentation: An overview of technical aspects and introduction to state-of-art applications[J]. Med Phys, 2020, 47(5): e148-e167. DOI:10.1002/mp.13649 |
| [2] |
McQuinlan Y, Brouwer CL, Lin Z, et al. An investigation into the risk of population bias in deep learning autocontouring[J]. Radiother Oncol, 2023, 186: 109747. DOI:10.1016/j.radonc.2023.109747 |
| [3] |
Ye X, Guo D, Ge J, et al. Comprehensive and clinically accurate head and neck cancer organs-at-risk delineation on a multi-institutional study[J]. Nat Commun, 2022, 13(1): 6137. DOI:10.1038/s41467-022-33178-z |
| [4] |
Duan J, Bernard ME, Castle JR, et al. Contouring quality assurance methodology based on multiple geometric features against deep learning auto-segmentation[J]. Med Phys, 2023, 50(5): 2715-2732. DOI:10.1002/mp.16299 |
| [5] |
Sherer MV, Lin D, Elguindi S, et al. Metrics to evaluate the performance of auto-segmentation for radiation treatment planning: A critical review[J]. Radiother Oncol, 2021, 160: 185-191. DOI:10.1016/j.radonc.2021.05.003 |
| [6] |
Leonardi MC, Pepa M, Luraschi R, et al. The dosimetric impact of axillary nodes contouring variability in breast cancer radiotherapy: An AIRO multi-institutional study[J]. Radiother Oncol, 2022, 168: 113-120. DOI:10.1016/j.radonc.2022.01.004 |
| [7] |
Vaassen F, Hazelaar C, Canters R, et al. The impact of organ-at-risk contour variations on automatically generated treatment plans for NSCLC[J]. Radiother Oncol, 2021, 163: 136-142. DOI:10.1016/j.radonc.2021.08.014 |
| [8] |
Wong J, Fong A, McVicar N, et al. Comparing deep learning-based auto-segmentation of organs at risk and clinical target volumes to expert inter-observer variability in radiotherapy planning[J]. Radiother Oncol, 2020, 144: 152-158. DOI:10.1016/j.radonc.2019.10.019 |
| [9] |
Poel R, Rüfenacht E, Hermann E, et al. The predictive value of segmentation metrics on dosimetry in organs at risk of the brain[J]. Med Image Anal, 2021, 73: 102161. DOI:10.1016/j.media.2021.102161 |
| [10] |
中国鼻咽癌临床分期工作委员会. 中国鼻咽癌分期2017版(2008鼻咽癌分期修订专家共识)[J]. 中华放射肿瘤学杂志, 2017, 26(10): 1119-1125. DOI:10.3760/cma.j.issn.1004-4221.2017.10.002 |
| [11] |
Brouwer CL, Steenbakkers RJ, Bourhis J, et al. CT-based delineation of organs at risk in the head and neck region: DAHANCA, EORTC, GORTEC, HKNPCSG, NCIC CTG, NCRI, NRG Oncology and TROG consensus guidelines[J]. Radiother Oncol, 2015, 117(1): 83-90. DOI:10.1016/j.radonc.2015.07.041 |
| [12] |
Bentzen SM, Constine LS, Deasy JO, et al. Quantitative analyses of normal tissue effects in the clinic (QUANTEC): an introduction to the scientific issues[J]. Inter J Radiat Oncol Biol Phys, 2010, 76(3Suppl): S3-S9. DOI:10.1016/j.ijrobp.2009.09.040 |
| [13] |
Taha AA, Hanbury A. Metrics for evaluating 3D medical image segmentation: analysis, selection, and tool[J]. BMC Med Imaging, 2015, 15: 29. DOI:10.1186/s12880-015-0068-x |
| [14] |
Wu B, Pang D, Simari P, et al. Using overlap volume histogram and IMRT plan data to guide and automate VMAT planning: a head-and-neck case study[J]. Med Phys, 2013, 40(2): 021714. DOI:10.1118/1.4788671 |
| [15] |
Miften M, Olch A, Mihailidis D, et al. Tolerance limits and methodologies for IMRT measurement - based verification QA: Recommendations of AAPM Task Group No. 218[J]. Med Phys, 2018, 45(4): e53-e83. DOI:10.1002/mp.12810 |
| [16] |
Ren XH, Xiang L, Nie D, et al. Interleaved 3D-CNN s for joint segmentation of small-volume structures in head and neck CT images[J]. Med phys, 2018, 45(5): 2063-2075. DOI:10.1002/mp.12837 |
| [17] |
McQuinlan Y, Brouwer CL, Lin Z, et al. An investigation into the risk of population bias in deep learning autocontouring[J]. Radiother Oncol, 2023, 186: 109747. DOI:10.1016/j.radonc.2023.109747 |
| [18] |
蒋璠, 吴昊, 张健, 等. 基于最优自动分割的放疗计划剂量学评估[J]. 中华放射肿瘤学杂志, 2017, 26(4): 423-428. Jiang F, Wu H, Zhang J, et al. A dosimetric evaluation of treatment planning based on optimal autosegmentation[J]. Chin J Radiat Oncol, 2017, 26(4): 423-428. DOI:10.3760/cma.j.issn.1004-4221.2017.04.012 |
| [19] |
Mackay K, Bernstein D, Glocker B, et al. A review of the metrics used to assess auto-contouring systems in radiotherapy[J]. Clin Oncol (R Coll Radiol), 2023, 35(6): 354-369. DOI:10.1016/j.clon.2023.01.016 |
| [20] |
van Rooij W, Dahele M, Ribeiro Brandao H, et al. Deep learning-based delineation of head and neck organs at risk: geometric and dosimetric evaluation[J]. Int J Radiat Oncol Biol Phys, 2019, 104(3): 677-684. DOI:10.1016/j.ijrobp.2019.02.040 |
 2024, Vol. 44
2024, Vol. 44


