2. 南华大学附属第二医院放射治疗科, 衡阳 421001;
3. 南华大学衡阳医学院, 衡阳 421001;
4. 南华大学附属第一医院肿瘤科, 衡阳 421001
2. Department of Radiation Oncology, The Second Affiliated Hospital, Hengyang Medical School, University of South China, Hengyang 421001, China;
3. Hengyang Medical School, University of South China, Hengyang 421001, China;
4. The First Affiliated Hospital, Hengyang Medical School, University of South China, Hengyang 421001, China
非小细胞肺癌(non-small cell lung cancer, NSCLC)是最为常见的恶性肿瘤,发病率和致死率均居第一位[1]。我国NSCLC患者的表皮生长因子受体(epidermal growth factor receptor, EGFR)突变率高达30%~40%,突变患者预后较野生型差[2-3]。EGFR外显子19缺失(19Del)和外显子21L858R突变是两个最主要的敏感突变,分别占所有EGFR突变的54.5%和36.1%[4]。针对EGFR突变患者,靶向治疗如络氨酸酶抑制剂(tyrosine kinase inhibitors, TKIs)能显著改善其无进展生存期和总生存期[5]。然而不同的EGFR突变亚型对于TKIs治疗的敏感性不同,19Del突变患者更适合接受二代和三代TKIs治疗,而21L858R突变患者更适合接受一代和二代TKIs联合单克隆抗体类靶向药物治疗[6]。因此,明确EGFR突变状态和突变亚型对于后续治疗十分重要。
目前,临床常用手术切除或穿刺活检等有创方式取得标本行病理分子检测,对于丧失手术机会的晚期NSCLC患者或不具备穿刺条件的人群往往无法进行,且肿瘤的空间异质性较强,所取标本差异性较大[7-8]。影像组学是近年来新兴的定量影像分析技术,通过量化影像内的异质性,进而以无创的方式实现精准诊断、预后预测[9-10]。本研究拟探讨基于治疗前胸部平扫CT影像组学特征、临床特征,并结合机器学习算法构建预测EGFR突变模型,评估预测EGFR突变状态和突变亚型的预测精度,以指导临床决策。
资料与方法1. 实验设计:本研究整体实验设计如图 1所示。本研究中所涉及患者采用连续入组方式,选取2014年4月1日至2020年5月31日在南华大学附属第一医院(NH1, 83例)和南华大学附属第二医院(NH2, 326例)初治的肺癌患者。纳入标准:①经手术标本或活检病理证实的非小细胞肺癌患者。②治疗前均行胸部平扫CT扫描和EGFR基因检测。③无其他原发恶性肿瘤病史。排除标准:①影像资料不完整或图像质量较差(87例)。②原发灶模糊,无法准确勾画肿瘤边界(30例);③除19Del和21L858R以外的EGFR突变(12例)。

|
注:NSCLC. 非小细胞肺癌;EGFR.表皮生长因子受体 图 1 整体研究路线图 Figure 1 Overall workflow of this study |
本研究中,EGFR突变状态采用下一代基因测序(NGS)技术进行检测,取材样本为外周血和/或肿瘤组织,最终入组患者280例,其中136例为EGFR突变患者(19Del突变64例,21L858R突变72例),两个医院的患者分别作为验证组(NH1)和训练组(NH2),验证组和训练组之间差异无统计学意义,一般临床资料如表 1所示。
|
|
表 1 入组患者的一般临床资料表 Table 1 General clinical characteristics of enrolled patients |
2. 检查方法:南华大学附属第一医院和第二医院的患者分别采用美国GE公司CT扫描仪(Discovery CT590 RT, GE Medical System, Netherlands)和荷兰飞利浦CT扫描仪(Brilliance iCT 256, Philip Medical System, Netherlands)获取胸部平扫CT图像,具体扫描协议如下:管电压120 kVp,管电流为自动mAs,分辨率(像素矩阵)512 × 512,像素尺寸0.625 mm × 0.625 mm,层厚3 mm。
3. 肿瘤勾画、影像组学特征提取:所有患者的CT图像在3D Slicer软件4.10版本中勾画原发大体肿瘤区域(gross tumor volume, GTV),由两位高年资影像医师和肿瘤医师在肺窗重建(窗位= -600 HU,窗宽=1 200 HU)图像中进行单独勾画并交叉检查,因此不同勾画提取特征的一致性及可重复性评估在本研究中不涉及。影像组学特征提取采用python编码平台中PyRadiomics 3.0.1版本代码包批量提取计算。每个患者提取851个影像组学特征,包括:①像素强度及灰度直方图特征IH。②灰度共生矩阵特征GLCM。③灰度游程矩阵特征GLRLM。④灰度区域大小矩阵特征GLSZM。⑤灰度邻域差矩阵特征NGTDM。⑥灰度依赖矩阵GLDM。⑦小波变换特征WAVELET。
4. 影像组学特征选择和模型建立与评估:影像组学特征选择采用两步法:第一步为相关性分析,采用Spearman相关分析每个特征和EGFR突变状态及突变亚型之间的相关性,相关性系数RS>0.75的特征纳入第二步;第二步为特征筛选,采用RELIEFF算法(relief特征选择算法的改进,基本原理:依据特征对目标变量的贡献大小赋予其相应的权重,并根据权重构成特征子集并对特征进行排序)筛选具有预测意义的特征。为了避免模型过拟合,最终纳入建模的特征应为患者样本数量的1/10至1/3[11]。
随后,采用序贯建模方式建立两个预测模型,其中模型1为EGFR突变状态预测模型,模型2为EGFR突变亚型预测模型。采用支持向量机(support vector machine, SVM)算法和径向基函数(radial basis function, RBF)方法建立预测模型,模型训练过程中采用十折交叉验证,重复1 000次避免过拟合和获得更好的预测性能。然后,使用验证集评估预测已建立的模型性能。使用受试者工作特征曲线下面积(area under receiver operating curve, AUC-ROC)评估模型预测性能。
本研究共建立3组模型,分别为:临床模型、影像组学模型和临床-影像组学模型,每组分别包含以上两个序贯模型(模型1预测EGFR突变状态,模型2预测EGFR突变亚型),比较预测精度以评估模型的效能。
5. 统计学处理:本研究中,Spearman相关分析采用R软件(版本:3.3.1)中的“psych”包。RELIEFF特征选择(特征选择算法库,版本:6.2.1)、模型训练和模型评估均在Matlab(版本:R2022b,Mathworks,USA)软件中进行。组间比较采用Wilcoxon rank-sum秩和检验。P < 0.05为差异有统计学意义。
结果1. 特征选择结果:临床特征选择具有预测意义的特征纳入后续模型建立过程,根据表 1,性别、吸烟史和吸烟数量对EGFR突变有预测能力。影像组学特征选择分为两步:第一步特征Spearman相关性分析中,分别有76、43个特征和EGFR突变状态、突变亚型直接具有相关性(RS>0.75);第二步RELIEFF特征选择用于选择最具预测意义的特征,在每个特征类别下选择权重排名前3位的特征用于输入SVM模型中,模型1和模型2各有21个输入影像组学特征,Spearman相关系数(RS)和RELIEFF权重(WR)如表 2所示。IH-Skewness、GLCM-Correlation、NGTDM-Coarseness、NGTDM-Busyness 4个特征对EFGR突变状态和突变亚型中均有预测能力。
|
|
表 2 影像组学两步特征选择最终结果 Table 2 Final results for radiomics features using two-step feature selection method |
2. 预测模型性能:序贯模型分别结合临床特征、影像组学特征、临床-影像组学特征,构建3组预测模型。其中,影像组学特征中7个特征类别各前3个最具预测意义的特征共计21个纳入模型建立过程。每组各包含模型1和模型2两个模型,模型1用于预测EGFR突变状态;模型2用于预测EGFR突变亚型。图 2为3组模型的ROC曲线,对应AUC数值如表 3和表 4所示,无论是训练组和验证组,临床-影像组学模型均取得较好的预测能力。
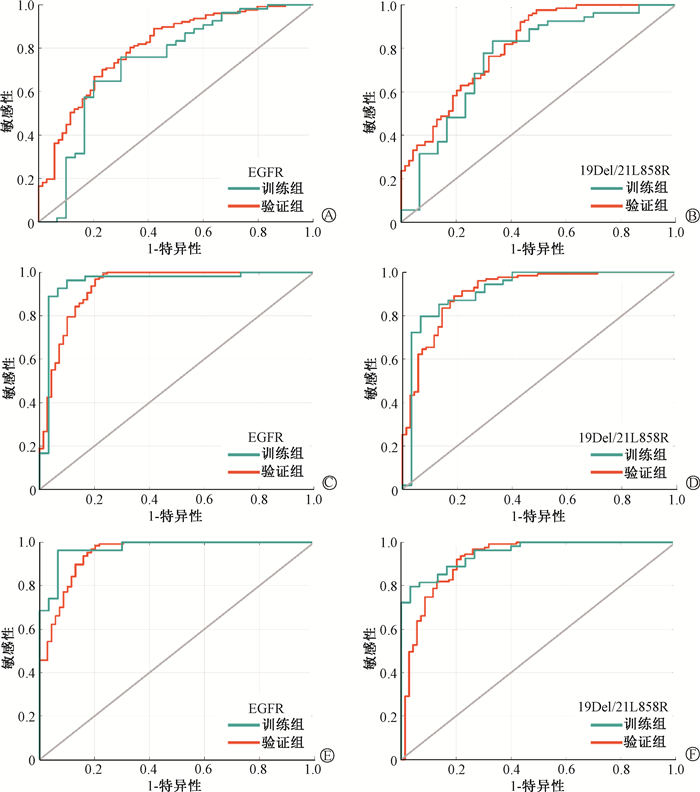
|
A、C、E为EGFR突变预测模型,B、D、F为19Del/21L858R突变亚型预测模型 图 2 3组模型(临床、影像组学、临床-影像组学)的ROC曲线图 A, C, E are prediction models of EGFR mutation; B, D, F are prediction models of EGFR subtype Figure 2 ROCs of three groups of models (clinical, radiomics, clinical-radiomics) |
|
|
表 3 3组预测模型中模型1预测EGFR突变状态的预测性能 Table 3 Prediction performance of model 1 for predicting EGFR mutation status among three groups of models |
|
|
表 4 三组预测模型中模型2预测EGFR突变亚型的预测性能 Table 4 Prediction performance of model 2 for predicting EGFR mutation status among three groups of models |
讨论
EGFR突变在我国肺癌患者中占比较高,为靶向治疗药物的研发和应用提供了可能。其中,外显子19和21突变亚型19Del、21L858R约占所有突变的90%,且两种突变亚型的治疗方式不尽相同,需分而治之[4, 12]。因此,明确NSCLC患者的突变状态和亚型十分重要。肿瘤的异质性决定了活检病理取材无法全面、准确地反映出病灶的完整信息。目前基于CT影像组学技术用于预测NSCLC患者EGFR基因突变的研究已有广泛报道,但CT影像组学预测EGFR突变亚型的研究报道较少[13-15]。以影像组学、人工智能技术为代表的无创诊断方法在基因诊断、分子分型中的作用不可忽视,本研究构建了序贯模型用于预测EGFR突变状态和突变亚型,结果证明了治疗前胸部平扫CT影像组学特征结合机器学习算法能够完成其精准预测,具有较好的临床诊断效能。
既往研究较多地解决了EGFR突变状态预测的临床问题,相关研究表明影像组学特征结合相关临床风险因子能够提升预测能力,但是提升幅度局限在10%以内[16-18]。同时,在提升有限预测精度的同时,引入一些临床因素会增加模型的复杂度,并可能导致模型的过拟合,且临床实践中一些主观性临床评价因素也会导致结果不可靠。为了证实这一观点,本研究使用具有预测效能的临床特征构建了临床模型,并将临床特征和影像组学特征结合构建了临床-影像组学模型,并和影像组学模型进行了比较。同时,本研究摒弃了先前广泛使用的可能会造成重要信息丢失的最小绝对收缩和选择算子方法(least absolute shrinkage and selection operator, LASSO),通过两步特征选择算法和序贯建模方式实现影像组学模型的高可靠性和高鲁棒性。结果显示,在训练集和验证集预测性能中,临床-影像组学模型均高于其他两种模型。相较于影像组学模型,临床-影像组学的预测性能提升在5%以内,预测EGFR突变状态AUC在训练集中达到了0.956(95%CI:0.912~1.000),外部验证集的AUC达到了0.961(95%CI:0.924~0.998),验证集的结果也证明未出现过拟合现象,高于同类研究所报道的预测精度[19-21]。
关于影像组学特征预测EGFR突变亚型预测的临床问题,部分研究基于PET代谢影像结合机器学习算法构建预测模型[22-23],另一部分研究基于CT解剖影像结合机器学习算法构建预测模型[24-26]。虽然PET代谢影像和CT解剖影像的组学特征均能够用于EGFR突变亚型预测,然而PET影像昂贵的医疗成本限制了临床广泛使用。本研究中所建立的临床-影像组学模型在既往CT影像组学研究的基础上,增加了鲁棒的特征选择方法、临床特征数据和外部数据验证保证了所建立预测模型的可靠性,预测性能较此类研究也有较大幅度提升[24-26]。
本研究仍有一些不足之处:首先,借鉴以往发表研究的经验,增强CT应能更好地预测NSCLC患者的临床问题,但由于回顾性研究的限制,只有部分患者具有增强CT;其次,由于其他类型的EGFR突变数量较少,本研究中的EGFR亚型仅涉及19Del和21L858R。
综上所述,基于治疗前平扫CT影像组学和临床特征能够预测NSCLC患者EGFR突变状态和突变亚型,在EGFR突变筛查中能够提供较为可靠的参考,可以为临床医师提供治疗指导决策。
利益冲突 无
作者贡献声明 胡江、贺睿敏负责设计研究方案、研究实施及论文撰写;程品晶负责论文总体修改和最终审校;刘小敏、刘霖霏参与论文写作及修改;伍海彪、王柏琦、成浩负责收集患者影像和临床数据;杨骏辉参与数据分析工作
| [1] |
Chen P, Liu Y, Wen Y, et al. Non-small cell lung cancer in China[J]. Cancer Commun (Lond), 2022, 42(10): 937-970. DOI:10.1002/cac2.12359 |
| [2] |
Zeng H, Chen W, Zheng R, et al. Changing cancer survival in China during 2003-15: a pooled analysis of 17 population-based cancer registries[J]. Lancet Glob Health, 2018, 6(5): e555-e567. DOI:10.1016/S2214-109X(18)30127-X |
| [3] |
Arrieta O, Ramírez-Tirado LA, Caballé-Perez E, et al. Response rate of patients with baseline brain metastases from recently diagnosed non-small cell lung cancer receiving radiotherapy according to EGFR, ALK and KRAS mutation status[J]. Thorac Cancer, 2020, 11(4): 1026-1037. DOI:10.1111/1759-7714.13359 |
| [4] |
Li D, Ding L, Ran W, et al. Status of 10 targeted genes of non-small cell lung cancer in eastern China: a study of 884 patients based on NGS in a single institution[J]. Thorac Cancer, 2020, 11(9): 2580-2589. DOI:10.1111/1759-7714.13577 |
| [5] |
李淑华, 杨昭, 王小雷, 等. 基于CT影像组学列线图预测肺腺癌EGFR突变的研究[J]. 临床放射学杂志, 2022, 41(9): 1676-1682. Li SH, Yang Z, Wang XL, et al. CT-based radiomics nomogram to predict EGFR mutation in lung adenocarcinoma[J]. J Clin Radiol, 2022, 41(9): 1676-1682. DOI:10.13437/j.cnki.jcr.2022.09.037 |
| [6] |
Liu SY, Zhang JT, Zeng KH, et al. Perioperative targeted therapy for oncogene-driven NSCLC[J]. Lung Cancer, 2022, 172: 160-169. DOI:10.1016/j.lungcan.2022.05.007 |
| [7] |
de Sousa VML, Carvalho L. Heterogeneity in lung cancer[J]. Pathobiology, 2018, 85(1): 96-107. DOI:10.1159/000487440 |
| [8] |
Lambin P, Leijenaar R, Deist TM, et al. Radiomics: the bridge between medical imaging and personalized medicine[J]. Nat Rev Clin Oncol, 2017, 14(12): 749-762. DOI:10.1038/nrclinonc.2017.141 |
| [9] |
Gillies RJ, Kinahan PE, Hricak H. radiomics: images are more than pictures, they are data[J]. Radiology, 2016, 278(2): 563-577. DOI:10.1148/radiol.2015151169 |
| [10] |
Qiu Q, Duan J, Yin Y. Radiomics in radiotherapy: applications and future challenges[J]. Prec Rad Oncol, 2020, 4(1): 29-33. DOI:10.1002/pro6.1087 |
| [11] |
Shen C, Liu Z, Wang Z, et al. Building CT radiomics based nomogram for preoperative esophageal cancer patients lymph node metastasis prediction[J]. Transl Oncol, 2018, 11(3): 815-824. DOI:10.1016/j.tranon.2018.04.005 |
| [12] |
Castellanos E, Feld E, Horn L. Driven by mutations: the predictive value of mutation subtype in EGFR-mutated non-small cell lung cancer[J]. J Thorac Oncol, 2017, 12(4): 612-623. DOI:10.1016/j.jtho.2016.12.014 |
| [13] |
Rossi G, Barabino E, Fedeli A, et al. Radiomics detection of EGFR mutations in NSCLC[J]. Cancer Res, 2021, 81(3): 724-731. DOI:10.1158/0008-5472.Can-20-0999 |
| [14] |
郭天慧, 王浩铭, 任瑞美, 等. 影像组学与影像基因组学在肺癌中的研究进展[J]. 国际肿瘤学杂志, 2018, 45(9): 566-569. Guo TH, Wang HM, Ren RM, et al. Progress of radiomics and radiogenomics in lung cancer[J]. Int Oncol, 2018, 45(9): 566-569. DOI:10.3760/cma.j.issn.1673-422X.2018.09.012 |
| [15] |
刘昊沅, 艾和平, 赵俊苹, 等. 影像组学在EGFR突变型非小细胞肺癌的研究[J]. 放射学实践, 2022, 37(10): 1318-1321. Liu HY, Ai HP, Zhao JP, et al. Research on radiomics of EGFR mutations in non-small cell lung cancer[J]. Radiol Prac, 2022, 37(10): 1318-1321. DOI:10.13609/j.cnki.1000-0313.2022.10.023 |
| [16] |
Chen W, Hua Y, Mao D, et al. A computed tomography-derived radiomics approach for predicting uncommon EGFR mutation in patients with nsclc[J]. Front Oncol, 2021, 11: 722106. DOI:10.3389/fonc.2021.722106 |
| [17] |
唐兴, 白国艳, 王虹, 等. 基于多序列MRI影像组学预测肺腺癌EGFR基因表型[J]. 放射学实践, 2021, 36(8): 1010-1015. Tang X, Bai GY, Wang H, et al. Predictive value for EGFR gene phenotype of lung adenocarcinoma based on multi-sequences MRI radiomics[J]. Radiol Pract, 2021, 36(8): 1010-1015. DOI:10.13609/j.cnki.1000-0313.2021.08.012 |
| [18] |
Tu W, Sun G, Fan L, et al. Radiomics signature: a potential and incremental predictor for EGFR mutation status in NSCLC patients, comparison with CT morphology[J]. Lung Cancer, 2019, 132: 28-35. DOI:10.1016/j.lungcan.2019.03.025 |
| [19] |
Yang X, Liu M, Ren Y, et al. Using contrast-enhanced CT and non-contrast-enhanced CT to predict EGFR mutation status in NSCLC patients—a radiomics nomogram analysis[J]. Eur Radiol, 2022, 32(4): 2693-2703. DOI:10.1007/s00330-021-08366-y |
| [20] |
Zhang J, Zhao X, Zhao Y, et al. Value of pre-therapy (18)F-FDG PET/CT radiomics in predicting EGFR mutation status in patients with non-small cell lung cancer[J]. Eur J Nucl Med Mol Imaging, 2020, 47(5): 1137-1146. DOI:10.1007/s00259-019-04592-1 |
| [21] |
Jia TY, Xiong JF, Li XY, et al. Identifying EGFR mutations in lung adenocarcinoma by noninvasive imaging using radiomics features and random forest modeling[J]. Eur Radiol, 2019, 29(9): 4742-4750. DOI:10.1007/s00330-019-06024-y |
| [22] |
王子阳, 尹国涛, 李小凤, 等. 机器学习结合18F-FDG PET/CT影像组学特征对肺腺癌EGFR突变亚型的预测价值[J]. 中华核医学与分子影像杂志, 2021, 41(8): 479-485. Wang ZY, Yin GT, Li XF, et al. Value of machine learning and 18F-FDG PET/CT radiomics features in lung adenocarcinoma EGFR mutation subtypes prediction[J]. Chin J Nucl Med Mol Imaging, 2021, 41(8): 479-485. DOI:10.3760/cma.j.cn321828-20201105-00401 |
| [23] |
Huang L, Cao Y, Zhou F, et al. Lung adenocarcinoma: development of nomograms based on PET/CT images for prediction of epidermal growth factor receptor mutation status and subtypes[J]. Nucl Med Commun, 2022, 43(3): 310-322. DOI:10.1097/mnm.0000000000001519 |
| [24] |
Wang C, Ma J, Shao J, et al. Predicting EGFR and PD-L1 status in NSCLC patients using multitask AI system based on CT images[J]. Front Immunol, 2022, 13: 813072. DOI:10.3389/fimmu.2022.813072 |
| [25] |
Li S, Ding C, Zhang H, et al. Radiomics for the prediction of EGFR mutation subtypes in non-small cell lung cancer[J]. Med Phys, 2019, 46(10): 4545-4552. DOI:10.1002/mp.13747 |
| [26] |
Zhang G, Deng L, Zhang J, et al. Development of a nomogram based on 3D CT radiomics signature to predict the mutation status of EGFR molecular subtypes in lung adenocarcinoma: a multicenter study[J]. Front Oncol, 2022, 12: 889293. DOI:10.3389/fonc.2022.889293 |
 2023, Vol. 43
2023, Vol. 43


