2. 山东第一医科大学第二附属医院肿瘤科,泰安 271000;
3. 深圳市联影高端医疗装备创新研究院,深圳 518045;
4. 北京联影智能影像技术研究院,北京 100094;
5. 上海联影医疗科技股份有限公司,上海 201807
2. Department of Oncology, Second Affiliated Hospital of Shandong First Medical University, Taian 271000, China;
3. Shenzhen United Imaging Research Institute of Innovative Medical Equipment, Shenzhen 518045, China;
4. Beijing United Imaging Research Institute of Intelligent Imaging, Beijing 100094, China;
5. Shanghai United Imaging Healthcare Co., Ltd., Shanghai 201807, China
脑胶质母细胞瘤(GBM)是中枢神经系统最常见的原发恶性肿瘤[1],而放射治疗是其标准治疗手段之一[2-3]。放射治疗技术的发展,包括常规放疗、立体定向放疗和影像引导技术等,使得肿瘤放疗更加精准[4],从而对肿瘤靶区的勾画质量和效率提出了新的要求。临床上,医师参考多模态核磁共振(MR)影像,在CT图像上完成靶区的勾画。精准的勾画是改进放疗计划、提升疗效并降低不良反应的关键因素之一。然而,临床实践表明,GBM靶区在CT和MR图像上均存在灰度差异小、边界模糊等问题,尽管参考多模态MR图像,对靶区范围的手工精准勾画,依然费时费力,且一致性差。因此,应用基于人工智能的靶区自动分割,将能有效减少人为因素造成的误差,提升临床医生的工作效率[5-8]。Tang等[9]用基于深度卷积神经网络的深度特征融合模型,并联合使用CT影像和多模态MR影像,使得GBM靶区自动分割的相似性系数(DSC)达到了0.836,达到了临床可接受的水平[9]。另一种基于模糊特征融合的自动分割方法,即对每个模态的图像进行模糊特征学习,并进行特征融合,将GBM靶区分割的DSC提升到了0.88[10]。结合多模态影像,利用深度学习网络模型对其特征进行挖掘和提取,将进一部提升GBM的诊疗水平。本研究通过改进现有的3D U-Net,使用多模态图像来分别训练该模型,并预测GBM的靶区(GTV、CTV1和CTV2),探索了多模态影像数据集对GBM放疗靶区自动勾画效果的影响。
资料与方法1. 数据的采集与标注:回顾性收集北京大学第三医院肿瘤放疗科30例经组织学证实为高级别胶质母细胞瘤(HGG)的病例,其中男16例,女14例,中位年龄54岁,每个病例包含1例非增强CT序列和对应两例MRI序列(对比增强T1加权序列,T1C;T2液体衰减反转恢复序列,T2-FLAIR)。每例CT序列包含GBM的原发肿瘤靶区(GTV)及其相应的临床靶区(CTV1,CTV2)的勾画。所有病例的影像数据的属性见表 1。
|
|
表 1 高级胶质母细胞瘤(HGG) 病例不同模态的数据集属性 Table 1 Properties of datasets of different models high grade glioblastoma |
所有的GBM病例的勾画(GTV,CVT1和CVT2)是通过将MRI与对应的CT刚性配准后,由两位经验丰富的专科医生根据美国肿瘤放射治疗协会(RTOG)的两步法勾画原则,参考MRI图像上靶区形态与特征,在CT上手动勾画完成,然后两位医生交叉修改对方的手动勾画,最后由主任医师完成手动勾画的评估,并在提出改进意见后进一步修改(图 1)。

|
图 1 不同模态数据CT(A)、T1C(B)和T-FLAIR(C) 上GTV(红色)、CTV1(绿色)、CTV2(蓝色)的手动勾画 Figure 1 Manually delineated GTV (red), CTV1 (green) and CTV2 (blue) on CT (A), T1C (B), and T2-FLAIR (C) |
2. 数据的预处理与增强:①配准:所有病例的T1C序列和T2-FLAIR序列均与对应的CT序列进行刚性配准。②去颅骨:用现有基于深度学习的全脑组织自动分割模型对所有的CT和配准后的MRI(T1C和T2-FLAIR)图像做去颅骨操作[11],获得去颅骨后的CT和MRI图像。③裁剪:对原始CT和MRI图像的背景进行裁剪,从而达到减小模型输入数据的尺寸,进而减少模型的计算负载。④归一化:通过三阶样条插值和邻近插值,分别将数据集中所有CT和MRI图像及其对应的人工勾画(GTV、CTV1和CTV2)的体素间距插值到中间体素间距,并且对所有CT和MRI图像的信号值进行Z-score归一化[12-13],从而使分割网络能够更好地学习空间语义信息。
3. 网络结构:U-Net是一种流行的编码器-解码器(Encoder-Decoder)网络[14],已广泛应用于图像分割领域。由于3D CNN在聚合3D医学图像中上下文的有价值信息方面表现出很高的有效性[15-16],因此,采用深度3D CNN网络来构建分割模型。本研究中的分割网络是基于一个5层编码器-解码器的3D U-Net。在每个编码器中,设计了两组卷积核为3×3×3的卷积层(convolutional layer)来提取输入图像(input volume)的特征(input map),每个卷积层后面采用dropout为0.5的实例归一化(instance normalization)和LeakyReLU(负斜率1e-2)激活函数,分别取代了目前深度学习模型中更常见的Batch归一化(batch normalization)和ReLU激活函数,最后使用2×2×2最大池(max-pooling)对每个编码器中的特征图进行下采样(down-sample);在每一个解码器的最后,使用2×2×2反卷积核(deconvolutional kernel)对每个解码器中的特征图进行上采样(up-sample)。整个模型中的每个编码器的输出特征图(output map)通过跳跃连接与相应解码器的输入特征图(input map)连接(图 2)。
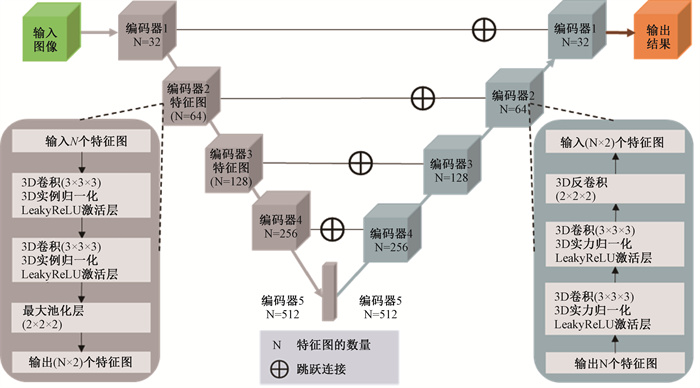
|
图 2 3D U-Net模型结构 Figure 2 Architecture of the 3D U-Net model |
4. 训练目标:由于可用的GPU内存有限,采用从原始图像中滑动裁剪较小的图像块(image patch)作为分割网络的输入。虽然这种基于图像块的训练方法限制了模型的视野,无法从原始图像中收集到足够的上下文信息,但对小目标分割的影响是微乎其微的。
分割网络的损失函数(objective function) L是Dice loss L_dice和交叉熵损失(cross-entropy loss) L_CE的加权和[17]:
| $ L=\alpha_1 L_{dice}+\beta_1 L_{C E} $ | (1) |
权重α1和β1分别设置为0.4和0.6。L_dice和L_CE分别定义为:
| $ L_{dice}=-\frac{2}{C} \sum\limits_{k \in C} \frac{\sum_{i \in N} G_i^k P_i^k}{\sum_{i \in N} G_i^k+\sum_{i \in N} P_i^k} $ | (2) |
| $ L_{C E}=1-\frac{1}{N} \sum\limits_{i \in N} \sum\limits_{k \in C} G_i^k \log P_i^k $ | (3) |
式中,C为被分割的目标类别数量;N为训练时每个patch中的体素数量;Gik和Pik分别为人工勾画和模型预测的勾画中第k个类别的第i个体素。
5. 实验实施:本研究中,整个数据集包含30组CT和配准后的MRI(T1C和T2-FLAIR)数据对,被划分为两组:25例数据对作为训练集(包括5例数据对作为验证集),用于训练分割网络和优化网络的超参数;以及5例数据对作为测试集,用于评估训练后网络的性能。从原始数据对中裁剪出32×256×256的图像块作为修改后的3D U-Net的输入图像,网络输入的batch size为2,优化器为RMSprop,初始学习率为0.001,梯度下降策略为随机梯度下降(SGD,动量为0.9),最大训练轮次(epochs)为150。对每个patch中每个类的分割结果进行高斯融合,得到全分辨率分割结果,并通过最大连通分域对每个类的全分辨率分割结果进行后处理。为了探究本研究提出的多模态分割方法对GTV、CVT1和CVT2自动分割的有效性,本研究设计了4组不同的数据集及其组合用于训练和测试同一分割网络:CT,仅用单模态CT图像训练和测试本研究中的分割网络;CT-T1C,使用双模态CT和MR T1C图像训练和测试本研究中的分割网络;CT-T2-FLAIR,使用双模态CT和MR T2-FLAIR图像训练和测试本研究中的分割网络;CT-MRIs,使用三种模态CT、T1C和T2-FLAI图像训练和测试本文中的分割网络。
6. 模型评估:分别计算了4组数据集(CT,CT-T1C、CT-T2-FLAIR、CT-MRIs)中模型自动分割的靶区(GTV、CTV1和CTV2)与人工勾画的靶区之间的Dice相似系数(Dice similar coefficient,DSC)、95% Hausdorff距离(95% Hausdorff distance,HD95)和相对体积误差(relative volume error, RVE),这些指标作为定量评估模型分割的准确性。
7. 统计学处理:采用SPSS 21.0软件进行分析。计算测试集上测试样本的DSC,HD95用于评估自动勾画轮廓与手动勾画轮廓之间的一致性;采用配对t 检验比较不同多模态方法与仅CT模态方法的DSC、HD95。P < 0.05为差异有统计学意义。
结果1. 自动勾画性能评估:表 2为GTV、CTV1和CTV2在4组数据集中相同测试集上的测试结果的统计分析。根据表 2可以看出,对于GTV的自动勾画,相较于数据集CT上的DSC(0.79±0.04)和HD95(12.33±5.32)mm,模型在数据集CT-T1C上的DSC(0.93±0.01)和HD95(2.16±0.35)mm有非常显著的提升,且两者的差异均有统计学意义(DSC: t=3.64, P < 0.05;HD95:t=2.85, P < 0.05);在数据集CT-T2-FLAIR上的DSC(0.85±0.03)和HD95(7.46±1.35) mm的提升并不明显,且两者的差异均无统计学意义(P>0.05);模型在数据集CT-MRIs上的DSC (0.94±0.01)和HD95(2.09±0.26)在4种数据集中取得最好的分割结果,且两者的差异均有统计学意义(t=3.78、4.07, P < 0.05)。
|
|
表 2 不同数据集上GTV、CTV1和CTV2的测试结果(x±s) Table 2 Test results of GTV, CTV1 and CTV2 on different datasets(x±s) |
对于CTV1的自动勾画,相较于数据集CT上的DSC(0.67±0.08)和HD95(22.80±9.66),模型在数据集CT-T1C上的DSC(0.73±0.07)和HD95(21.24±10.58)mm的提升并不明显,且两者的差异均无统计学意义(P>0.05);模型在数据集CT-T2-FLAIR上的DSC(0.90±0.04)和HD95(2.88±1.13)mm有非常显著的提升,其中DSC的差异有统计学意义(t=2.50, P < 0.05),但是HD95的差异无统计学意义(P>0.05),并且在4种数据集中取得最好的分割结果;模型在数据集CT-MRIs上的DSC(0.90±0.04)和HD95(3.78±1.06)mm有明显的提升,但是两者的差异均无统计学意义(P>0.05)。
对于CTV2的自动勾画,相较于数据集CT上的DSC(0.73±0.07)和HD95(17.67±7.80),模型在数据集CT-T1C上的DSC(0.76±0.08)和HD95(14.03±6.51)的提升并不明显,且两者的差异均无统计学意义(P>0.05);模型在数据集CT-T2-FLAIR上的DSC(0.90±0.04)和HD95(3.70±1.10) mm有非常显著的提升,其中DSC的差异有统计学意义(t=2.67, P < 0.05),但是HD95的差异无统计学意义(P>0.05);模型在方法CT-MRIs上的DSC(0.91±0.04)和HD95(2.40±1.16) mm在4种数据集中取得最好的分割结果,但是两者的差异均无统计学意义(P>0.05)。
2. 自动勾画靶区相对体积误差的评估:为了进一步评估自动分割的效果,计算相同测试集上4种基于不同数据集的模型分割出的GTV、CTV1和CTV2与人工勾画的相对体积误差(RVE)。如图 3所示,对于GTV,数据集CT-MRIs取得最小的平均RVE,1.16%;对于CTV1,数据集CT-T2-FLAIR取得最小的平均RVE,3.25%,略优于数据集CT-MRIs的平均RVE,3.61%;对于CTV2,数据集CT-T2-FLAIR取得最小的平均RVE,5.18%,略优于数据集CT-MRIs的平均RVE,5.35%。
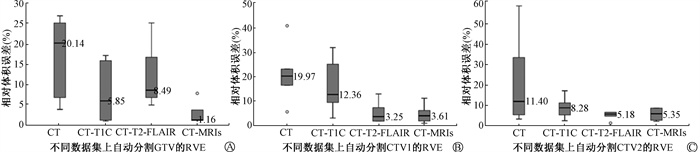
|
注:GTV. 胶质瘤原发肿瘤; CTV1. 胶质瘤临床靶区1; CTV2. 胶质瘤临床靶区2; RVE. 相对体积误差 图 3 基于不同数据集自动分割的GTV(A)、CTV1(B)和CTV2(C)与人工勾画的RVE Figure 3 The RVEs of GTV (A), CTV1 (B), and CTV2 (C) between automatic segmentation and manual delineation |
3. 自动勾画结果:数据集CT-MRIs自动勾画与人工勾画的结果(GTV、CTV1和CTV2)的对比。对于GTV,自动勾画主要在上下界会分别多勾画一层;对于CTV1,自动勾画与人工勾画一致性非常好;对比CTV2,自动勾画部分层面没有有效地避开脑干和眼球等器官。
讨论脑胶质母细胞瘤靶区勾画本质上是一种图像分割,是基于CT、MRI等影像并按照肿瘤放疗靶区勾画要求进行的[12-17]。手动分割过程耗时长,分割的准确性取决于医生的经验。而图像自动分割能显著提高分割的准确性和一致性,并提高医生的工作效率[18]。
近年来,深度学习方法在肿瘤放疗中得到了广泛的应用[19]。本研究改进了基于深度学习的3D U-Net,在每个编码器中,每个卷积层后面采用dropout为0.5的实例归一化和LeakyReLU激活函数,分别替代目前深度学习模型中较常见的Batch归一化和ReLU激活函数,提高模型的鲁棒性;并且使用多模态图像自动分割脑胶质母细胞瘤的GTV、CTV1和CTV2,提高模型的分割精度。结果表明,使用多模态图像对脑胶质母细胞瘤的分割性能明显优于仅使用单模态图像CT。改进后的3D-Unet在多模态影像数据集CT-MRIs上获得最好的GTV自动分割结果(平均DSC:0.94,平均HD95:2.09 mm,平均RVE:1.16%),并且与在单模态影像数据集CT的自动分割结果(平均DSC:0.79,平均HD95:12.33 mm,平均RVE:20.14%)相比,并且DSC和HD95的差异均有统计学意义,但是对于GTV的上下界的自动勾画会存在多勾画一层的情况。该模型在多模态影像数据集CT-MRIs的CTV1和CTV2自动分割结果(平均DSC: 0.90 vs. 0.91, 平均HD95: 3.78 mm vs. 2.41 mm, 平均RVE: 3.61% vs. 5.35%)也均有较好的一致性,但是与单模态影像数据集CT数据集的自动分割结果(平均DSC: 0.67 vs. 0.73, 平均HD95: 22.80 mm vs. 17.67mm, 平均RVE: 19.97% vs. 11.40%)相比,两个靶区的DSC和HD95的差异均无统计学意义;对于CTV1,自动勾画与人工勾画一致性非常好,对比CTV2,自动勾画部分层面没有有效“避开”脑干和眼球等器官。结果表明,基于深度学习方法在多模态医学图像上自动分割脑胶质母细胞瘤肿瘤靶区对放射治疗工作流程中缩短肿瘤勾画时间非常有潜力。
数据集的大小是决定深度学习水平的重要参数之一[20]。如果数据集很小,网络的参数数量很大,可能会导致过拟合。当前实验中使用了较少的测试样本,对于小测试样本中取得较好的自动分割结果仍需进一步验证,但是仍有理由相信将来提高训练和测试样本量,这种基于深度学习的多模态图像自动分割方法仍然有进一步提高分割效果的可能。此外,脑内有些脑组织的形态特征,如脑干,与T2-FLAIR序列中脑胶质瘤的临床靶区(CTV2)非常相似,导致少数脑组织通常被网络分割为临床靶区(CTV2),这对于脑胶质母细胞瘤的靶区自动分割来说是一项具有挑战性的任务。另外,不同厂商的设备或不同的扫描协议常常导致医学影像有不同的体素间距和分辨率,以及成像质量和风格,这些差异对于MRI来说尤为明显。AI算法对于多种设备和多种扫描协议的多“中心”医学影像数据的泛化性,仍然是一个重要的挑战[8]。在将来的工作中,会收集更多不同“中心”的数据来优化和测试改自动分割方法,进一步提高其自动分割的鲁棒性性和泛化性。
靶区和危及器官勾画准确性是放射治疗疗效的保证。人工无法保证勾画的准确性和一致性,其次是效率低[21]。随着基于深度学习神经网络的人工智能技术迁移到医疗领域,在影像自动分割研究领域的迅猛发展,能够改善分割效果,提高临床医生工作效率,并提高所勾画的靶区轮廓的一致性[22],最大限度地降低不同勾画者间产生的差异性,在临床应用时只需临床医师做出较小的修改[23]。然而,仅仅基于影像学的靶区自动勾画,必然存在其局限性。在未来该领域的人工智能研究中,应该纳入患者详细的临床病史、临床检查特征及既往治疗情况等,在完整数据的支撑下,获得更为精准的个性化自动勾画结果。
利益冲突 全体作者未因进行该研究而接受任何不当的财务或财务利益,在此对研究的独立性和科学性予以保证
作者贡献声明 田素青负责研究设计、数据收集及起草论文;许昕、姜玉良审核数据;刘应龙、戴卓捷、章卫、贾乐成负责数据整理和计算;王俊杰审核指导论文写作
| [1] |
Gritsch S, Batchelor TT, Gonzalez Castro LN. Diagnostic, therapeutic, and prognostic implications of the 2021 World Health Organization classification of tumors of the central nervous system[J]. Cancer, 2022, 128(1): 47-58. DOI:10.1002/cncr.33918 |
| [2] |
Souhami L, Seiferheld W, Brachman D, et al. Randomized comparison of stereotactic radiosurgery followed by conventional radiotherapy with carmustine to conventional radiotherapy with carmustine for patients with glioblastoma multiforme: report of Radiation Therapy Oncology Group 93-05 protocol[J]. Int J Radiat Oncol Biol Phys, 2004, 60(3): 853-860. DOI:10.1016/j.ijrobp.2004.04.011 |
| [3] |
Corso CD, Bindra RS, Mehta MP. The role of radiation in treating glioblastoma: here to stay[J]. J Neurooncol, 2017, 134(3): 479-485. DOI:10.1007/s11060-016-2348-x |
| [4] |
Narayana A, Yamada J, Berry S, et al. Intensity-modulated radiotherapy in high-grade gliomas: clinical and dosimetric results[J]. Int J Radiat Oncol Biol Phys, 2006, 64(3): 892-897. DOI:10.1016/j.ijrobp.2005.05.067 |
| [5] |
Pereira S, Pinto A, Alves V, et al. Brain tumor segmentation using convolutional neural networks in MRI images[J]. IEEE Trans Med Imaging, 2016, 35(5): 1240-1251. DOI:10.1109/TMI.2016.2538465 |
| [6] |
Kamnitsas K, Ledig C, Newcombe V, et al. Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation[J]. Med Image Anal, 2017, 36: 61-78. DOI:10.1016/j.media.2016.10.004 |
| [7] |
Ullah F, Ansari SU, Hanif M, et al. Brain MR image enhancement for tumor segmentation using 3D U-Net[J]. Sensors (Basel), 2021, 21(22): 7528. DOI:10.3390/s21227528 |
| [8] |
张瑞萍, 刘应龙, 张文静, 等. 基于人工智能的多模态影像辅助海马体自动勾画研究[J]. 中国医学物理学杂志, 2022, 39(3): 390-396. Zhang RP, Liu YL, Zhang WJ, et al. Auto-segmentation of the hippocampus in multimodal image using artificial intelligence[J]. Chin J Med Phys, 2022, 39(3): 390-396. DOI:10.3969/j.issn.1005-202X.2022.03.021 |
| [9] |
Tang F, Liang S, Zhong T, et al. Postoperative glioma segmentation in CT image using deep feature fusion model guided by multi-sequence MRIs[J]. Eur Radiol, 2020, 30(2): 823-832. DOI:10.1007/s00330-019-06441-z |
| [10] |
Guo L, Wang P, Sun R, et al. A fuzzy feature fusion method for auto-segmentation of gliomas with multi-modality diffusion and perfusion magnetic resonance images in radiotherapy[J]. Sci Rep, 2018, 8(1): 3231. DOI:10.1038/s41598-018-21678-2 |
| [11] |
Nalepa J, Marcinkiewicz M, Kawulok M. Data augmentation for brain-tumor segmentation: a review[J]. Front Comput Neurosci, 2019, 13: 83. DOI:10.3389/fncom.2019.00083 |
| [12] |
Isensee F, Jaeger PF, Kohl S, et al. nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation[J]. Nat Methods, 2021, 18(2): 203-211. DOI:10.1038/s41592-020-01008-z |
| [13] |
Ghaffari M, Sowmya A, Oliver R. Automated brain tumor segmentation using multimodal brain scans: a survey based on models submitted to the BraTS 2012-2018 Challenges[J]. IEEE Rev Biomed Eng, 2020, 13: 156-168. DOI:10.1109/RBME.2019.2946868 |
| [14] |
Valanarasu J, Sindagi VA, Hacihaliloglu I, et al. KiU-Net: Overcomplete convolutional architectures for biomedical image and volumetric segmentation[J]. IEEE Trans Med Imaging, 2022, 41(4): 965-976. DOI:10.1109/TMI.2021.3130469 |
| [15] |
Shelhamer E, Long J, Darrell T. Fully convolutional networks for semantic segmentation[J]. IEEE Trans Pattern Anal Mach Intell, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [16] |
Bitarafan A, Nikdan M, Baghshah MS. 3D Image segmentation with sparse annotation by self-training and internal registration[J]. IEEE J Biomed Health Inform, 2021, 25(7): 2665-2672. DOI:10.1109/JBHI.2020.3038847 |
| [17] |
Chen L, Bentley P, Mori K, et al. DRINet for medical image segmentation[J]. IEEE Trans Med Imaging, 2018, 37(11): 2453-2462. DOI:10.1109/TMI.2018.2835303 |
| [18] |
Menze BH, Jakab A, Bauer S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE Trans Med Imaging, 2015, 34(10): 1993-2024. DOI:10.1109/TMI.2014.2377694 |
| [19] |
Cardenas CE, McCarroll RE, Court LE, et al. Deep learning algorithm for auto-delineation of high-risk oropharyngeal clinical target volumes with built-in dice similarity coefficient parameter optimization function[J]. Int J Radiat Oncol Biol Phys, 2018, 101(2): 468-478. DOI:10.1016/j.ijrobp.2018.01.114 |
| [20] |
Vu MH, Grimbergen G, Nyholm T, et al. Evaluation of multislice inputs to convolutional neural networks for medical image segmentation[J]. Med Phys, 2020, 47(12): 6216-6231. DOI:10.1002/mp.14391 |
| [21] |
慕光睿, 杨燕平, 高耀宗, 等. 基于多尺度三维卷积神经网络的头颈部危及器官分割方法[J]. 南方医科大学学报, 2020, 40(4): 491-498. DOI:10.12122/j.issn.1673-4254.2020.04.07 |
| [22] |
Tian S, Wang C, Zhang R, et al. Transfer learning-based autosegmentation of primary tumor volumes of glioblastomas using preoperative MRI for radiotherapy treatment[J]. Front Oncol, 2022, 12: 856346. DOI:10.3389/fonc.2022.856346 |
| [23] |
葛迦, 宁丽华, 严森祥, 等. 两种软件Smart Segmentation与MIM Atlas自动勾画鼻咽癌危及器官的准确性研究[J]. 中华放射医学与防护杂志, 2019, 39(9): 668-672. Ge J, Ning LH, Yan SX, et al. Automatic segmentation of organs at risk for nasopharyngeal carcinoma with smart segmentation and MIM Atlas[J]. Chin J Radiol Med Prot, 2019, 39(9): 668-672. DOI:10.3760/cma.j.issn.0254-5098.2019.09.006 |
 2022, Vol. 42
2022, Vol. 42


