2. 中国科学院合肥物质科学研究院强磁场中心,合肥 230031
2. Key Laboratory of High Magnetic Field and Ion Beam Physical Biology, Hefei Institutes of Physical Science, Chinese Academy of Science, Hefei 230031, China
放疗和化疗是目前临床癌症治疗的主要手段[1]。辐射和化疗药物能通过诱导癌细胞的DNA双链断裂(double-strand breakage,DSB)达到抑制癌细胞存活的目的。DSB修复的主要途径是非同源末端连接(non-homologous end joining,NHEJ)和同源重组(homologous recombination,HR)[2]。针对DNA损伤修复因子的特异性靶向药物已应用于临床[3],药物通过干预这些修复因子的生物学功能,使肿瘤细胞丧失DNA损伤修复能力,进而增加肿瘤放化疗的敏感性[4-5]。
锌指蛋白451(ZNF451)是一种SUMO E3连接酶,它有两个N端SUMO2相对作用基序(SIMs),1个PilT N端“PIN”域,1个泛素交互基序(UIM),11个锌指区域[6]。之前的研究表明,ZNF451可以参与DNA-蛋白质交联、转化生长因子-β信号、PML小体和上皮细胞-间充质转化等多种生物学过程[7-11]。
目前对于ZNF451的研究未涉及DNA损伤修复且其在DNA损伤修复中的作用亟待阐明。本研究以ZNF451为关键靶点,探讨其对A549细胞和HeLa细胞DNA损伤修复的影响及相关的分子生物学机制,以期为癌症的诊断和治疗提供新的调控因子。
材料与方法1. 细胞培养和辐射处理:A549和HeLa细胞系购自中国科学院细胞库,并在补充有10%胎牛血清(以色列Biological公司)和1% (V/V)青霉素/链霉素(上海Beyotime公司)的DMEM(美国HyClone公司)中培养。细胞由德国贝欧宝BioBeam GM2000 γ射线辐照仪单次照射,吸收剂量率为3.27 Gy/min,总剂量6 Gy。辐照后放回培养箱继续培养。
2. 化学品和试剂:CCK-8试剂盒(合肥Biosharp公司),依托泊苷(上海TargetMol公司),Lipofectamine 2000(美国Thermo Fishier公司),蛋白A/G珠(美国Santa公司),ZNF451(美国Sigma公司),MDC1(英国Absin公司),γ-H2AX(上海Novus公司),53BP1(美国CST公司),β-肌动蛋白(美国Affinity公司)。siRNA序列为:5′ GCUCAACUGUAAGAUUUAUTTAUAAAUCUUACAGUUGAGCTT 3′。
3. 慢病毒包装shRNA细胞及细胞计数:使用pLKO.1-shRNA、psPAX2和pMD2.G的三质粒系统在HEK293 T细胞中构建慢病毒颗粒,将稳转细胞以80 000个/皿的初始密度接种在60 mm培养皿中。每24 h重悬细胞后计数。记录6 d内细胞生长情况并绘制生长曲线。细胞分为对照组和敲低组。
4. CCK-8法检测细胞活力:接种细胞悬液于96孔板中,每孔约5 000个细胞,用10~100 μmol/L的依托泊苷处理细胞。连续暴露48 h后,每孔加入10 μl CCK-8溶液,孵育4 h。使用酶标仪测定450 nm处的吸光度( A )值。
5. 蛋白质印迹分析:细胞裂解液于4℃、12 000×g离心15 min,上清液用BCA试剂盒(上海Sangon Biotech公司)检测 A 值。20~40 μg总蛋白样品通过8%~12%十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)分离并转移至聚偏二氟乙烯膜(PVDF)膜上,膜在室温下用0.1% Tween 20 (TBST)和5%牛血清白蛋白(BSA)封闭。4℃下孵育一抗,过夜后室温下孵育二抗1 h。化学发光底物(武汉BOSTER公司)显示蛋白条带。
6. 免疫沉淀分析:总细胞裂解液以12 000×g离心15 min。取上清,加入一抗4℃孵育12 h后,加入20 μlG蛋白修饰琼脂糖珠(protein G sepharose bead),捕获蛋白复合物。4℃温和翻转孵育3 h,离心半径13.5 cm,2 500 r/min,离心5 min。用预冷的磷酸盐缓冲液(PBS)洗涤复合颗粒,将免疫沉淀物重新悬浮在SDS-PAGE上样缓冲液中用于蛋白质印迹分析。
7. 免疫荧光染色:预冷至-20℃的甲醇固定细胞20 min。用PBS洗涤细胞,0.5% Triton X-100渗透细胞,含有5% BSA和0.5% Triton X-100的PBS室温封闭1 h。4℃孵育一抗,过夜后用荧光标记的二抗室温孵育细胞1 h。细胞核用DAPI复染。使用荧光显微镜捕获图像。细胞分为对照组、辐照处理组、依托泊苷处理组。
8. DR-GFP质粒及HR和NHEJ检测:基于I-SceI内切酶的DNA损伤报告系统由军事科学院王治东课题组馈赠。该质粒系统包含DR-GFP质粒、EJ-GFP质粒及I-SceI内切酶。对于NHEJ测定,NHEJ-GFP在20 μl Hind Ш酶体系(包括cutmart和ddH2O缓冲液)中孵育3 h。用Lipofectamine 2000将混合物转染到细胞中。对于HR测定,将DR-GFP在基础培养基中孵育并用Lipofectamine 2000转染到细胞悬浮液中,24 h后将I-SceI内切酶转染到细胞中。质粒转染6 h后,细胞均用完全培养基代替基础培养基培养至48 h。使用流式细胞仪对阳性细胞进行测定。
9. 统计学处理:所有实验至少独立进行3次,计量资料符合正态分布,以x±s表示,采用GraphPad Prism 9软件进行分析。Student′s t检验用于两组之间的比较,ANOVA用于多组之间的比较。P < 0.05为差异有统计学意义。
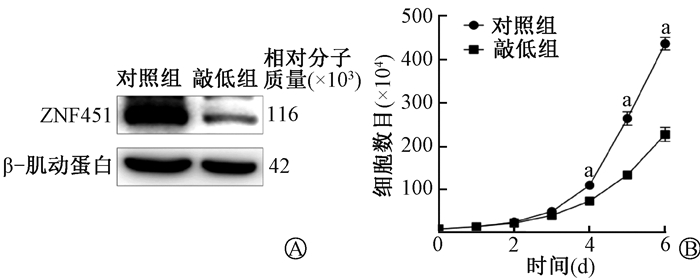
结果1. 化疗药物诱导ZNF451的表达:检测不同浓度的依托泊苷处理A549和HeLa细胞后不同时间ZNF451蛋白表达的变化,结果如表 1和表 2所示,依托泊苷抑制了ZNF451的表达,且呈时间及剂量依赖性(P < 0.01)。如图 1所示,利用慢病毒包装技术构建稳定敲低ZNF451的HeLa细胞系,该细胞中ZNF451蛋白表达水平显著降低,敲低组ZNF451的蛋白表达量相对于对照组降>低至20%±3%(t = 27.93,P < 0.01);ZNF451敲低组在接种第4~6天,生长速率较对照组明显变缓(t = 5.95、21.27、34.16,P < 0.01)。这些结果表明,ZNF451对临床化疗药物敏感,抑制ZNF451的表达阻滞了HeLa细胞的生长速率。
|
|
表 1 不同浓度依托泊苷处理细胞24 h后ZNF451的相对表达(x±s) Table 1 ZNF451 expression level in A549 and Hela cells after the treatment of etoposide with different concentrations for 24 h (x±s) |
|
|
表 2 50 μmol/L依托泊苷处理细胞不同时间后ZNF451的相对表达(x±s) Table 2 ZNF451 expression level in A549 and HeLa cells after the treatment of 50 μmol/L etoposide for 0-24 h (x±s) |
|
注:a与同一时间对照组比较,t = 5.95、21.27、34.16,P < 0.01 A.蛋白表达量变化;B.生长曲线变化 图 1 ZNF451敲低后对细胞生长的影响 A. The expression level of ZNF451 after ZNF451knockdown; B. Cell growth ability was simulated after ZNF451 knockdown Figure 1 The effect of ZNF451 knockdown on cell growth |
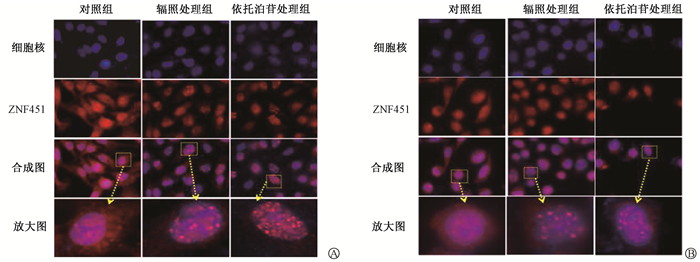
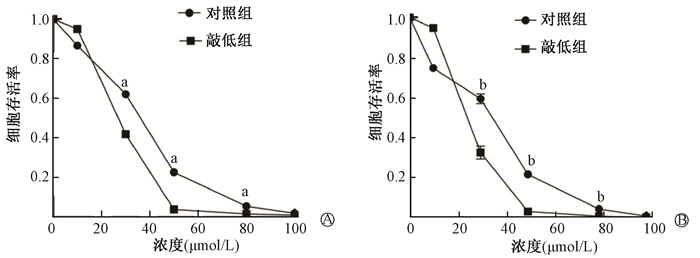
2. ZNF451对DNA损伤刺激的反应:6 Gy γ射线和50 μmol/L依托泊苷分别处理细胞6 h后,两处理组均出现了明显的焦点(foci)(图 2),说明ZNF451可以在DNA损伤位点形成foci。在30、50、80 μmol/L依托泊苷处理48 h后,ZNF451敲低组的细胞存活率均低于对照组(A549: t = 27.62、25.61、5.32, P < 0.01; HeLa: t = 30.77、21.28、4.18, P < 0.01),见图 3,表明ZNF451介导了辐射或依托泊苷诱导的DNA损伤反应,敲低ZNF451的A549和HeLa细胞在依托泊苷处理后增殖活力受到显著抑制。
|
图 2 依托泊苷(50 μmol/L)或6 Gy γ射线照射A549细胞(A)和HeLa细胞(B)6 h后ZNF451的定位变化荧光标记二抗染色×100 Figure 2 Immunofluorescence staining of the localization of ZNF451 in A549 cells (A) or HeLa cells (B) exposed to 50 μmol/L etoposide or 6 Gy γ-rays for 6 h ×100 |
|
注:a与同一浓度下A549对照组比较,t = 27.62、25.61、5.32,P < 0.01;b与同一浓度下HeLa对照组比较,t = 30.77、21.28、4.18,P < 0.01 图 3 ZNF451敲低组及对照组A549(A)和HeLa (B)细胞经不同浓度依托泊苷处理的存活曲线 Figure 3 Cell viabilities of ZNF451 knocked-down A549 (A) or HeLa (B) cells after the treatment of etoposide with different concentrations |
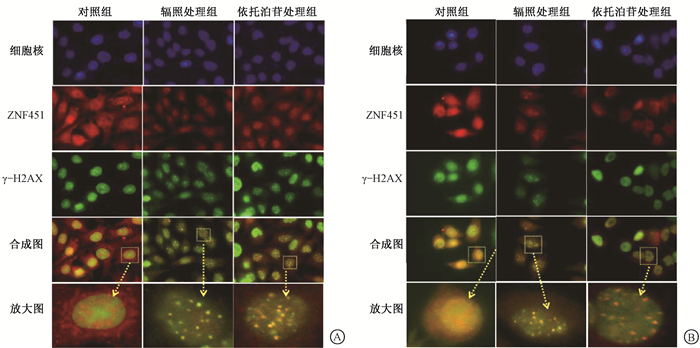
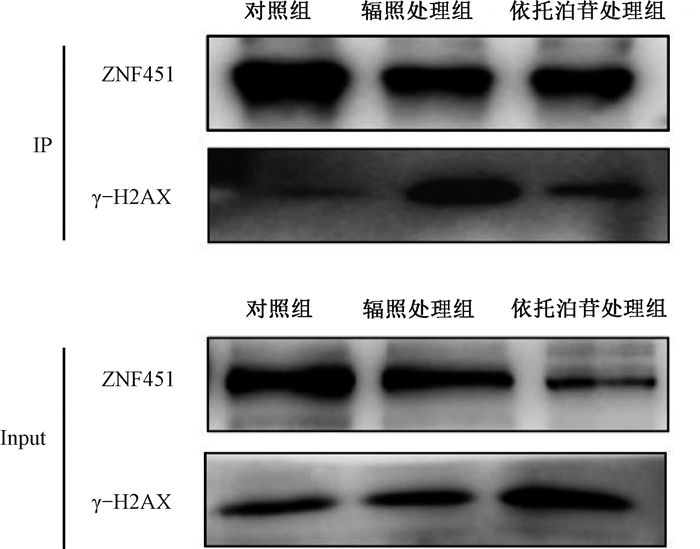
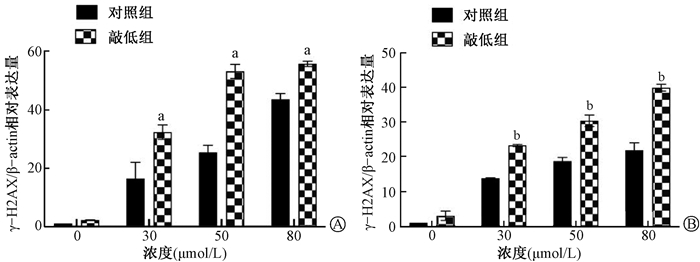
3. ZNF451对γ-H2AX表达的影响:结果如图 4所示,以6 Gy γ射线和50 μmol/L依托泊苷分别处理细胞6 h后,ZNF451和γ-H2AX在损伤位点均形成foci,且两者的foci在合成图中出现共定位现象,说明ZNF451与γ-H2AX在定位分布上存在密切的联系。此外,免疫共沉淀的结果表明,ZNF451与γ-H2AX具有内源性的相互作用(图 5)。如图 6所示,30、50、80 μmol/L依托泊苷诱导后,敲低组相较于对照组γ-H2AX的表达水平显著增加(A549:t = 6.12、10.67、4.68,P < 0.01;HeLa:t = 7.94、9.81、15.12,P < 0.01)。提示ZNF451通过与γ-H2AX相互作用而被招募至DNA损伤位点,抑制A549和HeLa细胞中的ZNF451蛋白表达量,加剧了依托泊苷诱导的DNA损伤水平。
|
图 4 6 Gy γ射线和50 μmol/L依托泊苷处理后A549(A)和HeLa(B)细胞中ZNF451和γ-H2AX免疫荧光分布图荧光标记二抗染色×100 Figure 4 Distribution of ZNF451 and γ-H2AX in A549 (A) and HeLa (B) cells after γ-ray irradiation and 50 μmol/L etoposide treatment ×100 |
|
注:IP. 以蛋白ZNF451作为诱饵蛋白,ZNF451的抗体做免疫沉淀,对靶蛋白γ-H2AX进行蛋白质印迹检测;Input. 阳性对照,用ZNF451和γ-H2AX的抗体进行蛋白质印迹检测 图 5 HeLa细胞中ZNF451和γ-H2AX免疫共沉淀图 Figure 5 Co-immunoprecipitation of ZNF451 and γ-H2AX in HeLa cells |
|
注:a与同一浓度下A549对照组比较,t = 6.12、10.67、4.68,P < 0.01;b与同一浓度下HeLa对照组比较,t = 7.94、9.81、15.12,P < 0.01 图 6 ZNF451敲低组及对照组A549(A)和HeLa(B)细胞经不同浓度依托泊苷处理后γ-H2AX的蛋白表达 Figure 6 The expression levels of γ-H2AX in ZNF451 knocked-down A549 (A) and HeLa (B) cells treated with different constructions of etoposide |
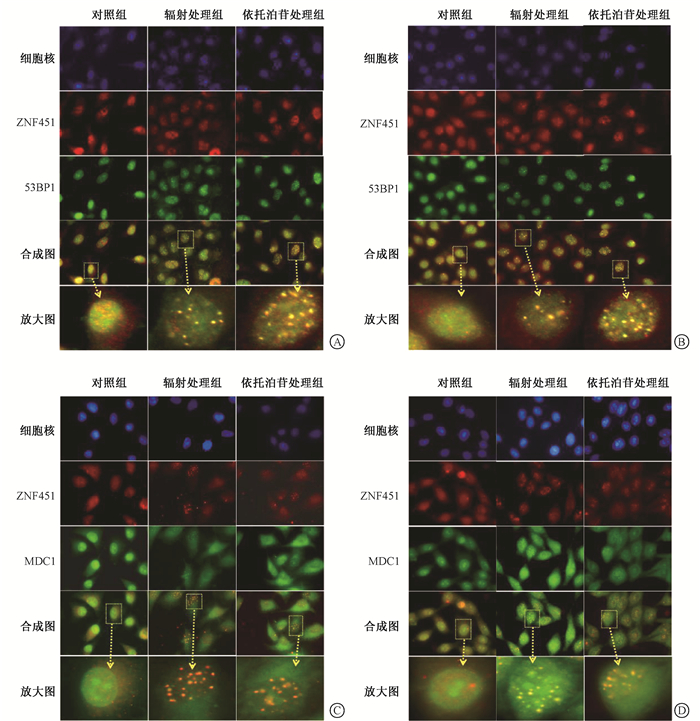
4. ZNF451与53BP1/MDC1形成共定位参与NHEJ修复:本研究利用NHEJ/DR-GFP报告质粒探究ZNF451参与的DNA损伤修复路径,敲低ZNF451会降低NHEJ的修复效率,敲低组的修复效率相当于对照组的76%(t = 18.60,P < 0.05),而HR的修复效率不受影响。以6 Gy γ射线和50 μmol/L依托泊苷分别处理细胞6 h,ZNF451与53BP1和MDC1的foci在合成图中存在共定位现象(图 7)。表明ZNF451通过与DNA损伤修复因子53BP1和MDC1共定位参与到NHEJ修复中。
|
A、B. A549、HeLa细胞中ZNF451和53BP1免疫荧光定位图;C、D. A549、HeLa细胞中ZNF451和MDC1免疫荧光定位图 图 7 辐射或依托泊苷处理对ZNF451与53BP1/MDC1蛋白共定位的影响荧光标记二抗染色×100 A, B. Immunofluorescence staining of the localizations of ZNF451 and 53BP1 in A549 and HeLa cells, respectively; C, D. Immunofluorescence staining of the localizations of ZNF451 and MDC1 in A549 and HeLa cells, respectively Figure 7 Influences of radiation or etoposide on the co-localization of ZNF451 and 53BP1/MDC1 protein in A549 and HeLa cells ×100 |
讨论
人类基因组DNA损伤可分为两大类:机体代谢引起的内源性损伤和环境因素引起的外源性损伤[12]。如果不及时修复DNA损伤,DSB会触发复杂的DNA损伤反应网络,包括DNA损伤的感知、DNA修复因子的组装、细胞凋亡、细胞周期传递的阻滞和DNA损伤修复的激活,以维持基因组的完整性[13-14]。
文献表明,ZNF451可能在调节DNA损伤修复和蛋白酶体降解的SUMO相关功能中发挥重要作用[15]。在不存在蛋白酶体的情况下,敲低ZATT会导致依托泊苷治疗中的细胞增敏现象和DSB修复的延迟[16]。因此,阐明ZNF451信号在DNA损伤修复中的作用对癌症的放化疗至关重要。使用临床化疗药物依托泊苷处理A549和HeLa细胞,ZNF451表达降低与依托泊苷处理的时间和剂量相关。本研究显示,ZNF451在受到DNA损伤刺激后,可以被招募到DNA损伤位点参与DNA损伤修复,降低ZNF451的表达,显著抑制了A549和HeLa细胞的增殖活力。这表明ZNF451作为一种调控因子参与DNA损伤修复和细胞的生长增殖过程。
DNA损伤反应是一系列信号级联放大途径,将各种DNA损伤修复因子准确有序地募集到DNA损伤位点。当DNA损伤发生时,PIKKs家族的成员如共济失调毛细血管扩张症突变基因(ATM)、共济失调毛细血管扩张症和Rad3相关激酶基因(ATR)、DNA依赖性蛋白激酶(DNA-PK)和聚(ADP-核糖)聚合酶(PARP)被激活[17],其中H2AX的Ser 139被ATM/ATR磷酸化并转化为γ-H2AX[18-19]。本研究显示,敲低ZNF451,γ-H2AX的表达水平增加,表明敲低ZNF451增强了A549和HeLa细胞对依托泊苷治疗诱导的DNA损伤。此外,免疫荧光结果与免疫共定位结果分别显示,ZNF451和γ-H2AX在DNA损伤后存在共定位和内源性的相互作用,进一步证实ZNF451直接参与了DNA损伤修复。
在真核生物中,HR和NHEJ是细胞修复DNA双链断裂的两种主要方式[20]。P53结合蛋白1(53BP1)是促进NHEJ的DSB加工反应的重要因素[21]。DNA损伤检查点介质1(MDC1)可以特异性识别γ-H2AX,并通过C端串联的两个乳腺癌易感基因羧基端(BRCT)结构域将其与DSB位点结合[22]。然后MDC1激活RNF8/RNF168介导的染色质泛素化,从而诱导53BP1募集到DNA损伤位点[22-24]。在本研究中,构建了I-SceI-HR/NHEJ报告基因系统,通过流式细胞术分析了细胞的荧光比,结果表明ZNF451影响NHEJ的修复效率,但不影响HR的修复效率。ZNF451与53BP1和MDC1在辐射和依托泊苷处理后有明显的共定位。这些结果证明ZNF451是γ-H2AX招募启动下游修复因子53BP1和MDC1的关键调节因子。
本研究表明在A549和HeLa细胞中敲低ZNF451的表达可阻断NHEJ修复通路,从而增强辐射或依托泊苷导致的DNA损伤水平。基于上述研究,推测ZNF451/MDC1/53BP1通路可能在增强A549和HeLa细胞放化疗敏感性研究中具有潜在的研究价值,ZNF451参与DNA损伤修复的具体机制还需进一步阐明。
利益冲突 无
作者贡献声明 夏琪负责实验和数据分析,撰写论文;赵国平指导实验和论文修改;吴李君负责实验设计
| [1] |
Sengupta R, Honey K. AACR Cancer Progress Report 2020: Turning science into lifesaving care[J]. Clin Cancer Res, 2020, 26(19): 5055. DOI:10.1158/1078-0432.CCR-20-3187 |
| [2] |
Sharma S, Raghavan SC. Nonhomologous DNA end joining[J]. Encyclopedia Cell Biol, 2016, 1: 451-455. DOI:10.1016/B978-0-12-394447-4.10047-1 |
| [3] |
Mahamud O, So J, Chua M, et al. Targeting DNA repair for precision radiotherapy: Balancing the therapeutic ratio[J]. Curr Probl Cancer, 2017, 41(4): 265-272. DOI:10.1016/j.currproblcancer.2017.04.002 |
| [4] |
Radhakrishnan SK, Jette N, Lees-Miller SP. Non-homologous end joining: emerging themes and unanswered questions[J]. DNA Repair (Amst), 2014, 17: 2-8. DOI:10.1016/j.dnarep.2014.01.009 |
| [5] |
Sanchez H, Reuter M, Yokokawa M, et al. Taking it one step at a time in homologous recombination repair[J]. DNA Repair (Amst), 2014, 20: 110-118. DOI:10.1016/j.dnarep.2014.02.012 |
| [6] |
Riccio AA, Schellenberg MJ, Williams RS. Molecular mechanisms of topoisomerase 2 DNA-protein crosslink resolution[J]. Cell Mol Life Sci, 2020, 77(1): 81-91. DOI:10.1007/s00018-019-03367-z |
| [7] |
Schellenberg MJ, Lieberman JA, Herrero-Ruiz A, et al. ZATT (ZNF451)-mediated resolution of topoisomerase 2 DNA-protein cross-links[J]. Science, 2017, 357(6358): 1412-1416. DOI:10.1126/science.aam6468 |
| [8] |
Karvonen U, Jääskeläinen T, Rytinki M, et al. ZNF451 is a novel PML body- and SUMO-associated transcriptional coregulator[J]. J Mol Biol, 2008, 382(3): 585-600. DOI:10.1016/j.jmb.2008.07.016 |
| [9] |
Koidl S, Eisenhardt N, Fatouros C, et al. The SUMO2/3 specific E3 ligase ZNF451-1 regulates PML stability[J]. Int J Biochem Cell Biol, 2016, 79: 478-487. DOI:10.1016/j.biocel.2016.06.011 |
| [10] |
Feng Y, Wu H, Xu Y, et al. Zinc finger protein 451 is a novel Smad corepressor in transforming growth factor-β signaling[J]. J Biol Chem, 2014, 289(4): 2072-2083. DOI:10.1074/jbc.M113.526905 |
| [11] |
Zeng W, Gu S, Yu Y, et al. ZNF451 stabilizes TWIST2 through SUMOylation and promotes epithelial-mesenchymal transition[J]. Am J Cancer Res, 2021, 11(3): 898-915. |
| [12] |
Lindahl T, Barnes DE. Repair of endogenous DNA damage[J]. Cold Spring Harb Symp Quant Biol, 2000, 65: 127-133. DOI:10.1101/sqb.2000.65.127 |
| [13] |
Panier S, Boulton SJ. Double-strand break repair: 53BP1 comes into focus[J]. Nat Rev Mol Cell Biol, 2014, 15(1): 7-18. DOI:10.1038/nrm3719 |
| [14] |
Song Y, Park SY, Wu Z, et al. Hybrid inhibitors of DNA and HDACs remarkably enhance cytotoxicity in leukaemia cells[J]. J Enzyme Inhib Med Chem, 2020, 35(1): 1069-1079. DOI:10.1080/14756366.2020.1754812 |
| [15] |
Eisenhardt N, Chaugule VK, Koidl S, et al. A new vertebrate SUMO enzyme family reveals insights into SUMO-chain assembly[J]. Nat Struct Mol Biol, 2015, 22(12): 959-967. DOI:10.1038/nsmb.3114 |
| [16] |
Zagnoli-Vieira G, Caldecott KW. TDP2, TOP2, and SUMO: what is ZATT about?[J]. Cell Res, 2017, 27(12): 1405-1406. DOI:10.1038/cr.2017.147 |
| [17] |
Falck J, Coates J, Jackson SP. Conserved modes of recruitment of ATM, ATR and DNA-PKcs to sites of DNA damage[J]. Nature, 2005, 434(7033): 605-611. DOI:10.1038/nature03442 |
| [18] |
Matsuoka S, Ballif BA, Smogorzewska A, et al. ATM and ATR substrate analysis reveals extensive protein networks responsive to DNA damage[J]. Science, 2007, 316(5828): 1160-1166. DOI:10.1126/science.1140321 |
| [19] |
Rogakou EP, Pilch DR, Orr AH, et al. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139[J]. J Biol Chem, 1998, 273(10): 5858-5868. DOI:10.1074/jbc.273.10.5858 |
| [20] |
Pfister SX, Ahrabi S, Zalmas LP, et al. SETD2-dependent histone H3K36 trimethylation is required for homologous recombination repair and genome stability[J]. Cell Rep, 2014, 7(6): 2006-2018. DOI:10.1016/j.celrep.2014.05.026 |
| [21] |
Panier S, Boulton SJ. Double-strand break repair: 53BP1 comes into focus[J]. Nat Rev Mol Cell Biol, 2014, 15(1): 7-18. DOI:10.1038/nrm3719 |
| [22] |
Zhou T, Yi F, Wang Z, et al. The functions of DNA damage factor RNF8 in the pathogenesis and progression of cancer[J]. Int J Biol Sci, 2019, 15(5): 909-918. DOI:10.7150/ijbs.31972 |
| [23] |
Bekker-Jensen S, Mailand N. The ubiquitin- and SUMO-dependent signaling response to DNA double-strand breaks[J]. FEBS Lett, 2011, 585(18): 2914-2919. DOI:10.1016/j.febslet.2011.05.056 |
| [24] |
Lukas J, Lukas C, Bartek J. More than just a focus: The chromatin response to DNA damage and its role in genome integrity maintenance[J]. Nat Cell Biol, 2011, 13(10): 1161-1169. DOI:10.1038/ncb2344 |
 2022, Vol. 42
2022, Vol. 42


