2. 军事科学院军事医学研究院辐射医学研究所 北京市放射生物学重点实验室,北京 100850
2. Beijing Key Laboratory for Radiobiology, Institute of Radiation Medicine, Academy of Military Medical Sciences, Academy of Military Sciences, Beijing 100850, China
DNA双链断裂(DSBs)是最严重的DNA损伤类型之一,由外源性因素及内源性代谢应激产生[1-2]。DSB修复缺陷导致基因组不稳定,细胞修复DSBs对于维持基因组稳定和影响细胞放射敏感性密切相关[3-4]。DSBs主要由非同源末端连接(NHEJ)和同源重组(HR)两种途径修复[5]。NHEJ是一种易错的修复方式,其可发生于细胞分裂的所有阶段[6-8]。与NHEJ相比,HR修复是一种更为保守和无错的修复[9-10]。
TAB182是一个与多聚ADP核糖聚合酶家族成员TNKS1相互作用的蛋白,因此又称TNKS1BP1[11-14]。TAB182通过肌动蛋白骨架重排调节癌细胞的侵袭[11];腺病毒感染后,TAB182被靶向降解;且TAB182是分解代谢阻抑物4的一个组成部分,具有调节转录功能、去腺苷酸化和E3连接酶活性[15]。在HeLa细胞中,辐射照射后TAB182表达上调,TAB182的缺失增加癌细胞的放射敏感性。在A549细胞中,TAB182通过抑制RAD51焦点(foci)的形成影响同源重组途径,调节基因组的稳定性[16-17]。且TAB182缺失的细胞过表达TAB182可以恢复G2/M检查点及放射抗性[18]。通过公共数据库的数据发现,TAB182高表达与乳腺癌患者的生存率呈现正相关关系(Proteinatlas.Version: 20.1:http://www.proteinatlas.org/)。TAB182在DSB修复中发挥着重要作用,本研究旨在阐述TAB182如何调控DSBs的HR修复及反应机制,为TAB182作为临床放疗中的治疗靶点提供参考。
材料与方法1. 试剂:anti-TAB182购自美国Santa Cruz公司,anti-actin购自北京中杉金桥公司,anti-RPA2/32、anti-RAD51购自英国Abcam公司,anti-BrdU购自武汉Proteintech公司,anti-FLAG购自美国Sigma公司;Anti-Alexa FluorTM488,Anti-Alexa Fluor@568,购自美国Life technology公司;嘌呤霉素、lipofectmine2000 (11668-019) 购自美国Invitrogen公司,放射菌素D(Act-D)购自美国Selleck公司,Olaparib购自美国Selleck公司,反转录试剂、qPCR试剂购自日本TOYOBO公司,凋亡试剂、周期试剂购自日本DOJINDO公司。
2. 细胞培养:人乳腺癌细胞(MCF-7细胞)购自美国模式培养物集存库(ATCC)细胞库,由本实验室保存。MCF-7细胞在含有10%胎牛血清,100 U/ml青霉素和100 μg/ml链霉素的高糖培养基DMEM中生长,细胞培养均在37℃,5% CO2的细胞培养箱中进行。
3. 细胞照射:在军事科学院军事医学研究院辐射医学研究所用60Co γ射线进行8 Gy单次照射,60.3 cGy/min,源靶距3 m,照射后4 h收样。
4. 慢病毒的培养与感染:TAB182的慢病毒液购自苏州吉玛基因公司,TAB182 shRNA序列为:5′AAGACGAGGAGUAAUCUUCACCCUG 3′(sense),将TAB182慢病毒液感染MCF-7细胞,加入2 μg/ml嘌呤霉素进行筛选得到稳定表达的细胞系。shRNA慢病毒感染后筛选TAB182敲低的MCF-7细胞为TAB182沉默组,加入慢病毒阴性对照物TAB182未敲低的MCF-7细胞为TAB182阴性对照组。
5. qRT-PCR:细胞经过处理后,磷酸盐缓冲溶液(PBS)洗3遍,加入TRIzol裂解液,按照RNA提取步骤提取RNA,用NANO Drop 2000C(美国赛默飞科技公司)测定RNA浓度。将得到的RNA储存在-80℃或进行下一步实验,按试剂盒说明书进行反转录及qRT-PCR反应。RPA2上游引物:5′AGGGAGAGCACCTATCAGCA 3′,下游引物:5′AGATGTGCCCCTCATTGCTC 3′。β-肌动蛋白上游引物:5′TGGCACCCAGCACAATGAA 3′,下游引物5′CTAAGTCATAGTCCGCCTAGAAGCA 3′。反应完成后,对实验数据以2-ΔΔCt法计算出目的基因mRNA表达水平的相对变化。此实验检测RPA2基因在mRNA的表达。
6. 放射菌素D处理:在对照组细胞及TAB182沉默组细胞中添加RNA合成抑制剂放射菌素D(Act-D)处理,在不同时间点收取细胞,qRT-PCR检测RPA2 mRNA表达水平的相对变化。
7. 免疫印迹实验:细胞经过处理后,用PBS洗3遍,收集细胞,3 000 r/min、离心半径6 cm,离心3 min,弃上清。加入适量细胞裂解液,冰上裂解15~20 min,12 000 r/min,离心半径6 cm,离心10 min,取上清,用BCA法沉淀蛋白浓度。加入5 × SDS上样缓冲液(loading buffer)沸水中煮10 min,每孔按40 μg的蛋白进行凝胶电泳,随后转移至硝酸纤维素膜(NC)膜上,用牛奶室温封闭1 h,然后与配好的一抗4℃孵育过夜。次日,等渗盐溶液加Tris-HCl缓冲液(TBST)清洗3次后,加入相应二抗,室温孵育1 h,TBST清洗3次,显影液按1 ∶1的比例配制,显影,观察结果。此实验用于检测RPA2蛋白水平的表达。
8. 免疫荧光实验:取对数生长期的各组细胞,接种于6孔培养板中;次日,60 Co γ射线8 Gy照射后4 h吸去细胞培养液,PBS溶液洗涤3次,加入4%多聚甲醛后常温固定30 min;用通透液(0.25% Triton X-100/PBS)通透15 min,用牛血清蛋白(BSA)封闭液37℃封闭1 h,然后与稀释好的一抗4℃孵育过夜;加入稀释好的二抗,室温避光孵育1 h;封片,4℃保存;使用共聚焦显微镜观察各组细胞。此实验用于检测RPA2、Rad51、BrdU荧光水平的表达。
9. 集落形成实验:用PBS将对数生长期细胞洗3遍,用胰酶消化制备成细胞悬液,进行细胞计数,将细胞接种于6孔板中,接种密度为每孔200、200、400、1 000和2 000个细胞,12 h后进行0、1、2、4和8 Gy照射处理,并继续培养10 d,姬姆萨染液固定染色,计数各组细胞克隆数量并进行分析。
10. 细胞凋亡:将对数生长期的细胞以每升1×105的密度接种于6孔板中,12 h后进行8 Gy照射,24及48 h后用胰酶消化,1 000 r/min,离心半径11 cm,离心3 min,去上清,PBS清洗两遍。每个样本依次加入500 μl结合缓冲液(binding buffer)、5 μl磷脂结合蛋白-异硫氰酸荧光素(annexin V-FITC)、5 μl碘化丙啶(PI),用TAB182阴性对照组照射后的细胞额外分出3组,一组不加染液,一组加入磷脂结合蛋白-异硫氰酸荧光素染液及一组加入碘化丙啶,室温混匀避光反应15 min,上机检测。
11. 细胞周期检测:用PBS将对数生长期细胞洗3遍后用胰酶消化制备成细胞悬液,传代至培养皿中,待细胞生长密度达到60%~80%时,对细胞进行8 Gy 60Co γ射线照射,照后4、8、12、24 h收取细胞,未照射细胞设为对照组。细胞经PBS洗3遍,胰酶消化后加入培养基终止消化,3 000 r/min,离心半径6 cm,离心3 min。收集的细胞用70%的乙醇4℃固定过夜。第2天加入300 μl含RNAase的PI溶液重悬细胞,室温孵育10~30 min,流式细胞仪检测细胞周期的变化。
12. 统计学处理:采用SPSS 24.0统计软件进行分析,数据符合正态分布,计量资料以x±s表示。两组间比较采用独立样本t检验。计数资料采用百分率表示,组间比较采用χ2检验。P < 0.05为差异有统计学意义。
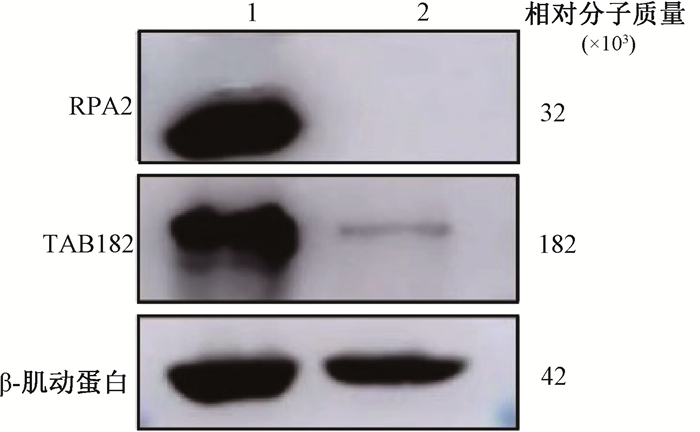
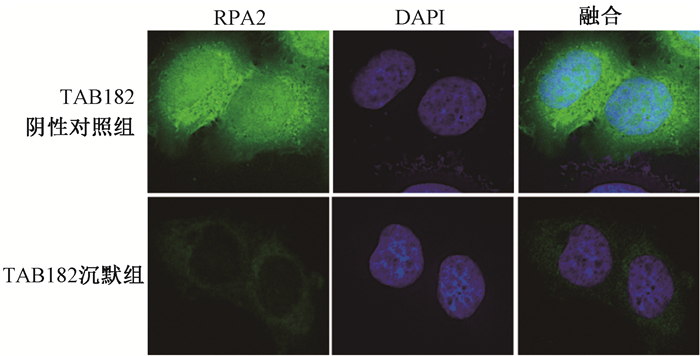
结果1. TAB182影响HR修复蛋白RPA2的表达:对TAB182沉默组细胞进行了RNA测序,发现DNA损伤修复相关蛋白RPA2 mRNA表达降低。在TAB182沉默组的细胞中,RPA2的mRNA表达水平降低了73%,差异具有统计学意义(t=17.97,P < 0.05);相比于TAB182阴性对照组,TAB182沉默组细胞的RPA2蛋白水平也降低了97%,见图 1。相比于TAB182阴性对照组,TAB182沉默组中RPA2的foci显著降低,见图 2。提示TAB182的减少导致作为HR修复途径中的重要蛋白RPA2减少,可能影响DNA损伤的HR修复途径。
|
注:1.TAB182阴性对照组;2.TAB182沉默组 图 1 RPA2蛋白在TAB182阴性对照组和TAB182沉默组表达的变化 Figure 1 RPA2 protein expression in TAB182 negative control group and TAB182 knockdown group |
|
Anti-Alexa Fluor 488 568二抗染色×100 图 2 RAP2 foci在TAB182阴性对照组和TAB182沉默组表达的变化 Anti-Alexa Fluor 488 568 secondary antibody ×100 Figure 2 RPA2 foci expression in TAB182 negative control group and TAB182 knockdown group |
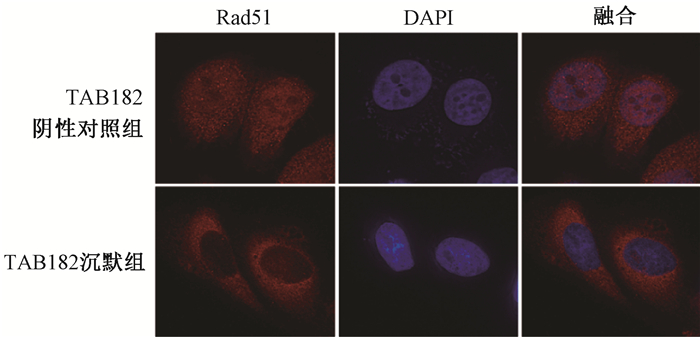
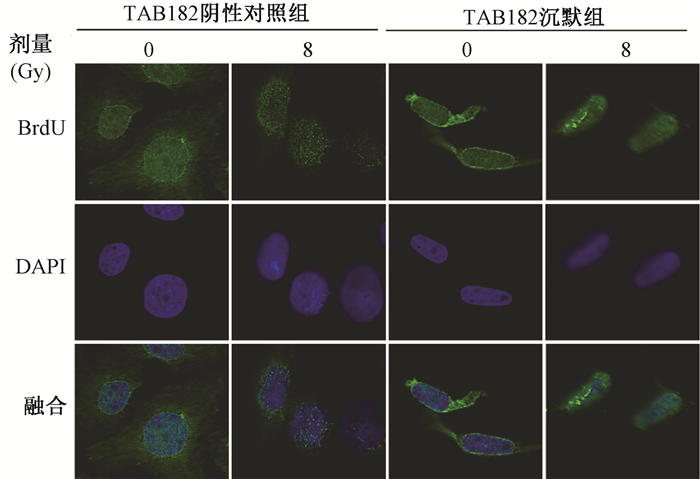
2. TAB182的缺失导致同源重组修复异常:相比于TAB182阴性对照组,在TAB182沉默组的细胞中,Rad51在细胞核的定位显著减少,见图 3。照射后,TAB182阴性对照组的BrdU foci水平显著高于TAB182沉默组的细胞,见图 4。提示TAB182的缺失可影响HR修复过程中的关键蛋白Rad51及BrdU的形成,导致同源重组修复异常。
|
Anti-Alexa Fluor 488 568二抗染色×100 图 3 Rad51 foci在TAB182阴性对照组和TAB182沉默组的表达变化 Anti-Alexa Fluor 488 568 secondary antibody ×100 Figure 3 Rad51 foci expression in TAB182 negative control group and TAB182 knockdown group |
|
Anti-Alexa Fluor 488 568二抗染色×100 图 4 BrdU foci在TAB182阴性对照组和TAB182沉默组形成的变化 Anti-Alexa Fluor 488 568 secondary antibody ×100 Figure 4 The formation of BrdU foci in TAB182 negative control group and TAB182 knockdown group |
3. TAB182对RPA2 mRNA稳定性的影响:在TAB182阴性对照组及TAB182沉默组细胞中分别转染不同浓度的FLAG-RPA2质粒,结果表明TAB182不影响RPA2的外源性表达,见图 5。相比于TAB182阴性对照组,放射菌素D处理后第4、8、12小时,TAB182沉默组细胞中的RPA2 mRNA的衰减加快,差异具有统计学意义(t=5.37、3.79、3.69,P < 0.05),见图 6。且TAB182沉默组RPA2 mRNA衰减一半的速度是TAB182阴性对照组的3倍(2 h和6 h),差异具有统计学意义(t=3.48,P < 0.05)。表明TAB182通过维持RPA2 mRNA的稳定性,从而促进同源重组修复。
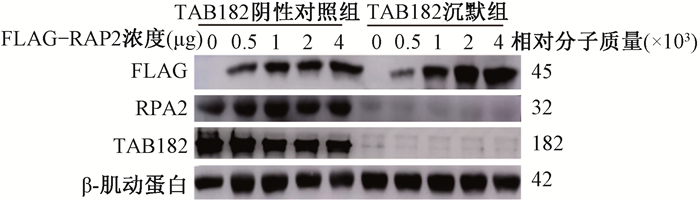
|
图 5 FLAG-RPA2在TAB182阴性对照组和TAB182沉默组表达的变化 Figure 5 FLAG-RPA2 expression in TAB182 negative control group and TAB182 knockdown group |
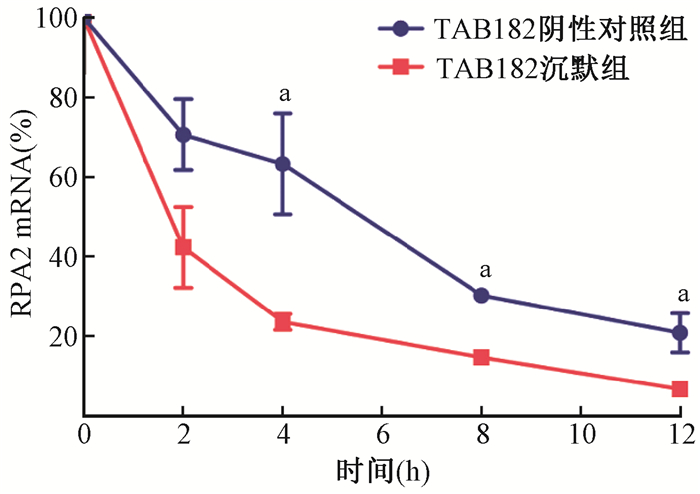
|
注:a与同一时间TAB182阴性对照组比较,t=5.37、3.79、3.69,P < 0.05 图 6 Act-D处理后不同时间RPA2 mRNA在TAB182阴性对照组和TAB182沉默组表达的变化 Figure 6 RPA2 mRNA expression in TAB182 negative control group and TAB182 knockdown group at different times after Act-D treatment |
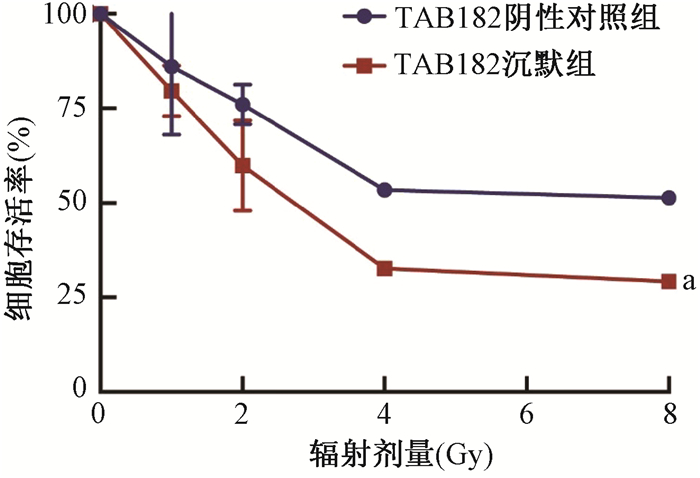
4. TAB182沉默对细胞放射敏感性的影响:照射后TAB182沉默组的细胞凋亡率较TAB182阴性对照组显著增高,差异具有统计学意义(12.94% vs.7.24%;t=11.05,P < 0.001),见图 7。相比于TAB182阴性对照组,TAB182沉默组的细胞在第24小时周期G2/M的阻滞时间延长,差异具有统计学意义(t=8.40,P < 0.01),见图 8。相比于TAB182阴性对照组,TAB182沉默组的细胞对辐照更加敏感,差异具有统计学意义(t=8.18,P < 0.05),见图 9。上述结果揭示了TAB182缺失的细胞对辐照十分敏感。
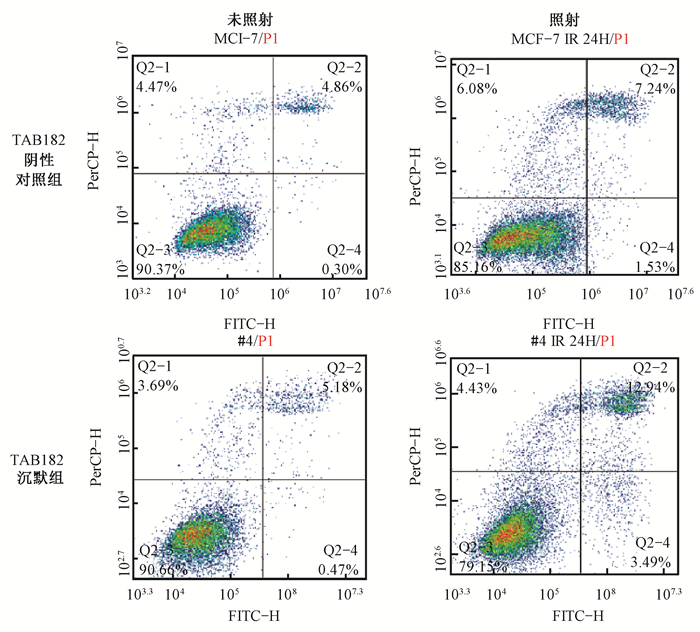
|
图 7 照射对TAB182阴性对照组和TAB182沉默组细胞凋亡的影响 Figure 7 Effects of irradiation on apoptosis of cells in TAB182 negative control group and TAB182 knockdown group |
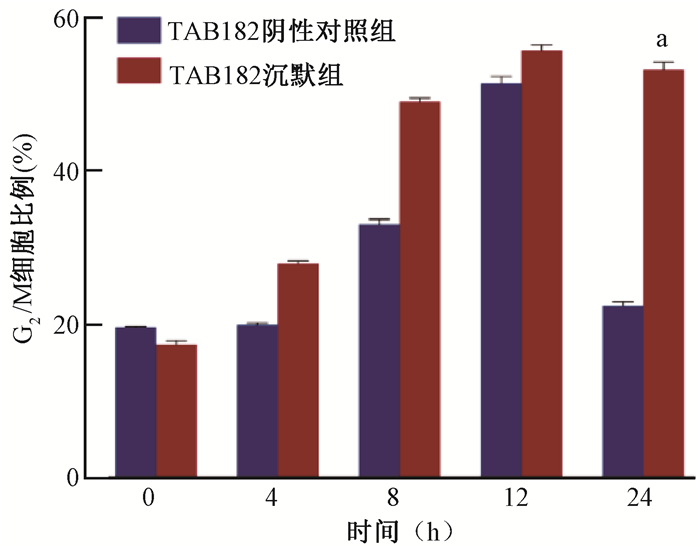
|
注:a与TAB182阴性对照组比较,t=8.40,P < 0.01 图 8 照射后不同时间TAB182阴性对照组和TAB182沉默组细胞周期的比例变化 Figure 8 Cell cycle distributions of TAB182 negative control group and TAB182 knockdown group at different times after irradiation |
|
注:a与TAB182阴性对照组比较,t=8.18,P < 0.05 图 9 照射对TAB182阴性对照组和TAB182沉默组细胞存活率的影响 Figure 9 Effects of irradiation on the survival rate of TAB182 negative control group and TAB182 knockdown group |
讨论
研究表明,TAB182作为一种DNA损伤修复相关蛋白参与DNA双链断裂修复,在A549细胞中通过HR途径参与DNA双链断裂修复,且主要是通过抑制Rad51 foci的形成[16],但其具体机制还不清楚。本研究发现,在TAB182沉默的乳腺癌MFC-7细胞中RPA2的表达降低。Rad51作为RPA2下游蛋白,RPA2的减少导致Rad51招募到DNA损伤位点减少,与之前的研究报道TAB182通过抑制Rad51 foci的形成参与HR修复结果相吻合。BrdU是单链DNA标志物,HR修复途径末端剪切后生成单链DNA,照射后发现,TAB182沉默组的细胞BrdU foci数量显著少于TAB182阴性对照组,说明在DNA损伤后,由于TAB182的缺失导致RPA2的降低,DNA断裂末端ssDNA可能因未受保护而被降解,或者可能影响到HR途径上游的末端剪切过程。上述研究表明,TAB182可能通过影响RPA2的表达参与HR修复,但TAB182调节RPA2蛋白水平的机制还不清楚。
本研究添加mRNA合成抑制剂Act-D观察TAB182阴性对照组以及TAB182沉默组的MCF-7细胞中RPA2 mRNA稳定性,结果表明TAB182的缺失使RPA2 mRNA半衰期变短,说明TAB182通过促进RPA2 mRNA的稳定性进而促进RPA2的蛋白表达。在TAB182沉默组的MCF-7细胞系中梯度转染外源性FLAG-RPA2质粒,发现TAB182的缺失不影响外源性RPA2的表达。因与细胞内源RPA2基因不同,外源FLAG-RPA2表达载体中只含有RPA2的外显子区域,因此推测TAB182还有可能参与RPA2 mRNA的剪接与成熟过程。
TAB182通过维持RPA2 mRNA的稳定性参与HR修复。TAB182的缺失会损害同源重组修复,因此TAB182对细胞的生存十分重要。凋亡及集落形成实验发现,TAB182沉默组的细胞表现出更强的放射敏感性。但形成的细胞集落大小大于TAB182阴性对照组,与之前研究报道的TAB182的缺失可以加快细胞的S期进程,促使细胞的增殖加快相一致[17]。细胞周期检测结果表明,TAB182的缺失可延长电离辐射诱发MCF-7细胞的G2/M周期阻滞,此结果与本实验室前期报道在TAB182沉默的HeLa细胞中可引起G2/M期阻滞延长相吻合[19]。有研究报道TAB182可以通过G2/M期细胞累积,调节癌细胞的放射敏感性[18]。通过数据库挖掘的数据发现TAB182与乳腺癌患者的生存呈正相关,分子水平也验证了TAB182对乳腺癌细胞的生存十分重要。
同源重组修复(HR)在DNA双链断裂(DSBs)的修复中发挥重要作用。HR修复依赖姐妹染色单体,这种特性限制了HR修复只能发生在S/G2期[20-21]。HR修复通过末端剪切形成的3′ ssDNA将被RPA复合物包裹,RPA复合物由RPA1、RPA2和RPA3亚单位组成[22]。RPA2是一种ssDNA结合蛋白,可以保护3′ssDNA末端免被降解,并防止ssDNA自身退火形成二级结构[23]。随后,RPA复合物会被RAD51所取代,RAD51与DNA相互作用形成RAD51核纤丝结构,RAD51能促进ssDNA与同源DNA模板的结合,从而促进D环的形成[24-25]。RPA2作为HR途径的核心分子发挥着重要作用。本研究揭示了TAB182能促进RPA2 mRNA稳定性,维护HR修复功能。
总而言之,TAB182通过调控RPA2 mRNA的稳定性参与HR途径介导的DNA双链断裂修复,且TAB182缺失的肿瘤细胞对电离辐射高度敏感。在未来的工作中,需进一步探索TAB182是如何正确精准地进行DNA修复,降低肿瘤细胞的DNA损伤修复能力,从而为抗肿瘤治疗提供一个更好的应用前景。
利益冲突 无
作者贡献声明 李刚负责实验操作,撰写论文;赖淑婷、韩阳协助实验操作;白琛俊、关华提供相关建议;高山山、周平坤指导课题设计和修改论文
| [1] |
Kong M, Greene EC. Mechanistic insights from single-molecule studies of repair of double strand breaks[J]. Front Cell Dev Biol, 2021, 9: 745311. DOI:10.3389/fcell.2021.745311 |
| [2] |
Ohnishi T, Mori E, Takahashi A. DNA double-strand breaks: their production, recognition, and repair in eukaryotes[J]. Mutat Res, 2009, 669(1-2): 8-12. DOI:10.1016/j.mrfmmm.2009.06.010 |
| [3] |
Tubbs A, Nussenzweig A. Endogenous DNA damage as a source of genomic instability in cancer[J]. Cell, 2017, 168(4): 644-656. DOI:10.1016/j.cell.2017.01.002 |
| [4] |
Streffer C. Strong association between cancer and genomic instability[J]. Radiat Environ Biophys, 2010, 49(2): 125-131. DOI:10.1007/s00411-009-0258-4 |
| [5] |
Lieber MR. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway[J]. Annu Rev Biochem, 2010, 79: 181-211. DOI:10.1146/annurev.biochem.052308.093131 |
| [6] |
Menon V, Povirk LF. End-processing nucleases and phosphodiesterases: An elite supporting cast for the non-homologous end joining pathway of DNA double-strand break repair[J]. DNA Repair (Amst), 2016, 43: 57-68. DOI:10.1016/j.dnarep.2016.05.011 |
| [7] |
Pryor JM, Conlin MP, Carvajal-Garcia J, et al. Ribonucleotide incorporation enables repair of chromosome breaks by nonhomologous end joining[J]. Science, 2018, 361(6407): 1126-1129. DOI:10.1126/science.aat2477 |
| [8] |
Chang H, Pannunzio NR, Adachi N, et al. Non-homologous DNA end joining and alternative pathways to double-strand break repair[J]. Nat Rev Mol Cell Biol, 2017, 18(8): 495-506. DOI:10.1038/nrm.2017.48 |
| [9] |
Yao Y, Li X, Chen W, et al. ATM promotes RAD51-mediated meiotic DSB repair by inter-sister-chromatid recombination in arabidopsis[J]. Front Plant Sci, 2020, 11: 839. DOI:10.3389/fpls.2020.00839 |
| [10] |
Mazón G, Mimitou EP, Symington LS. SnapShot: Homologous recombination in DNA double-strand break repair[J]. Cell, 2010, 142(4): 646, 646.e1. DOI:10.1016/j.cell.2010.08.006 |
| [11] |
Chalabi Hagkarim N, Ryan EL, Byrd PJ, et al. Degradation of a novel DNA damage response protein, tankyrase 1 binding protein 1, following adenovirus infection[J]. J Virol, 2018, 92(12): e02034-17. DOI:10.1128/JVI.02034-17 |
| [12] |
Seimiya H, Smith S. The telomeric poly(ADP-ribose) polymerase, tankyrase 1, contains multiple binding sites for telomeric repeat binding factor 1 (TRF1) and a novel acceptor, 182-kDa tankyrase-binding protein (TAB182)[J]. J Biol Chem, 2002, 277(16): 14116-14126. DOI:10.1074/jbc.M112266200 |
| [13] |
Sbodio JI, Chi NW. Identification of a tankyrase-binding motif shared by IRAP, TAB182, and human TRF1 but not mouse TRF1. NuMA contains this RXXPDG motif and is a novel tankyrase partner[J]. J Biol Chem, 2002, 277(35): 31887-31892. DOI:10.1074/jbc.M203916200 |
| [14] |
卫秦芝, 庄志雄. Tankyrase的结构和功能的研究进展[J]. 卫生毒理学杂志, 2004, 18(4): 259-262. Wei QZ, Zhuang ZX. Research progress on the structure and function of Tankyrase[J]. J Health Toxicol, 2004, 18(4): 259-262. DOI:10.3969/j.issn.1002-3127.2004.04.023 |
| [15] |
Ohishi T, Yoshida H, Katori M, et al. Tankyrase-binding protein TNKS1BP1 regulates actin cytoskeleton rearrangement and cancer cell invasion[J]. Cancer Res, 2017, 77(9): 2328-2338. DOI:10.1158/0008-5472.CAN-16-1846 |
| [16] |
Tan W, Guan H, Zou LH, et al. Overexpression of TNKS1BP1 in lung cancers and its involvement in homologous recombination pathway of DNA double-strand breaks[J]. Cancer Med, 2017, 6(2): 483-493. DOI:10.1002/cam4.995 |
| [17] |
Zou LH, Shang ZF, Tan W, et al. TNKS1BP1 functions in DNA double-strand break repair though facilitating DNA-PKcs autophosphorylation dependent on PARP-1[J]. Oncotarget, 2015, 6(9): 7011-7022. DOI:10.18632/oncotarget.3137 |
| [18] |
Cao Y, Gao A, Li X, et al. Elevated TAB182 enhances the radioresistance of esophageal squamous cell carcinoma through G2-M checkpoint modulation[J]. Cancer Med, 2021, 10(9): 3101-3112. DOI:10.1002/cam4.3879 |
| [19] |
邹联洪, 徐勤枝, 刘晓丹, 等. TAB182对电离辐射诱发细胞周期G2/M阻滞的调节作用[J]. 中国生物化学与分子生物学报, 2012, 28(9): 811-817. Zou LH, Xu QZ, Liu XD, et al. The regulatory effect of TAB182 on ionizing radiation-induced cell cycle G2/M arrest[J]. Chin J Biochem Mol Biol, 2012, 28(9): 811-817. |
| [20] |
Trenner A, Sartori AA. Harnessing DNA double-strand break repair for cancer treatment[J]. Front Oncol, 2019, 9: 1388. DOI:10.3389/fonc.2019.01388 |
| [21] |
Jiang M, Jia K, Wang L, et al. Alterations of DNA damage response pathway: Biomarker and therapeutic strategy for cancer immunotherapy[J]. Acta Pharm Sin B, 2021, 11(10): 2983-2994. DOI:10.1016/j.apsb.2021.01.003 |
| [22] |
Symington LS, Gautier J. Double-strand break end resection and repair pathway choice[J]. Annu Rev Genet, 2011, 45: 247-271. DOI:10.1146/annurev-genet-110410-132435 |
| [23] |
Maréchal A, Zou L. RPA-coated single-stranded DNA as a platform for post-translational modifications in the DNA damage response[J]. Cell Res, 2015, 25(1): 9-23. DOI:10.1038/cr.2014.147 |
| [24] |
Allera-Moreau C, Rouquette I, Lepage B, et al. DNA replication stress response involving PLK1, CDC6, POLQ, RAD51 and CLASPIN upregulation prognoses the outcome of early/mid-stage non-small cell lung cancer patients[J]. Oncogenesis, 2012, 1(10): e30. DOI:10.1038/oncsis.2012.29 |
| [25] |
Atwell S, Disseau L, Stasiak AZ, et al. Probing Rad51-DNA interactions by changing DNA twist[J]. Nucleic Acids Res, 2012, 40(22): 11769-11776. DOI:10.1093/nar/gks1131 |
 2022, Vol. 42
2022, Vol. 42


