2. 郑州大学医学科学院,郑州 450001;
3. 洛桑大学附属医院放疗科,洛桑 瑞士 1002
2. Academy of Medical Science, Zhengzhou University, Zhengzhou 450001, China;
3. Department of Radiation Oncology, Lausanne University Hospital, Lausanne 1002, Switzerland
超高剂量率(FLASH)放疗在超高剂量率的基础上(通常>40 Gy/s),可于极短时间内将设定的放疗剂量全部注入肿瘤靶区。在保持相同抗肿瘤效果的同时,该技术可明显减轻正常组织放射损伤,这一特性被称为FLASH效应[1]。FLASH放疗对于改进肿瘤放疗技术,提高治疗效率,减轻放疗相关不良反应具有重要意义,受到了放射肿瘤学界的极大关注。瑞士洛桑大学附属医院2019年报告了全球首例实施FLASH放疗的皮肤CD30+T细胞淋巴瘤患者[2],此外,国际上有两项临床试验(NCT04986696、NCT04592887)正在开展中。目前,对于FLASH效应的生物学机制已经有了初步的认识。然而,既往对于常规剂量率放疗敏感的患者,是否也对FLASH放疗敏感,以及是否存在一些关键基因参与调控FLASH放疗敏感性,这些问题仍鲜有研究。寻找FLASH放疗敏感性基因,对于发展精准放疗及探索FLASH放疗的适用患者群体,至关重要。本研究收集基因表达综合(GEO)数据库中有关FLASH放疗的芯片数据,采用生物信息学方法筛选调控FLASH放疗敏感性的枢纽基因及信号通路,与常规剂量率放疗进行对比,以期为进一步了解FLASH放疗的生物学机制及筛选适合FLASH放疗的患者群体提供新思路。
资料与方法1. 基因芯片数据:以“FLASH radiotherapy”和“carcinoma”、“neoplasma”、“cancer”、“tumor”为关键词在GEO数据库(https://www.ncbi.nlm.nih.gov/gds)中检索与FLASH放疗相关的基因表达信息。本研究选择GSE100718芯片数据集,该数据由来自法国的研究团队提供,样本来自于急性T淋巴细胞白血病患者,其中M106和M114为FLASH放疗敏感组,M108为常规放疗敏感组[3-4]。具体放疗剂量参数见表 1。
|
|
表 1 放疗剂量参数 Table 1 Main parameters of radiotherapy |
2. 差异基因筛选:使用limma包进行差异基因分型,筛选条件设置为差异表达上调或者下调2倍(log2FC绝对值≥1),且P<0.01。对比FLASH放疗敏感组与常规放疗敏感组,筛选出差异表达基因,后续用于基因富集分析。
3. 差异表达基因的生物信息学分析:基因富集分析采用R包clusterProfiler并在R软件(version 3.6.2)中实现。对筛选出的差异表达基因进行基因本体论(gene ontology,GO)、特征基因集合和疾病本体论(disease ontology, DO)分析。分析差异基因主要参与的生物学过程以及主要涉及的信号转导通路,以P<0.01作为纳入标准。研究中应用的R包主要有: clusterProfiler(version 3.14.3), pathview (version 3.11)。
4. 蛋白相互作用(PPI)网络的构建和枢纽(Hub)基因的筛选:使用STRING(http://string-db.org,用于检索相互作用基因的搜索工具,旨在预测蛋白质-蛋白质相互作用)在线工具,以构建选定基因的PPI网络。然后,选择得分≥0.4的基因,使用Cytoscape插件(v3.7.2) 构建可视化网络模型。采用最大集团中心度(maximal clique centrality, MCC) 算法寻找中心节点。每个节点的MCC由Cytoscape中的插件CytoHubba计算得到,选取MCC值排名前5的基因为Hub基因。
5. Hub基因的表达验证及临床意义:采用肿瘤基因组图谱(TCGA)和基因组织表达(GTEx)数据库验证所筛选出的Hub基因在急性T淋巴细胞白血病和正常组织中的表达。
结果1. 差异基因的筛选:通过GEO数据库中获得的GSE100718芯片信息,FLASH放疗敏感组(M106和M114)与常规放疗敏感组(M108)对比后共筛选出12 800例差异表达基因,其中表达量上调基因7 251例,表达量下调基因5 549例。进一步将筛选条件设定为差异表达水平超过2倍(log2FC绝对值≥1)且P<0.01,从上述数据集中可得到60余个差异表达显著的基因(表 2)。
|
|
表 2 差异表达显著的前61个基因 Table 2 Top 61 differentially expressed genes (DEGs) |
2. GO基因功能富集分析:对差异表达基因进行GO功能富集分析,结果表明,差异基因在细胞组成方面主要富集于细胞膜、外泌体、跨膜区域、细胞核质等。生物学过程方面主要参与应激反应、免疫应答、糖代谢和炎症反应等;在分子功能方面,差异基因主要富集于酶调节活性、酶抑制剂活性、内肽酶抑制剂活性、内肽酶调节活性和肽酶抑制剂活性等。
3. 信号通路的富集分析:使用HALLMARK基因集对差异表达的基因所参与的信号通路富集分析表明,差异信号通路主要富集于氧化磷酸化、未折叠蛋白应答、脂质代谢、Myc通路、mTORc1通路、糖酵解过程、P53通路、乏氧调节、补体级联反应、TNFα/NF-κB信号通路等信号通路,见图 1。
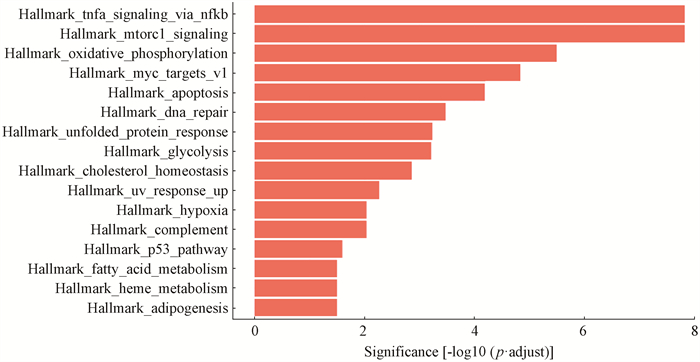
|
图 1 差异表达基因富集分析 Figure 1 Gene set enrichment analysis of differentially expressed genes |
4. DO分析:进一步对差异显著的基因进行DO分析,以识别与差异表达基因有关联的疾病。结果表明,差异基因在非小细胞肺癌、白血病等多种恶性肿瘤中富集(图 2)。这表明所筛选的差异基因在调控恶性肿瘤的发生及发展过程中发挥重要的作用。
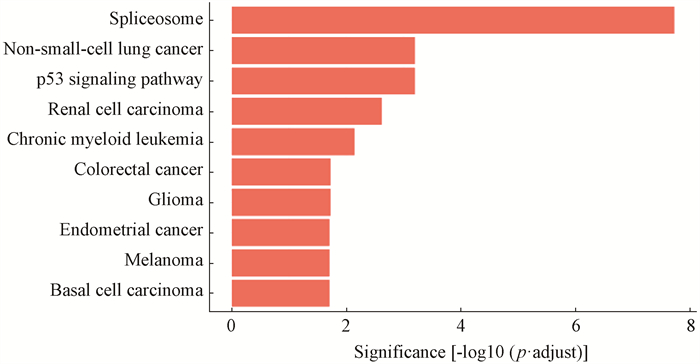
|
图 2 差异表达基因的DO分析 Figure 2 DO analysis of differentially expressed genes |
5. 差异基因的PPI分析及Hub基因筛选:应用STRING数据库对61个显著差异基因所编码的蛋白进行PPI分析,结果表明,部分差异基因间存在广泛的蛋白间相互作用(图 3)。进一步采用Cytoscape将蛋白网络可视化,应用cytoHubba插件根据节点数筛选出前5个Hub基因,分别为HSPA5、SOD2、UCP1、ACSL3、SCD,颜色越红代表节点数越多(图 3)。
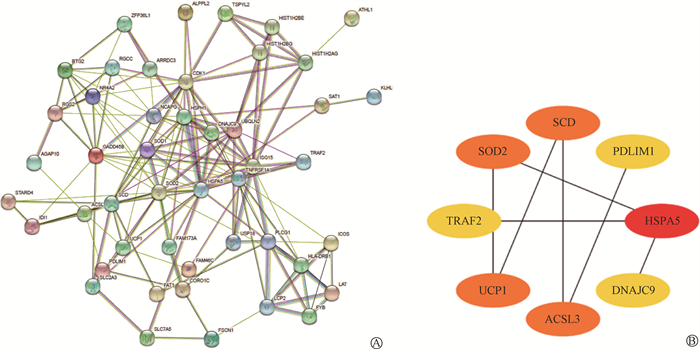
|
注:使用最大集团中心度(MCC) 算法从PPI网络中识别Hub基因。红色节点代表具有高MCC得分的基因,而黄色节点代表具有低MCC得分的基因 图 3 差异基因PPI网络分析和Hub基因的筛选 A. 差异基因的PPI网络分析;B. Hub基因的筛选 Figure 3 PPI network analysis of differentially expressed genes (DEGs) and the screening of Hub genes A. PPI network analysis of DEGs; B. Screening of Hub genes |
6. Hub基因在急性T淋巴细胞白血病中的表达及临床意义:采用TCGA和GTEx数据库验证所筛选出的Hub基因在正常组织和急性T淋巴细胞白血病中的表达情况。结果显示,5个Hub基因中,HSPA5、ACSL3及SCD在在急性T淋巴细胞白血病中平均表达水平均低于正常组织(图 4);在不同亚型的急性T淋巴细胞白血病患者中,合并TRD/LMO2融合基因的患者SCD及HSPA5表达水平均高于未合并TRD/LMO2融合基因的患者(图 4),其余Hub基因无明显差异(图 4)。因此,可以考虑以SCD及HSPA5作为分层因素,根据其表达量对急性T淋巴细胞白血病患者实施个体化精准治疗。
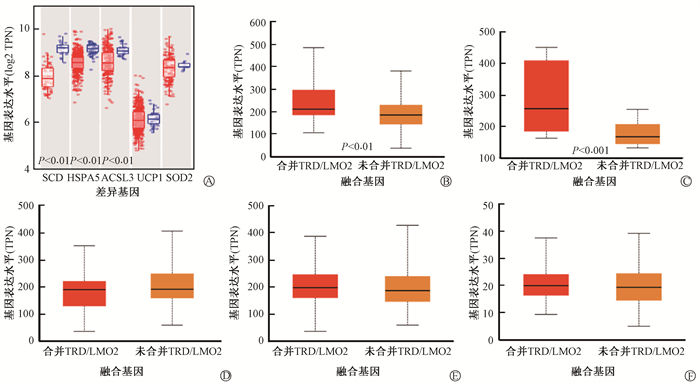
|
图 4 差异基因表达水平分析 A.差异基因在正常组织(蓝色)及急性T淋巴细胞白血病(红色)中的表达水平;B-F. Hub基因在合并TRD/LMO2融合基因与未合并TRD/LMO2融合基因的急性T淋巴细胞白血病中的表达水平验证B. HSPA5; C. SCD; D. ACSL3; E. SOD2; F. UCP1 Figure 4 The analysis of differential expressed genes (DEGs) A. The analysis of DEGs in acute T lymphoblastic leukemia (red) and normal tissues (blue); B-F. Verification of Hub gene expressions in acute T lymphoblastic leukemia with or without TRD/LMO2-fusion B. HSPA5; C. SCD; D. ACSL3; E. SOD2; F. UCP1 |
讨论
本研究通过生物信息学分析GSE100718芯片数据,筛选出调控FLASH放疗敏感性的差异基因。其中,通路富集分析发现差异基因主要富集于氧化磷酸化、未折叠蛋白应答等信号通路。在未折叠蛋白应答通路中发挥核心功能的HSPA5基因及脂质代谢中的关键基因SCD差异显著。进一步验证显示,在合并TRD/LMO2融合基因的急性T淋巴细胞白血病中,HSPA5及SCD显著高表达。因此,可考虑根据HSPA5、SCD表达水平进行患者分层,实施个体化精准放疗。
HSPA5基因编码重链结合蛋白(BIP),BIP为分子伴侣蛋白,主要定位于内质网,属于热休克蛋白70家族[5]。内质网是生物体内蛋白质合成、加工、运输、糖基化等的场所,参与调控氧化应激反应、钙离子平衡等,在维持细胞蛋白质稳态中发挥重要作用[6]。相对于机体正常细胞,肿瘤细胞代谢活跃,对蛋白质合成与加工需求极高,因此,肿瘤细胞内质网功能更为强大[7]。细胞在受到各种内外因素如氧化应激、炎症、乏氧、药物、放疗等的刺激下,细胞内蛋白质稳态受到影响,诱发内质网应激[8]。当细胞发生内质网应激时,启动未折叠蛋白应答通路和内质网相关蛋白质降解途径,HSPA5表达水平升高,以促进蛋白质折叠和错误折叠蛋白的降解,缓解内质网应激并恢复细胞内蛋白稳态。但若处于持续高强度的内质网应激条件下,则CCAAT增强子结合蛋白同源蛋白(CHOP)高表达,启动凋亡信号通路,诱导细胞凋亡[9]。内质网应激已成为肿瘤的特征之一,参与调控肿瘤的侵袭、转移、血管生成、免疫应答和放化疗抵抗[10]。HSPA5通过未折叠蛋白应答通路调控常规剂量率放疗敏感性,其中,HSPA5高表达肿瘤对常规剂量率放疗不敏感,恶性程度高[11]。在本研究中,FLASH放疗与常规剂量率放疗组织基因富集分析表明,未折叠蛋白应答通路在调控放疗敏感性中发挥作用,HSPA5低表达组对常规剂量率放疗敏感;值得注意的是,HSPA5高表达组表现为FLASH放疗敏感。因此,有可能根据HSPA5表达水平,对患者分层,分别实施个体化精准治疗。
尽管正常细胞依赖外源性脂质来保持细胞器功能和维持生存,但脂质代谢异常与某些肿瘤的风险增加有关;肿瘤细胞通过调节脂肪酸合成与分解来改变代谢途径以满足自身的能量需求[12]。SCD基因调控内质网酶硬脂酰辅酶A去饱和酶的生成,该酶负责催化饱和脂肪酸(棕榈酸和硬脂酸)转化为单不饱和脂肪酸(油酸和棕榈油酸)[13]。油酸在肿瘤细胞淋巴转移中发挥保护性作用[14]。SCD在多种恶性肿瘤中高表达,并参与维持肿瘤细胞增殖和癌症干细胞特性[15]。卵巢癌细胞系和小鼠原位异种移植模型中,抑制SCD可诱导脂质过氧化和肿瘤细胞死亡;过表达SCD及给予外源性棕榈油酸或油酸可保护肿瘤细胞免于铁死亡[16]。既往研究显示,SCD过表达与常规放疗抵抗相关,具体机制为,SCD调控脂质代谢,通过合成单不饱和脂肪酸减轻放疗诱导的脂质过氧化,减轻肿瘤细胞铁死亡[17]。本研究提示,SCD高表达者对超高剂量率放疗敏感,这为患者的分层治疗提供了新思路。
放疗除常用于实体瘤的治疗,也在血液系统肿瘤中发挥作用。临床工作中,全身放疗或全骨髓照射常用于异基因自体干细胞移植前的清髓[18-19]。FLASH放疗已在电子、光子和质子中得以实现,FLASH效应已在多种正常组织和动物模型中观察到[20-22]。Chabi等[4]报道,相对于常规剂量率放疗,FLASH放疗可以有效地减轻造血干细胞损伤;而且,基因表达谱分析提示FLASH放疗可能适合于恶性程度高、常规剂量率放疗不敏感的患者。肿瘤细胞乏氧微环境是包括常规剂量率放疗在内抗肿瘤治疗抵抗的重要原因之一[23-24]。本研究中,基因富集分析显示氧化磷酸化、未折叠蛋白应答、脂质代谢、myc通路、mtorc1通路、糖酵解过程、p53通路、补体级联反应等信号通路与FLASH放疗敏感性相关。而乏氧会影响氧化磷酸化、未折叠蛋白应答、脂质代谢、Myc通路、mTORc1通路、糖酵解过程、P53通路、补体级联反应通路的调控[25-28]。此外,氧消耗及其相关学说表明乏氧,也是诱发FLASH效应的原因之一[1]。基于上述证据,FLASH放疗的敏感性有可能与乏氧相关,需进一步研究来证实该推断。
全身放疗或全骨髓照射主要适用于急性淋巴细胞白血病,其次为急性髓系白血病和慢性髓系白血病[18-19]。急性T淋巴细胞白血病是一种未成熟T细胞的恶性克隆性疾病,该恶性肿瘤具有高侵袭性和复发率[29]。其中,t(11;14)(p13;q11)易位可导致TRD/LMO2融合,占急性T淋巴细胞白血病发病率为5%~20%[30]。尽管合并TRD/LMO2融合基因的患者预后相对较好,但有研究认为该融合基因与造血干细胞移植后较早复发相关[31]。依据基因表达谱,对急性白血病患者进行分层,有利于实施精准放疗,以提高肿瘤控制率和耐受性。在本研究中,基于生物信息学分析揭示HSPA5、SCD等可作为急性T淋巴细胞白血病患者放疗的分层因素,高表达患者对FLASH放疗敏感,低表达患者则对常规放疗敏感。进一步分析显示,在合并TRD/LMO2融合的急性T淋巴细胞白血病患者中,HSPA5和SCD高表达,因此,此亚型患者可考虑采用FLASH放疗。FLASH放疗不仅可减轻治疗相关不良反应,而且可提高治疗效率,有可能为该亚型疾病带来潜在益处。
综上所述,本研究通过对GEO数据库中和FLASH放疗有关芯片数据进行生物信息学分析,筛选出与FLASH放疗敏感性相关的差异表达基因及相应的信号通路。其中,未折叠蛋白通路的关键基因HSPA5及脂质代谢过程的重要基因SCD,参与调控FLASH放疗敏感性。在合并TRD/LMO2融合基因的急性T淋巴细胞白血病中,HSPA5及SCD表达差异显著,FLASH放疗较常规放疗可能更适合该亚型患者群体。基因表达谱可用于指导肿瘤患者分层,从而实施个体化精准放疗。
利益冲突 所有作者宣称没有任何利益冲突,未接受任何不当的职务或财务利益
志谢 感谢瑞士洛桑大学附属医院放疗科全体成员对本文的指导与帮助,感谢瑞士洛桑大学生物医学系的支持,感谢郑州大学附属肿瘤医院血液科孙艳燕、刘丽娜与河南省人民医院血液科时明月对本研究的建议
作者贡献声明 罗辉、王刘祥负责数据搜集整理与撰写;Ron Leavitt、孙亚楠、宋帅、王晓辉、毛荣虎、马蕾杰、雷宏昌负责部分文献的搜集与整理,协助论文撰写;葛红指导论文命题、思路、结构及修订
| [1] |
罗辉, 闫明阳, 贾雪超, 等. 超高剂量率放疗在恶性肿瘤中的应用[J]. 中华放射医学与防护杂志, 2021, 41(8): 636-640. Luo H, Yan MY, Jia XC, et al. Application of FLASH radiotherapy with an ultra-high dose rate in malignant tumor[J]. Chin J Radiol Med Prot, 2021, 41(8): 636-640. DOI:10.3760/cma.j.issn.0254-5098.2021.08.013 |
| [2] |
Bourhis J, Sozzi WJ, Jorge PG, et al. Treatment of a first patient with FLASH-radiotherapy[J]. Radiother Oncol, 2019, 139: 18-22. DOI:10.1016/j.radonc.2019.06.019 |
| [3] |
Armstrong F, Brunet de la Grange P, Gerby B, et al. NOTCH is a key regulator of human T-cell acute leukemia initiating cell activity[J]. Blood, 2009, 113(8): 1730-1740. DOI:10.1182/blood-2008-02-138172 |
| [4] |
Chabi S, To T, Leavitt R, et al. Ultra-high-dose-rate FLASH and conventional-dose-rate irradiation differentially affect human acute lymphoblastic leukemia and normal hematopoiesis[J]. Int J Radiat Oncol Biol Phys, 2021, 109(3): 819-829. DOI:10.1016/j.ijrobp.2020.10.012 |
| [5] |
Elfiky AA, Baghdady AM, Ali SA, et al. GRP78 targeting: Hitting two birds with a stone[J]. Life Sci, 2020, 260: 118317. DOI:10.1016/j.lfs.2020.118317 |
| [6] |
De Leonibus C, Cinque L, Settembre C. Emerging lysosomal pathways for quality control at the endoplasmic reticulum[J]. FEBS Lett, 2019, 593(17): 2319-2329. DOI:10.1002/1873-3468.13571 |
| [7] |
Banerjee S, Zhang W. Endoplasmic reticulum: target for next-generation cancer therapy[J]. Chembiochem, 2018, 19(22): 2341-2343. DOI:10.1002/cbic.201800461 |
| [8] |
Oakes SA, Papa FR. The role of endoplasmic reticulum stress in human pathology[J]. Annu Rev Pathol, 2015, 10: 173-194. DOI:10.1146/annurev-pathol-012513-104649 |
| [9] |
Almanza A, Carlesso A, Chintha C, et al. Endoplasmic reticulum stress signalling-from basic mechanisms to clinical applications[J]. FEBS J, 2019, 286(2): 241-278. DOI:10.1111/febs.14608 |
| [10] |
Urra H, Dufey E, Avril T, et al. Endoplasmic reticulum stress and the hallmarks of cancer[J]. Trends Cancer, 2016, 2(5): 252-262. DOI:10.1016/j.trecan.2016.03.007 |
| [11] |
Luo H, Song H, Mao R, et al. Targeting valosin-containing protein enhances the efficacy of radiation therapy in esophageal squamous cell carcinoma[J]. Cancer Sci, 2019, 110(11): 3464-3475. DOI:10.1111/cas.14184 |
| [12] |
Jin Z, Chai YD, Hu S. Fatty acid metabolism and cancer[J]. Adv Exp Med Biol, 2021, 1280: 231-241. DOI:10.1007/978-3-030-51652-9_16 |
| [13] |
Khan W, Augustine D, Rao RS, et al. Lipid metabolism in cancer: A systematic review[J]. J Carcinog, 2021, 20: 4. DOI:10.4103/jcar.JCar_15_20 |
| [14] |
Ubellacker JM, Tasdogan A, Ramesh V, et al. Lymph protects metastasizing melanoma cells from ferroptosis[J]. Nature, 2020, 585(7823): 113-118. DOI:10.1038/s41586-020-2623-z |
| [15] |
Oatman N, Dasgupta N, Arora P, et al. Mechanisms of stearoyl CoA desaturase inhibitor sensitivity and acquired resistance in cancer[J]. Sci Adv, 2021, 7(7): eabd7459. DOI:10.1126/sciadv.abd7459 |
| [16] |
Tesfay L, Paul BT, Konstorum A, et al. Stearoyl-CoA desaturase 1 protects ovarian cancer cells from ferroptotic cell death[J]. Cancer Res, 2019, 79(20): 5355-5366. DOI:10.1158/0008-5472.CAN-19-0369 |
| [17] |
Lei G, Mao C, Yan Y, et al. Ferroptosis, radiotherapy, and combination therapeutic strategies[J]. Protein Cell, 2021, 12(11): 836-857. DOI:10.1007/s13238-021-00841-y |
| [18] |
Wong J, Filippi AR, Dabaja BS, et al. Total body irradiation: guidelines from the International Lymphoma Radiation Oncology Group (ILROG)[J]. Int J Radiat Oncol Biol Phys, 2018, 101(3): 521-529. DOI:10.1016/j.ijrobp.2018.04.071 |
| [19] |
Mancosu P, Cozzi L, Muren LP. Total marrow irradiation for hematopoietic malignancies using volumetric modulated arc therapy: A review of treatment planning studies[J]. Phys Imaging Radiat Oncol, 2019, 11: 47-53. DOI:10.1016/j.phro.2019.08.001 |
| [20] |
Moeckli R, Gonçalves Jorge P, Grilj V, et al. Commissioning of an ultra-high dose rate pulsed electron beam medical LINAC for FLASH RT preclinical animal experiments and future clinical human protocols[J]. Med Phys, 2021, 48(6): 3134-3142. DOI:10.1002/mp.14885 |
| [21] |
Gao F, Yang Y, Zhu H, et al. First demonstration of the FLASH effect with ultrahigh dose rate high-energy X-rays[J]. Radiother Oncol, 2022, 166: 44-50. DOI:10.1016/j.radonc.2021.11.004 |
| [22] |
Hughes JR, Parsons JL. FLASH radiotherapy: current knowledge and future insights using proton-beam therapy[J]. Int J Mol Sci, 2020, 21(18): 6492. DOI:10.3390/ijms21186492 |
| [23] |
Shen H, Cook K, Gee HE, et al. Hypoxia, metabolism, and the circadian clock: new links to overcome radiation resistance in high-grade gliomas[J]. J Exp Clin Cancer Res, 2020, 39(1): 129. DOI:10.1186/s13046-020-01639-2 |
| [24] |
Kumar B, Adebayo AK, Prasad M, et al. Tumor collection/processing under physioxia uncovers highly relevant signaling networks and drug sensitivity[J]. Sci Adv, 2022, 8(2): eabh3375. DOI:10.1126/sciadv.abh3375 |
| [25] |
Chen PS, Chiu WT, Hsu PL, et al. Pathophysiological implications of hypoxia in human diseases[J]. J Biomed Sci, 2020, 27(1): 63. DOI:10.1186/s12929-020-00658-7 |
| [26] |
Hernández-Reséndiz I, Gallardo-Pérez JC, López-Macay A, et al. Mutant p53(R248Q) downregulates oxidative phosphorylation and upregulates glycolysis under normoxia and hypoxia in human cervix cancer cells[J]. J Cell Physiol, 2019, 234(5): 5524-5536. DOI:10.1002/jcp.27354 |
| [27] |
Bartoszewska S, Collawn JF. Unfolded protein response (UPR) integrated signaling networks determine cell fate during hypoxia[J]. Cell Mol Biol Lett, 2020, 25: 18. DOI:10.1186/s11658-020-00212-1 |
| [28] |
Zhang C, Liu J, Wang J, et al. The interplay between tumor suppressor p53 and hypoxia signaling pathways in cancer[J]. Front Cell Dev Biol, 2021, 9: 648808. DOI:10.3389/fcell.2021.648808 |
| [29] |
Terwilliger T, Abdul-Hay M. Acute lymphoblastic leukemia: a comprehensive review and 2017 update[J]. Blood Cancer J, 2017, 7(6): e577. DOI:10.1038/bcj.2017.53 |
| [30] |
De Bie J, Demeyer S, Alberti-Servera L, et al. Single-cell sequencing reveals the origin and the order of mutation acquisition in T-cell acute lymphoblastic leukemia[J]. Leukemia, 2018, 32(6): 1358-1369. DOI:10.1038/s41375-018-0127-8 |
| [31] |
Safavi M, Sharari AS. T cell acute lymphoblastic leukemia with t(11;14)(p13;q11) and trisomy 8[J]. Blood Res, 2021, 56(3): 128. DOI:10.5045/br.2021.2021059 |
 2022, Vol. 42
2022, Vol. 42


