诱导产生DNA双链断裂(DNA double-strand break,DSB)是电离辐射的核心生物学效应[1],早期研究显示,在噬菌体中电离辐射诱导DSB的部位不是随机的[2],提示基因组不同区域的放射敏感性是不同的,深入阐明这种差异及其机制对认识电离辐射引发的基因变异、染色体异常和细胞效应具有重要意义。近年来出现了多种以二代测序为基础的DSB检测技术,可提供DSB在全基因组范围内的定位定量信息[3],但这些实验方法存在技术门槛高、价格昂贵等问题。随着机器学习广泛应用于生物大数据分析[4],随机森林已被成功运用于内源性DSB的预测[5-6],本研究拟建立一个随机森林分类模型,通过表观遗传学信息来预测电离辐射诱导DSB的水平,并初步分析DSB在基因组中的分布规律。
材料与方法1. 数据来源:电离辐射诱导DSB的基因组注释文件(bed格式)获取于GSE134798(https://www.ncbi.nlm.nih.gov/geo/)[7],该实验中采用10 Gy γ射线照射乳腺癌细胞系MCF-7,照射后立即固定细胞,采用原位标记和测序(breaks labeling in situ and sequencing,BLISS)方法[8]测定DSB在全基因组上的分布。MCF-7细胞的8种表观遗传学ChIP-seq文件下载于ENCODE数据库[9],以GRCh38为参考基因组序列。数据信息如表 1所示。
|
|
表 1 数据来源 Table 1 Data source |
2. 数据预处理:采用UCSC tools(http://hgdownload.cse.ucsc.edu/admin/exe/)先将DSB的基因组注释文件转化为bigWig文件,再将GRCh38参考基因组分解为50 kb的片段,共61 751个片段,最后计算每个片段内的电离辐射诱导DSB的比例。采用同样方法计算50 kb片段中各个表观遗传学特征的数值。对不平衡数据使用imblearn包[10]进行过采样。
3. 随机森林模型:将电离辐射诱导DSB和表观遗传学数据集随机分入训练集和测试集,采用scikit-learn包[11]中的RandomForestClassifier模块构建分类预测模型,决策树的数量定为100,其余参数使用默认值。使用SciPy包计算混淆矩阵、真阳性率和假阳性率,用Matplotlib包[12]绘制受试者操作特征(receiver operating characteristic,ROC)曲线,并计算ROC曲线下面积。
4. 基因组表观遗传学的特征重要性:随机森林模型重复训练5次,以基尼指数作为评价指标来衡量各个表观遗传学的特征重要性。采用deepTools工具[13]中的computeMatrix命令计算DSB不同水平区域的表观遗传学特征富集值,随后采用plotProfile命令绘制50 kb的基因组片段的富集图。
5. 统计学处理:使用SciPy 1.4.0包进行正态分布检验,符合正态分布即采用两独立样本t检验。P < 0.05为差异有统计学意义。
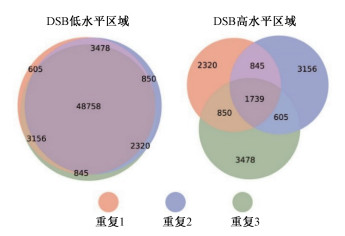
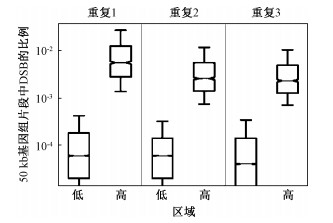
结果1. DSB分类数据的生成:电离辐射诱导DSB的全基因组数据来自3次重复的BLISS实验,原始数据为DSB在基因组的位置信息,首先将位置信息转化为50 kb基因组片段上DSB的数量比例,再将DSB比例低于平均值的片段定义为DSB低水平区域,高于平均值的片段定义为DSB高水平区域,并绘制图 1。韦恩图显示3次实验的共有区域,为了得到相对可靠的DSB数据,选取了3次重复中共有的DSB低水平区域(n=48 758)和高水平区域(n=1 739)进行随机森林模型的构建,选取的区域占总基因组的81.78%。图 2显示了选取的DSB区域在3次重复实验中的DSB数量比例,两者差异具有统计学意义(t重复3次=5.47、4.41、5.38,P < 0.05)。最后将DSB低、高水平区域的分类标记设定为0和1。
|
图 1 3次实验获得的50 kb基因组片段上电离辐射诱导DSB低、高水平区域的韦恩图 Figure 1 Venn diagram of low and high regions of ionizing radiation-induced DSB in 50 kb genomic fragments obtained from three independent experiments |
|
图 2 选取的DSB低、高水平区域在3次实验中DSB比例的箱式图 Figure 2 Boxplot of the DSB ratio of the chosen low and high regions in three independent experiments |
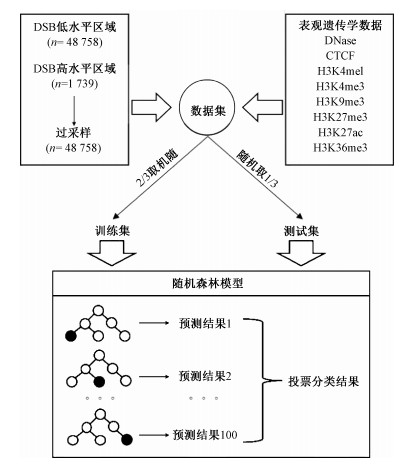
2. 构建分类预测电离辐射诱导DSB的随机森林模型:构建流程示意图如图 3所示,DSB低水平区域样本数量明显高于DSB高水平区域,为消除数据不平衡的影响,将DSB高水平区域进行过采样,使其样本数量和低水平区域一致。随机将数据集的2/3列为训练集,1/3列为测试集,将训练集输入随机森林分类模型中进行训练学习。
|
图 3 随机森林分类模型的构建流程 Figure 3 Chart of the construction of random forest classification model |
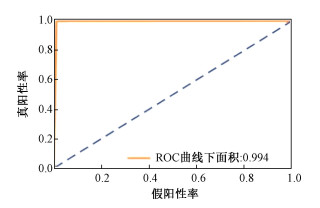
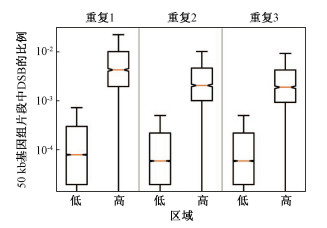
3. 随机森林分类模型在测试集上的表现:模型完成训练后对测试集进行预测,预测结果准确率为99.4%、精准率为98.9%、召回率为99.9%。ROC曲线如图 4所示,曲线下面积为0.994,接近于1,说明该模型分类预测性能优秀,能够准确识别电离辐射诱导DSB的高水平区域。接着使用该模型对全基因组50 kb片段(n=61 751)进行DSB水平的分类,分类结果在3次重复实验中的DSB分布如图 5所示,预测不同区域的DSB比例,差异具有统计学意义(t重复3次=5.26、4.27、5.20,P < 0.05)。
|
图 4 分类预测电离辐射诱导DSB的ROC曲线 Figure 4 ROC curve for the classification prediction of ionizing radiation-induced DSB |
|
图 5 预测的DSB低、高水平区域在3次实验中DSB比例的箱式图 Figure 5 Boxplot of the DSB ratio of the predictive low and high regions in three independent experiments |
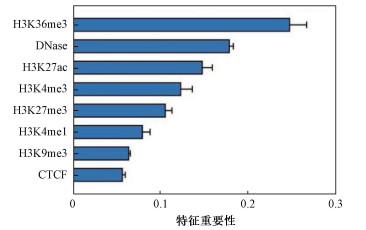
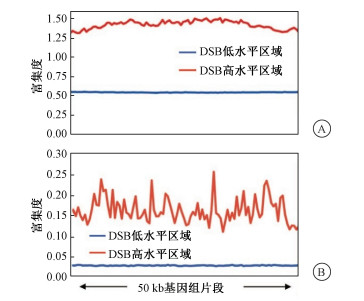
4. 特征重要性分析:随机森林建模后可返回特征重要性,从而显示表观遗传学标记对DSB分类预测的贡献度。图 6显示了8种表观遗传学标记的重要性,其中H3K36me3标记和DNase标记的贡献度最高,提示基因组中这两类标记与电离辐射诱导的DSB密切相关。为了进一步研究DSB与这两类标记的关系,分析了不同DSB水平区域的H3K36me3标记和DNase标记的富集度,如图 7所示,DSB高水平区域的两类表观遗传学标记富集程度高于DSB低水平区域,说明基因组中富含H3K36me3标记和DNase标记的区域更容易发生电离辐射诱导的DSB。
|
图 6 基因组特征重要性 Figure 6 Variable importance of genomic features |
|
图 7 DSB低、高水平区域的H3K36me3(A)和DNase(B)标记的富集程度 Figure 7 Enrichment levels of H3K36me3(A) and DNase (B) marks at the low- and high-region of DSB |
讨论
电离辐射诱导DSB是其发挥生物学效应的基础,如DSB得不到及时修复,可破坏DNA分子完整性并阻碍遗传信息的传递,引发细胞凋亡、坏死或衰老,明确DSB的发生发展规律对于辐射防护和肿瘤放射治疗具有重要意义[14]。由于真核生物基因组的复杂性和实验技术的局限性,长期以来对于DSB在基因组中的分布规律并不清楚。随着二代测序技术的迅速兴起,诞生了多种针对DSB的高通量富集测序技术,主要用于基因编辑中的脱靶效应检测,同时也可检测各种内源性和外源性因素诱导的DSB [3]。Brambilla等[7]采用了BLISS技术[8]检测了MCF-7细胞内DSB在全基因组上的分布,数据分析显示,无核小体区域在γ射线照射后容易发生DSB,而富含核小体的区域的DSB水平较低,提示基因组的状态一定程度上决定了其对电离辐射诱导DSB的敏感性。
虽然测序技术可提供全基因组水平上的DSB数据,但需要繁琐的测序文库构建和专业的生物信息学分析过程,目前只有少数几种细胞系完成了内源性DSB的检测。近年来,机器学习在生物医学大数据领域中展现了广阔的应用前景[4],随机森林作为一种稳健的机器学习方法在基因组序列和功能相关预测中得到了广泛的应用[15-16],与其他机器学习方法相比,随机森林能够处理高维度数据,不需要特征工程,抗过拟合能力强,训练速度较快,而且可以给出特征值对结果预测的重要性指标。Mourad等[5]与Ballinger等[6]采用随机森林分别构建了对内源性DSB的分类与回归预测模型,证实机器学习方法可从表观遗传学特征中抽取关于DSB分布的信息,并发现了DSB分布与基因组结构异常之间的关联。
本研究以γ射线诱导MCF-7细胞的DSB数据为对象,首先根据DSB水平的不同将50 kb基因组片段分成两类,即DSB低水平和高水平区域,随后构建了随机森林分类模型,以8种表观遗传学数据作为输入,预测性能评价表明该模型可以准确预测50 kb范围的DSB水平。理论上,只要已知相应的表观遗传学信息,该模型也适用于其他细胞系,但目前缺乏其他细胞系的电离辐射诱导DSB的测序数据,较难对该模型的通用性做出评价。如将来获取了电离辐射在对多种细胞系中诱导产生的DSB数据,就可进一步精确地分析DSB在全基因组范围的分布规律,并优化预测DSB分布的机器学习模型。
随机森林特征重要性分析显示,H3K36me3标记和DNase标记对DSB水平高低预测贡献度最高,表示这两类标记可能是决定电离辐射诱导DSB水平的关键因素,富集分析进一步显示,DSB高水平区域的H3K36me3标记和DNase标记明显高于DSB低水平区域,说明基因组中H3K36me3和DNase富集区域对电离辐射较为敏感。H3K36me3修饰是一种在真核生物中高度保守的表观遗传学修饰[17],其与RNA聚合酶Ⅱ的相互作用对mRNA生成具有重要作用,所以在H3K36me3在转录活跃的蛋白编码区富集程度高,是一种转录活跃区域的表观遗传学标记[18]。DNase-seq获取的是开放染色质的区域[19],是各类反式作用因子的结合部位,与基因表达调控密切相关[20]。H3K36me3和DNase的共定位部位一般是细胞中转录活跃的基因区域及其调控部位,本研究结果提示,基因组的这些部位对电离辐射较为敏感,细胞受照后更易发生DSB。
综上所述,本研究成功构建了预测MCF-7细胞中电离辐射诱导DSB水平的随机森林分类模型,输入8种表观遗传学数据后在测试数据集上有准确的预测性能,说明基因组中的表观遗传学修饰决定了其对电离辐射诱导DSB的敏感性。通过分析该模型初步揭示了DSB在基因组中的分布规律,电离辐射诱导DSB容易发生在H3K36me3和DNase的富集位点,表明DSB主要发生在基因组中转录活跃的部位。本研究初步揭示了真核细胞基因组不同部位的放射敏感性,为进一步阐明电离辐射对基因组DNA的损伤作用提供了新的思路。
利益冲突 无
志谢 感谢苏州大学大学生创新创业训练计划(201910285125Y)对本研究的资助
作者贡献声明 陈锦华负责模型构建和论文撰写;黄晓婷和金佳颖负责数据收集;丁伯洋、朱冉、李文艳负责数据分析和制图;刘芬菊和俞家华负责实验设计与论文修改
| [1] |
Jackson SP, Bartek J. The DNA-damage response in human biology and disease[J]. Nature, 2009, 461(7267): 1071-1078. DOI:10.1038/nature08467 |
| [2] |
刘树铮. 医学放射生物学[M]. 北京: 原子能出版社, 2006. Liu SZ. Medical radiobiology[M]. Beijing: Atomic Energy Press, 2006. |
| [3] |
Oster S, Aqeilan RI. Programmed DNA damage and physiological DSBs: mapping, biological significance and perturbations in disease states[J]. Cells, 2020, 9(8): 1870. DOI:10.3390/cells9081870 |
| [4] |
Goecks J, Jalili V, Heiser LM, et al. How machine learning will transform biomedicine[J]. Cell, 2020, 181(1): 92-101. DOI:10.1016/j.cell.2020.03.022 |
| [5] |
Mourad R, Ginalski K, Legube G, et al. Predicting double-strand DNA breaks using epigenome marks or DNA at kilobase resolution[J]. Genome Biol, 2018, 19(1): 34. DOI:10.1186/s13059-018-1411-7 |
| [6] |
Ballinger TJ, BAM B, Mirzazadeh R, et al. Modeling double strand break susceptibility to interrogate structural variation in cancer[J]. Genome Biol, 2019, 20(1): 28. DOI:10.1186/s13059-019-1635-1 |
| [7] |
Brambilla F, Garcia-Manteiga JM, Monteleone E, et al. Nucleosomes effectively shield DNA from radiation damage in living cells[J]. Nucleic Acids Res, 2020, 48(16): 8993-9006. DOI:10.1093/nar/gkaa613 |
| [8] |
Yan WX, Mirzazadeh R, Garnerone S, et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks[J]. Nat Commun, 2017, 8: 15058. DOI:10.1038/ncomms15058 |
| [9] |
Davis CA, Hitz BC, Sloan CA, et al. The encyclopedia of DNA elements (ENCODE): data portal update[J]. Nucleic Acids Res, 2018, 46(D1): D794-D801. DOI:10.1093/nar/gkx1081 |
| [10] |
Lemaȋtre G, Nogueira F, Aridas CK. Imbalanced-learn: a python toolbox to tackle the curse of imbalanced datasets in machine learning[J]. J Mach Learn Res, 2017, 18(17): 1-5. |
| [11] |
Pedregosa F, Varoquaux G, Gramfort A, et al. Scikit-learn: Machine learning in python[J]. J Mach Learn Res, 2011, 12: 2825-2830. |
| [12] |
Hunter JD. Matplotlib: A 2D graphics environment[J]. Comput Sci Eng, 2007, 9(3): 90-95. DOI:10.1109/MCSE.2007.55 |
| [13] |
Ramírez F, Ryan DP, Grüning B, et al. deepTools2:a next generation web server for deep-sequencing data analysis[J]. Nucleic Acids Res, 2016, 44(W1): W160-165. DOI:10.1093/nar/gkw257 |
| [14] |
Toulany M. Targeting DNA double-strand break repair pathways to improve radiotherapy response[J]. Genes (Basel), 2019, 10(1): 25. DOI:10.3390/genes10010025 |
| [15] |
Polak P, Karlić R, Koren A, et al. Cell-of-origin chromatin organization shapes the mutational landscape of cancer[J]. Nature, 2015, 518(7539): 360-364. DOI:10.1038/nature14221 |
| [16] |
Whalen S, Truty RM, Pollard KS. Enhancer-promoter interactions are encoded by complex genomic signatures on looping chromatin[J]. Nat Genet, 2016, 48(5): 488-496. DOI:10.1038/ng.3539 |
| [17] |
Barski A, Cuddapah S, Cui K, et al. High-resolution profiling of histone methylations in the human genome[J]. Cell, 2007, 129(4): 823-837. DOI:10.1016/j.cell.2007.05.009 |
| [18] |
Li J, Moazed D, Gygi SP. Association of the histone methyltransferase Set2 with RNA polymerase Ⅱplays a role in transcription elongation[J]. J Biol Chem, 2002, 277(51): 49383-49388. DOI:10.1074/jbc.M209294200 |
| [19] |
Liu Y, Fu L, Kaufmann K, et al. A practical guide for DNase-seq data analysis: from data management to common applications[J]. Brief Bioinform, 2019, 20(5): 1865-1877. DOI:10.1093/bib/bby057 |
| [20] |
Cockerill PN. Structure and function of active chromatin and DNase I hypersensitive sites[J]. FEBS J, 2011, 278(13): 2182-2210. DOI:10.1111/j.1742-4658.2011.08128.x |
 2021, Vol. 41
2021, Vol. 41


