放射治疗是口腔癌的重要治疗方式,但放射耐受会导致患者治疗失败。已有研究发现,阴阳1(yin yang 1,YY1)可通过调控PTEN/Rad5信号通路诱导射线抗性[1];CD47-SIRPα能通过调节上皮-间质转化和肿瘤干细胞的表型,使癌细胞对射线产生耐受[2]。但该类研究均不是从整体水平上探寻放射抵抗相关分子。有研究者通过分析已有的一份基因表达汇编(gene expression omnibus,GEO)数据,发现CD225可通过调控pSTAT/p21信号通路增加口腔癌细胞的放射抗性[3]。本研究将利用慢病毒shRNA文库结合二代测序技术,从人全部激酶基因中筛选影响放射抵抗的基因,探究射线对癌细胞的作用机制,为口腔鳞癌的临床治疗决策提供理论基础。
材料与方法1. 试剂与材料:Dharmacon GIPZ蛋白激酶shRNA慢病毒文库(RHS6078)购自美国GE Healthcare公司,人舌鳞癌细胞系Cal-27购自中国科学院上海细胞库,DMEM培养基购自美国Hyclone公司,胎牛血清购自以色列BI公司,嘌呤霉素(puromycin)、高保真DNA聚合酶(Phusion Hot Start Ⅱ DNA Polymerase)购自美国Thermo公司,聚凝胺(polybrene)购自美国Sigma公司,基因组DNA提取试剂盒和凝胶回收试剂盒购自德国QIAGEN公司,X-tremeGENE HP DNA转染试剂购自德国Roche公司。人HEK293T细胞、pCMV-DR8.9-EGFP质粒和pCMV-VSV-G质粒为本实验室自有。
2. 细胞培养:舌鳞癌Cal-27(用于基因筛选)和HEK293T细胞(用于大量扩增慢病毒颗粒)均置37℃、5% CO2、饱和湿度培养箱中,用含10%胎牛血清和1%青霉素-链霉素的DMEM进行培养,每次铺板实验均使用对数生长期细胞。
3. 慢病毒制备及感染率确定:在HEK293T细胞中进行慢病毒包装,3种质粒质量比为DR8.9 ∶VSV-G ∶GIPZ-shRNA=9 ∶1 ∶10,以X-tremeGene HP为转染试剂。收集病毒后,按10倍梯度稀释感染Cal-27细胞,以聚凝胺(polybrene)为感染增强剂,24 h后,流式检测绿色荧光细胞比例,并计算病毒含量。结果发现,感染率控制在30%时可基本保证1个细胞只接受1个病毒颗粒,避免发生单个细胞的多基因敲减。
4. 细胞分组及照射:Cal-27细胞感染病毒24 h后,以嘌呤霉素作用48 h去除未成功感染细胞后,更换为新鲜培养基,对细胞进行辐照。按照射剂量分为0、10和15 cGy组。其中,10和15 cGy组实验重复2次。在差异基因热图分析中,重复组用10 cGy-1、10 cGy-2、15 cGy-1、15 cGy-2组表示。采用美国Varian 600CD直线加速器,6 MV光子射线,源靶距(SSD)为97.85 cm,剂量率为600 cGy/min,单次照射。15 cm培养盘和6孔板的射野范围分别为20 cm ×20 cm和14 cm × 10 cm,底部垫1.5 cm厚组织等效物,照射液面中间平面,各培养盘(孔板)单独照射。
5. 细胞存活率检测:Cal-27细胞感染慢病毒并经嘌呤霉素筛选后,以不同剂量射线(0、5、10、15、20、25、30、35 cGy)处理。照射后培养72 h,消化细胞并计数。每个照射剂量设2个复孔,实验重复2次。以0 cGy照射组存活率为100%进行计算,并绘制细胞存活曲线。
6. 基因组DNA提取及目的序列扩增:Cal-27细胞辐照后继续培养72 h,去除培养基,预冷磷酸盐缓冲液(PBS)洗2次,然后按照QIAGEN试剂盒抽提基因组DNA,经DM2000测定浓度后,取相同量进行PCR扩增目的片段,反应体系参照慢病毒文库说明书。引物序列(5′→3′)如下:正向引物:A A T G A T A C G G C G A C C A C C G A G A T C T A C A C C G C A G C A A C A G A T G G A A G G C C,反向引物:C A A G C A G A A G A C G G C A T A C G A G A T A T T T A T A C C A T T T T A A T T C A G C。
7. PCR产物纯化及送样:PCR产物先以醋酸钠法浓缩,然后经琼脂糖凝胶电泳,切割目的条带后以QIAGEN胶回收试剂盒纯化,再经凝胶电泳检测,各组无杂带且浓度基本一致后,送样测序。
8. 建库及测序:经Qubit 3.0质量检测合格后,以PCR产物建库,文库片段大小为660 bp,以Illumina Hiseq 4000平台的SE 50进行测序,使用慢病毒文库提供引物进行测序,读长49 bp。数据质量控制标准为:每组样本测得的有效序列数>10×106个(clean reads>10 M),碱基识别正确率>98.5% (Q20>98.5%)。去除污染、接头序列后的数据通过微生物重测序分析软件与人类基因组hg19(GRCh37)为参考序列进行比对,生成初步分析数据。
9. 数据分析:细胞存活曲线和目的shRNA覆盖率采用GraphPad Prism 7软件进行分析。测序数据的生物信息学分析采用R软件(version 2.15.2)进行,分析方法参照Dharmacon生物信息学分析技术说明书。
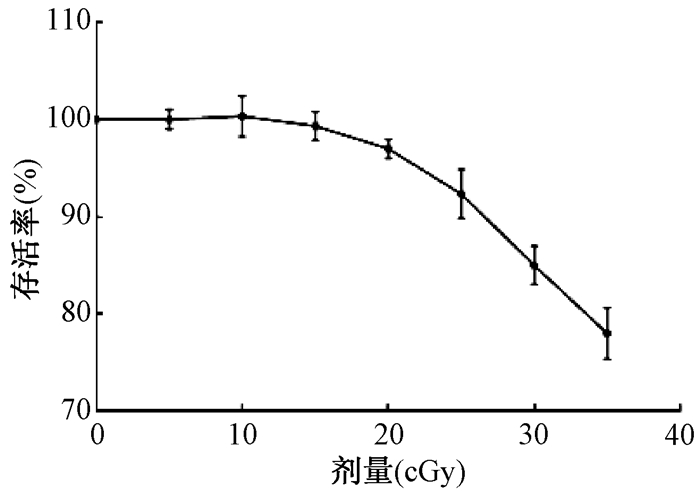
结果1. 照射剂量选择:结果如图 1所示,由于细胞本身先经shRNA敲减处理,照射剂量过大,受影响基因过多,会难以筛选到合适的基因,因此选择对细胞总体存活率影响较小的10和15 cGy进行照射。
|
图 1 不同剂量照射下Cal-27细胞存活曲线 Figure 1 Cell survival curve of Cal-27 cells under different doses of irradiation |
2. shRNA整合情况分析:GIPZ文库中共含709个激酶基因,每个基因有3 ~ 8个针对不同位点设计的shRNA敲减序列,合计4 675个不同序列。在所有样本组中,均能检测到94%以上的shRNA序列,覆盖全部激酶基因,表明shRNA敲减文库构建成功。
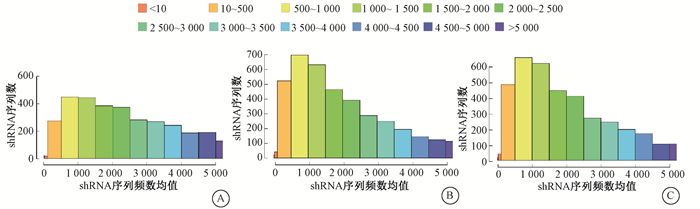
3. shRNA序列数(read count)分布情况检测:不同shRNA敲减基因后会导致细胞对射线敏感性产生差异,继续培养过程中,敏感细胞的丰度会不断降低,能检测到的对应shRNA read count也会降低。由Read count频数分布图(图 2)可知,各剂量组都呈偏态分布,且Read count在1 000左右的shRNA分布最多。与照射组相比,0 cGy组的shRNA频数分布比较均匀,可能是由于一些shRNA敲减本身就会影响细胞生长速度。此外,由于各组接受的照射剂量差别不大,因此Read count分布情况基本一致。
|
注:二代测序中,每个测序反应得到的序列称为一个“read”,测序得到的原始图像数据文件经碱基识别分析转化为测序序列,称之为Rawreads或Rawdata,本图中统计的是cleanreads,即将Rawreads去除带接头的、低质量的reads后得到的。统计某一Read在整个测序中出现的数量,即Readcount,该数值与RNA丰度成正比。横坐标shRNA序列频数分布(Read counts distribution)表示每个shRNA reads数的频数分布,纵坐标shRNA序列数(shRNAnumbers)表示reads数在对应频数区间的shRNA数量 图 2 shRNA文库敲减后的Cal-27细胞经不同剂量放射线处理后检测到的shRNA序列频数分布图A. 0 cGy组;B. 10 cGy组;C. 15 cGy组 Figure 2 The distribution map of shRNA sequences detected in Cal-27 cells with shRNA library knockdown after treatment with different doses of radiation A. 0 cGy group; B. 10 cGy group; C.15 cGy group |
4. 数据均一化处理:进行基因差异表达分析前,先将每组的read count数进行均一化处理,各组中基因的shRNA被测定的平均read count数基本一致,表明各组测序深度基本一致,可进行差异分析。
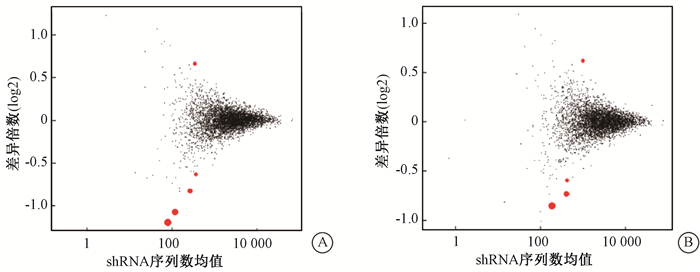
5. 差异基因分布分析:将各照射剂量组分别与0 cGy组进行差异基因分析(图 3),红色圆点为差异基因(cut值为P < 0.005及Fold Change值>1.5或 < 0.667)。位于黑色主集落下方红色圆点代表的基因是照射后丰度减少的基因,即敲减后会导致细胞对射线的敏感性增加,即为放疗抵抗基因。反之,上方红色圆点为放射敏感基因。由图 3可见,在10和15 cGy照射组分别发现5个和4个显著差异基因。
|
A.10 cGy组与0 cGy组间的差异;B.15 cGy组与0 cGy组间的差异 图 3 shRNA文库敲减后的Cal-27细胞不同剂量照射组组间基因差异倍数分布图 A.The difference between 10 cGy group and the 0 cGy group; B. The difference between 15 cGy group and 0 cGy group Figure 3 Distribution of the fold change of differential expressed genes in Cal-27 cells with shRNA library knockdown |
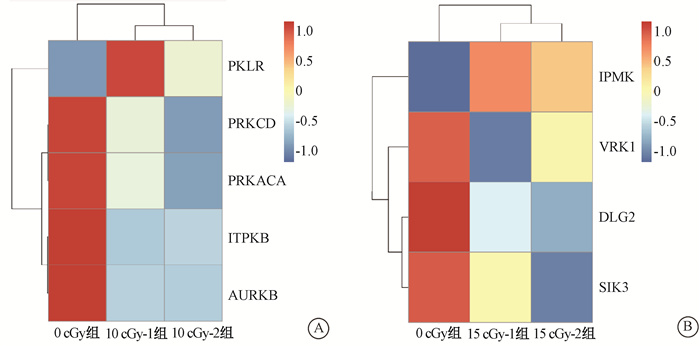
6. 差异基因热图分析:上述显著差异基因中,在经shRNA敲减后,与0 cGy组相比,10 cGy组对放射线抗性增加的基因为丙酮酸激酶1(pyruvate kinase isozymes L/R,即pyruvate kinase 1,PKLR),抗性降低的为PRKC3、PRKACA、肌醇-1, 4, 5-三磷酸3-激酶B(inositol-1, 4, 5-trisphosphate 3-kinase-B,ITPKB)和极光激酶B(aurora kinase B,AURKB);15 cGy组敲减后抗性增加的为肌醇多磷酸盐多激酶(inositol polyphosphate multikinase,IPMK),减少的为VRK1、碟状大同系物2(discs large homolog 2,DLG2)和SIK3。为确认上述差异基因在重复组中变化是否一致,进行了热图分析,结果显示,在重复组中有相同变化趋势的基因为PKLR、IPMK、ITPKB、AURKB和DLG2(图 4)。
|
A.0 cGy组与10 cGy重复组之间的差异;B. 0 cGy组与15 cGy重复组之间的差异 图 4 shRNA文库敲减后的Cal-27细胞不同剂量照射组组间差异基因分布热图 A. The differences between 0 cGy group and 10 cGy groups; B. The differences between 0 cGy group and 15 cGy groups Figure 4 Heat map of differential expressed gene in Cal-27 cells after shRNA library knockdown |
7. 信号通路分析:通过对5个差异基因进行信号通路分析,发现放射敏感基因(PKLR、IPMK)主要调控代谢途径和细胞膜的稳定性;而抗性基因(ITPKB、AURKB和DLG2)主要调控细胞周期和凋亡途径。
讨论放射治疗是癌症治疗的重要手段,但是,对于放疗生物学效应的分子机制研究不足一直是阻碍放疗进步的重要原因之一。高通量筛选技术可从整体水平快速筛选出特定目标分子,shRNA慢病毒文库是其中一种,将该技术与二代测序技术结合,可一次性对几百甚至上万个基因进行快速筛选。
本研究所用文库为GE Dharmacon研发的人蛋白激酶组文库,其主要工作流程为:先用包含数万个shRNA的慢病毒文库以低感染率感染目的细胞,使单个细胞基因组中只整合特定的单一shRNA;筛选出成功感染并表达shRNA的细胞,以不同筛选因素(本研究中为射线)作用后,使表达某些shRNA的细胞在细胞群中的丰度发生变化,富集或耗尽;然后对提取的基因组DNA进行二代测序,0 cGy组和各照射剂量组之间存在显著丰度变化的基因即为目的基因[4]。由于其shRNA文库及测序引物已经过优化,可以最大化减小偏倚,且shRNA靠近测序引物一侧带有22 bp可变区,其序列与目的敲减shRNA序列一一对应,可根据测序结果快速匹配目的基因。
本研究从709个激酶基因中筛选出5个可能与放射线敏感性相关的基因:其中AURKB,ITPKB和DLG2为放疗抵抗基因,经shRNA敲减后会致敏细胞,而PKLR和IPMK的功能则相反。
AURKB属于丝氨酸/苏氨酸激酶家族,主要调控有丝分裂,其高表达与鼻咽癌[5]和骨肉瘤[6]患者预后不良相关。已有研究证明,抑制AURKB的表达可抑制肺癌细胞经放射线照射后的增殖能力并诱导癌细胞凋亡[7-9],MET/AXL/FGFR抑制剂S49076也可获得同样效果[10],但受影响的信号通路尚不清楚。本研究检测到AURKB敲减后的细胞丰度在照射后减少,与文献[9-10]结果一致。
ITPKB在细胞代谢中有重要作用,其缺乏会导致严重的联合免疫缺陷[11],最初有研究发现肺癌细胞中ITPKB下调[12],因此认为是抑癌基因,在嗜铬细胞瘤[13]和结直肠癌[14]中的研究也支持这一观点。但后续研究发现,ITPKB可通过调节钙的释放和细胞骨架运动促进肺癌细胞的转移[15],其抑制剂可增强顺铂对肿瘤生长的抑制作用[16]。
DLG2属于膜相关鸟苷激酶(membrane-associated guanylate kinase,MAGUK)家族,其敲除之后,会导致培养的骨肉瘤癌细胞的3D克隆形成能力丧失,小鼠异种移植瘤生长变慢[17]。DLG2广泛参与细胞生理过程调控,与膀胱癌[18]、卵巢癌[19]和骨肉瘤[20]的发生发展密切相关,并受miR-23a调控[20],是抑癌基因。
PKLR和IPMK敲减后均会导致细胞产生放射耐受,二者分别为胞质内调节糖酵解[21]和核内磷脂代谢[22]中的关键激酶,与癌症的发生发展有着密切关系[23-24]。Bong等[25]以高剂量(0.8 Gy/min,单次4.5 Gy)或低剂量(0.7 mGy/h,累积1.7 Gy)137Cs照射小鼠胸腺,在两组中获得的显著响应基因完全不同,其中PKLR是高剂量照射组的显著响应基因。本研究使用低剂量照射也发现PKLR的响应,提示其对放射抗性的调控中存在重要作用,但是体内和体外研究可能会存在较大差异。
本实验并未在不同剂量组找到相同的耐受基因,原因一是本研究所用细胞株Cal-27生长速度较快,为满足实验周期,所以每剂量组有7盘细胞,不同培养盘中的细微差异可能在长时间培养中被放大;二是本研究中,射线的作用面是培养基中间平面,与直接照射组织相比,可能更容易引起实验误差。
由以上分析可知,慢病毒shRNA文库结合二代测序的方法适用于高通量筛选放射线敏感性相关基因,本研究中发现的5个放疗抵抗相关基因中,有2个基因与已有研究一致,其余3个作用尚不明确。由于下游调控的信号通路比较广泛,包括代谢途径、膜稳定性、细胞周期和凋亡途径,因此还需更多深入研究进行验证。
利益冲突 无
作者贡献声明 罗丽萍负责病毒制备、细胞感染、目的基因扩增和文章撰写;曹邦荣负责数据分析;马士淇负责细胞培养;任源负责细胞计数;祁国海负责细胞照射;王卫东负责实验设计及文章审校
| [1] |
Zhao L, Li R, Qiu JZ, et al. YY1-mediated PTEN dephosphorylation antagonizes IR-induced DNA repair contributing to tongue squamous cell carcinoma radiosensitization[J]. Mol Cell Probes, 2020, 53: 101577. DOI:10.1016/j.mcp.2020.101577 |
| [2] |
Pai S, Bamodu OA, Lin YK, et al. CD47-SIRPα signaling induces epithelial-mesenchymal transition and cancer stemness and links to a poor prognosis in patients with oral aquamous cell carcinoma[J]. Cells, 2019, 8(12): 1658. DOI:10.3390/cells8121658 |
| [3] |
Yang J, Li L, Xi Y, et al. Combination of IFITM1 knockdown and radiotherapy inhibits the growth of oral cancer[J]. Cancer Sci, 2018, 109(10): 3115-3128. DOI:10.1111/cas.13640 |
| [4] |
Schaefer C, Mallela N, Seggewi BJ, et al. Target discovery screens using pooled shRNA libraries and next-generation sequencing: a model workflow and analytical algorithm[J]. PLoS One, 2018, 13(1): e0191570. DOI:10.1371/journal.pone.0191570 |
| [5] |
Huang PY, Li Y, Luo DH, et al. Expression of aurora B and FOXM1 predict poor survival in patients with nasopharyngeal carcinoma[J]. Strahlenther Onkol, 2015, 191(8): 649-655. DOI:10.1007/s00066-015-0840-4 |
| [6] |
Zhao Z, Jin G, Yao K, et al. Aurora B kinase as a novel molecular target for inhibition the growth of osteosarcoma[J]. Mol Carcinog, 2019, 58(6): 1056-1067. DOI:10.1002/mc.22993 |
| [7] |
Woo JK, Kang JH, Shin D, et al. Daurinol enhances the efficacy of radiotherapy in lung cancer via suppression of aurora kinase A/B expression[J]. Mol Cancer Ther, 2015, 14(7): 1693-1704. DOI:10.1158/1535-7163.MCT-14-0960 |
| [8] |
Sak A, Stuschke M, Groneberg M, et al. Inhibiting the aurora B kinase potently suppresses repopulation during fractionated irradiation of human lung cancer cell lines[J]. Int J Radiat Oncol Biol Phys, 2012, 84(2): 492-499. DOI:10.1016/j.ijrobp.2011.12.021 |
| [9] |
Wu X, Liu W, Cao Q, et al. Inhibition of Aurora B by CCT137690 sensitizes colorectal cells to radiotherapy[J]. J Exp Clin Cancer Res, 2014, 33: 13. DOI:10.1186/1756-9966-33-13 |
| [10] |
Clémenson C, Chargari C, Liu W, et al. The MET/AXL/FGFR inhibitor S49076 impairs aurora B activity and improves the antitumor efficacy of radiotherapy[J]. Mol Cancer Ther, 2017, 16(10): 2107-2119. DOI:10.1158/1535-7163.MCT-17-0112 |
| [11] |
Almutairi A, Wallace JG, Jaber F, et al. Severe combined immunodeficiency caused by inositol-trisphosphate 3-kinase B (ITPKB) deficiency[J]. J Allergy Clin Immunol, 2020, 145(6): 1696-1699. DOI:10.1016/j.jaci.2020.01.014 |
| [12] |
Jin Y, Liu Y, Zhang J, et al. The expression of miR-375 is associated with carcinogenesis in three subtypes of lung cancer[J]. PLoS One, 2015, 10(12): e0144187. DOI:10.1371/journal.pone.0144187 |
| [13] |
Koenig S, Moreau C, Dupont G, et al. Regulation of NGF-driven neurite outgrowth by Ins(1, 4, 5)P3 kinase is specifically associated with the two isoenzymes ITPKA and ITPKB in a model of PC12 cells[J]. FEBS J, 2015, 282(13): 2553-2569. DOI:10.1111/febs.13300 |
| [14] |
Li B, Shi C, Zhao J, et al. Long noncoding RNA CCAT1 functions as a ceRNA to antagonize the effect of miR-410 on the down-regulation of ITPKB in human HCT-116 and HCT-8 cells[J]. Oncotarget, 2017, 8(54): 92855-92863. DOI:10.18632/oncotarget.21612 |
| [15] |
Bäder S, Glaubke E, Grüb S, et al. Effect of the actin- and calcium-regulating activities of ITPKB on the metastatic potential of lung cancer cells[J]. Biochem J, 2018, 475(12): 2057-2071. DOI:10.1042/BCJ20180238 |
| [16] |
Pan C, Jin L, Wang X, et al. Inositol-triphosphate 3-kinase B confers cisplatin resistance by regulating NOX4-dependent redox balance[J]. J Clin Invest, 2019, 129(6): 2431-2445. DOI:10.1172/JCI124550 |
| [17] |
Shao YW, Wood GA, Lu J, et al. Cross-species genomics identifies DLG2 as a tumor suppressor in osteosarcoma[J]. Oncogene, 2019, 38(2): 291-298. DOI:10.1038/s41388-018-0444-4 |
| [18] |
Lee HY, Chen YJ, Chang WA, et al. Effects of epigallocatechin gallate (EGCG) on urinary bladder urothelial carcinoma-next-generation sequencing and bioinformatics approaches[J]. Medicina (Kaunas), 2019, 55(12): 768. DOI:10.3390/medicina55120768 |
| [19] |
Zhuang RJ, Bai XX, Liu W. MicroRNA-23a depletion promotes apoptosis of ovarian cancer stem cell and inhibits cell migration by targeting DLG2[J]. Cancer Biol Ther, 2019, 20(6): 897-911. DOI:10.1080/15384047.2019.1579960 |
| [20] |
Shao YW, Wood GA, Lu J, et al. Cross-species genomics identifies DLG2 as a tumor suppressor in osteosarcoma[J]. Oncogene, 2019, 38(2): 291-298. DOI:10.1038/s41388-018-0444-4 |
| [21] |
Yang Y, Zhu G, Dong B, et al. The NQO1/PKLR axis promotes lymph node metastasis and breast cancer progression by modulating glycolytic reprogramming[J]. Cancer Lett, 2019, 453: 170-183. DOI:10.1016/j.canlet.2019.03.054 |
| [22] |
Malabanan MM, Blind RD. Inositol polyphosphate multikinase (IPMK) in transcriptional regulation and nuclear inositide metabolism[J]. Biochem Soc Trans, 2016, 44(1): 279-285. DOI:10.1042/BST20150225 |
| [23] |
Fan Z, Fan K, Deng S, et al. HNF-1a promotes pancreatic cancer growth and apoptosis resistance via its target gene PKLR[J]. Acta Biochim Biophys Sin (Shanghai), 2020, 52(3): 241-250. DOI:10.1093/abbs/gmz169 |
| [24] |
Liu P, Qi X, Bian C, et al. MicroRNA-18α inhibits ovarian cancer growth via directly targeting TRIAP1 and IPMK[J]. Oncol Lett, 2017, 13(6): 4039-4046. DOI:10.3892/ol.2017.5961 |
| [25] |
Bong JJ, Kang YM, Shin SC, et al. Identification of radiation-sensitive expressed genes in the ICR and AKR/J mouse thymus[J]. Cell Biol Int, 2013, 37(5): 485-494. DOI:10.1002/cbin.10065 |
 2021, Vol. 41
2021, Vol. 41


