2. 北京诺禾致源科技股份有限公司, 北京 100015
2. Novogene Co., Ltd, Beijing 100015, China
造血系统损伤是放射损伤的重要表现,骨髓的造血功能抑制是放射事故致死和放射治疗不良反应的重要原因之一[1-2]。环状RNA(circular RNA,circRNA)是一类经反向剪接、使得3′和5′末端共价结合形成的闭合环状非编码RNA分子,广泛存在于多种细胞类型中,具有结构稳定、序列保守、丰度高及细胞或组织特异性表达等特征[3-4]。研究表明,circRNAs在多种疾病的发生、发展中起重要作用,如肿瘤[5]、神经退行性疾病[6]、心血管系统疾病[7]及消化系统疾病[8]等。RNA转录后修饰近年来已成为生命科学和医学领域的研究重点和热点,其中,发生于真核信使RNA(messenger RNAs,mRNAs)及非编码RNA内部的N6-甲基腺嘌呤(N6-methyladenine,m6A)修饰被认为是最普遍和保守的RNA表观遗传修饰方式[9]。这种遗传信息的精细调控模式可灵敏应答外部刺激信号,然而,γ-射线照射如何通过circRNA的m6A修饰诱导造血系统的损伤效应目前尚未见报道。本研究旨在以受照C57BL/6 J小鼠构建γ射线诱导的造血系统损伤模型,探究4 Gy 137Cs γ射线全身照射对小鼠骨髓细胞中circRNA的m6A修饰谱的影响,为研究辐射损伤的表观遗传学分子机制和开发造血系统损伤早期阻断方法提供理论基础和实验依据。
材料与方法1. 实验动物与分组:C57BL/6 J雄性小鼠24只,6~8周龄,体重18 ~ 20 g,健康无特定病原体级,许可证号:SCXK(京)2014-0008,购于北京华阜康生物科技有限公司。饲养于中国医学科学院放射医学研究所无特定病原体级动物房屏障环境内,饲养温度18~25℃,相对湿度为40% ~ 70%。所有小鼠饲养1周适应环境后按体重随机区组法分为健康对照组(12只)和照射组(12只)。
2. 仪器与照射:137Cs γ射线照射源购自加拿大原子能有限公司(USD Autocell40)。照射组小鼠经4 Gy 137Cs γ射线全身照射,吸收剂量率为0.88 Gy/min。健康对照组小鼠在相同条件下接受0 Gy假照射。
3. 样品收集:照射后将两组小鼠脱颈处死,无菌分离小鼠双侧股骨,用1 ml注射器和预冷的生理盐水将骨髓细胞冲出。将同组中每4只小鼠的骨髓细胞混合,每组制成3份平行样品,经TRIzol裂解法提取总RNA后,送由北京诺禾致源科技股份有限公司进行MeRIP-Seq分析。本课题组前期研究结果显示,照射5 min时小鼠骨髓细胞的mRNA m6A修饰谱变化较为明显[10],circRNA的m6A修饰谱初步分析亦如此,故本课题重点呈现照射完成5 min后的circRNA m6A修饰谱变化情况。
4. MeRIP文库制备及测序:MeRIP-Seq技术是在已有实验方法[11]的基础上开发而来,方法简述如下:片段化的RNA与m6A抗体(英国Abcam公司)在免疫共沉淀缓冲液(immunoprecipitation buffer,IPP缓冲液,中国云序生物公司,150 mmol/L NaCl,0.1% NP-40,10 mmol/L Tris-HCl,pH 7.4)中4℃共孵育2 h。反应混合物进一步用protein A磁珠(美国Thermo Fisher公司)在4℃免疫沉淀2 h。然后用游离的m6A腺苷类似物(美国Sigma-Aldrich公司)洗脱磁珠上结合的RNA。洗脱的RNA进一步通过TRIzol试剂(美国Thermo Fisher公司)进行抽提。抽提纯化的RNA通过RNA建库试剂盒(美国New England Biolabs,NEB公司)建库,并在Illumina Hiseq测序仪(美国Illumina公司)上进行双端150 bp测序。
5. 数据分析:经过Illumina HiSeq 4000测序仪测序、图像分析、碱基识别和质量控制后,产生原始reads(Raw Data)。首先使用Q30进行质量控制,Q30 > 80%表示测序质量良好。然后,使用cutadapt软件(v1.9.3)[12]去接头,去低质量reads,获得高质量clean reads。使用STAR软件[13]将input文库的clean reads匹配到参考基因组(UCSC MM10)上,再使用DCC软件[14]进行circRNA的识别。使用Hisat2软件(v2.0.4)[15]将全部样本的clean reads匹配到参考基因组上。再使用MACS软件[16]识别每个样本中的甲基化峰,对每组3个生物学重复的峰进行合并,并对合并峰取交集后分析数目。
6. 差异甲基化circRNA识别:使用diffReps软件[17]进行差异甲基化位点的识别。该软件是常用的差异分析软件,分析过程采用特有的数学模型对每组各生物学重复的甲基化信息进行总体组间差异计算,不涉及对单个样本峰的计算。筛选标准为P < 10-10并且倍数筛选阈值(Fold-change)> 5。再通过自有程序筛选位于circRNA的exon上的峰,进行相应的注释。
7. CircRNA MeRIP-qPCR实验验证:对小鼠骨髓细胞RNA进行片段化处理,制备结合有RNA m6A特异性抗体的磁珠,留取适量片段化的RNA样品作为Input后,另将一部分上述样品与制备好的磁珠孵育进行免疫共沉淀,并将沉淀得到的RNA洗脱收集,用于后续qPCR实验检测。circRNA qPCR验证引物见表 1。
|
|
表 1 circRNA qPCR验证引物 Table 1 Primers of circRNAs for qPCR |
8. 差异甲基化circRNA来源基因基因本体论(gene ontology,GO)分析:基因本体论分成3个部分:分子功能(molecular function,MF)、生物过程(biological process,BP)和细胞组分(cell component,CC)。本研究利用差异甲基化circRNA的来源基因进行GO功能分析,以注释并推测这些差异甲基化基因可能的作用。P < 0.05的GO条目为差异有统计学意义。
9. 差异甲基化circRNA来源基因京都基因与基因组百科数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)分析:本研究利用差异甲基化circRNA的来源基因进行通路分析,以注释并推测这些基因可能参与的通路。P < 0.05为显著富集的阈值。
10. 统计学处理:qPCR实验重复3次。采用SPSS 13.0软件进行统计学分析,数据经正态性检验符合正态分布,用x±s表示,两组间比较采用两独立样本t检验,使用Origin 8.0进行作图。P < 0.05为差异有统计学意义。
结果1. 4 Gy 137Cs γ射线全身照射对小鼠骨髓细胞中circRNA m6A修饰位点及甲基化circRNA来源基因的影响:照后5 min,健康对照组和照射组中各鉴定出325和455个circRNA的m6A修饰位点。其中两组共有相同位点178个,健康对照组特有位点147个,照射组特有位点277个。同时,健康对照组和照射组中各鉴定出1 275和1 017个发生m6A修饰的circRNA的来源基因。其中,两组共有767个,健康对照组特有508个,照射组特有250个。以上结果说明,4 Gy 137Cs γ射线全身照射可以引起小鼠骨髓细胞中circRNA m6A修饰情况的快速改变。
2. 4 Gy 137Cs γ射线全身照射导致的小鼠骨髓细胞中circRNA差异m6A修饰位点分析:与健康对照组相比,照射组小鼠骨髓细胞中circRNA上414个位点的m6A修饰水平(富集倍数)显著上调(图 1 A),178个位点的m6A修饰水平(富集倍数)显著下调(图 1B),差异具有统计学意义(P < 10-10;倍数筛选阈值> 5)。上述代表性位点的变化趋势经MeRIP-qPCR实验得以验证,如位于mmu_circ_0001273(图 1C和1E)和mmu_circ_0000174(图 1D和1F)上的位点。
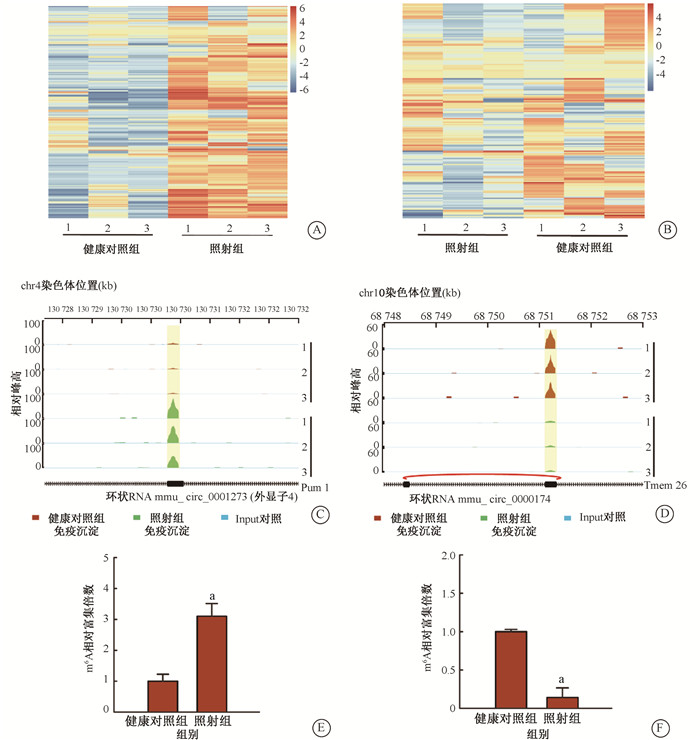
|
注:1、2、3为平行样;图C、D坐标轴上的0、100和60为衡量每个通道的相对峰高的坐标轴起点;a与健康对照组比较,t=3.285、6.967,P < 0.05 图 1 照后5 min小鼠骨髓细胞中发生差异修饰的circRNA m6A位点分析 A. 修饰水平上调的circRNA m6A位点热图分析;B. 修饰水平下调的circRNA m6A位点热图分析;C. mmu_circ_0001273上的上调m6A修饰位点示意图;D. mmu_circ_0000174上的下调m6A修饰位点示意图;E. MeRIP-qPCR实验验证mmu_circ_0001273上的m6A修饰位点变化趋势;F. MeRIP-qPCR实验验证mmu_circ_0000174上的m6A修饰位点变化趋势 Figure 1 The analysis of differentially methylated circRNA m6 A sites in mouse bone marrow cells at 5 min after ionizing radiation A. The heat-map of up-methylated circRNA m6A sites; B. The heat-map of down-methylated circRNA m6A sites; C. A schematic representing up-regulated m6A sites on mmu_circ_0001273; D. A schematic representing down-regulated m6A sites on mmu_circ_0000174; E. The variation tendency of m6A peaks on mmu_circ_0001273 was verified by MeRIP-qPCR; F. The variation tendency of m6A peaks on mmu_circ_0000174 was verified by MeRIP-qPCR |
3. 4 Gy 137Cs γ射线全身照射导致的小鼠骨髓细胞中差异m6A修饰circRNA来源基因的GO功能分析:如图 2所示,4 Gy 137Cs γ射线全身照射后,小鼠骨髓细胞中m6A修饰水平显著改变的circRNA来源基因涉及多种GO功能,如发生上调的生物进程中的对脂蛋白颗粒刺激的细胞应答(图 2A)、细胞组分中的臂间异染色质(图 2B)和分子功能中的赖氨酸乙酰化组蛋白结合(图 2C)等功能;以及发生下调的生物进程中的趋化因子分泌的正调控(图 2D)、细胞组分中的纤毛转换纤维(图 2E)和分子功能中的胰岛素受体底物结合(图 2F)等功能。
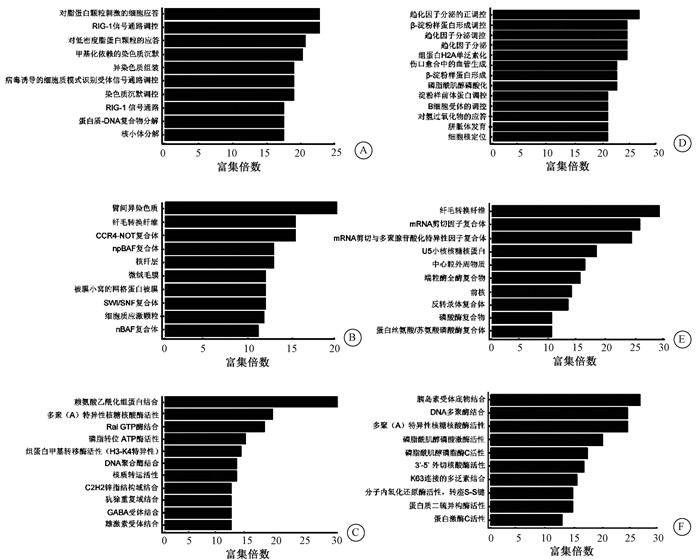
|
图 2 照射后5 min小鼠骨髓细胞中发生差异m6A修饰的circRNA来源基因的GO分析 A~C. m6A富集倍数上调的circRNA来源基因的生物进程(A)、细胞组分(B)和分子功能(C)分析;D~E. m6A富集倍数下调的circRNA来源基因的生物进程(D)、细胞组分(E)和分子功能(F)分析 Figure 2 GO analysis of circRNAs with differentially methylated m6A sites in mouse bone marrow cells at 5 minutes after ionizing radiation A-C. The analysis of biological process (A), cellular component (B) and molecular function (C) of deriving genes of circRNAs with fold-enrichment up-regulated m6A peaks; D-F. The analysis of biological process (D), cellular component (E) and molecular function (F) of deriving genes of circRNAs with fold-enrichment down-regulated m6A peaks |
4. 4 Gy 137Cs γ射线全身照射导致的小鼠骨髓细胞中差异m6A修饰circRNA来源基因的KEGG通路分析:如图 3所示,4 Gy 137Cs γ射线全身照射后,小鼠骨髓细胞中m6A修饰水平显著改变的circRNA来源基因涉及多条KEGG通路,如血小板激活、破骨细胞分化和白细胞跨内皮迁移等(上调,图 3 A);以及Fc γ R-介导的吞噬作用、Fc ε RI信号通路和B细胞受体信号通路等(下调,图 3B)。
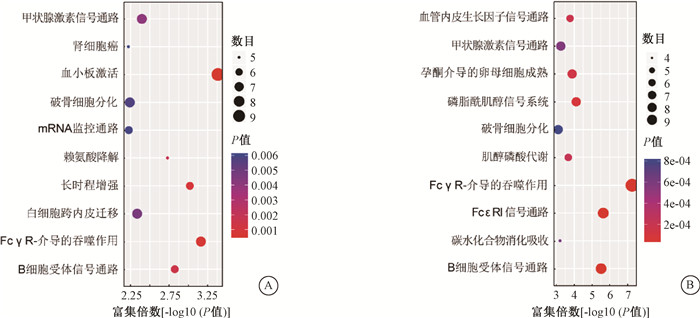
|
图 3 照射后5 min小鼠骨髓细胞中发生差异m6A修饰的circRNA来源基因的KEGG分析 A. m6A. 富集倍数上调;B. m6A富集倍数下调 Figure 3 KEGG analysis of circRNAs with differentially methylated m6A sites in mouse bone marrow cells at 5 minutes after ionizing radiation A. The deriving genes of circRNAs with fold-enrichment up-regulated m6A peaks; B. The deriving genes of circRNAs with fold-enrichment down-regulated m6A peaks |
讨论
骨髓是辐射敏感器官,电离辐射导致的骨髓损伤可进一步引发造血功能障碍[18]。通常认为,直接或间接作用导致DNA双链断裂,进而引发不可控的基因突变,是电离辐射损伤的主要机制[19]。然而,伴随表观遗传修饰研究的开启和深入,RNA转录后修饰在电离辐射诱导造血系统损伤中的作用亟待阐明。本课题聚焦电离辐射对早期造血系统辐射综合征中骨髓细胞的RNA转录后修饰的影响。m6A修饰被认为是存在于真核mRNA及非编码RNA内部的最普遍和保守的RNA表观遗传修饰方式[9],其研究方法成熟可靠,因此,本研究主要关注电离辐射对骨髓细胞中RNA的m6A修饰谱的影响。ENCODE计划揭示,非编码RNA在人类基因组中占据了> 97%的比例[20],这些“暗物质”虽然不编码蛋白质,但是却起着重要的调控作用,比如miRNA、lncRNA和circRNA等[21-22]。以往的研究多集中于mRNA的m6A修饰在多种生理病理进程中的作用,而本研究选取比线性mRNA更丰富稳定地circRNA为研究对象,具有较高的新颖性。
m6A修饰是一动态、可逆的甲基化过程[23]。本课题组前期研究结果显示,照后5 min,小鼠骨髓细胞的mRNA m6A修饰谱变化较为明显[10],circRNA的m6A修饰谱初步分析呈现相似的趋势,故本课题收集健康对照组和照射组小鼠的骨髓细胞为研究对象,并重点呈现照射完成5 min后的circRNA m6A修饰谱变化情况。后续研究中将进一步延长、增加采样时间点以阐明其较为完整的变化趋势。m6A修饰过程由负责不同作用的酶执行完成,可形象地称为“写入器(writers)”、“擦除器(erasers)”和“阅读器(readers)”。“writers”是催化完成甲基化过程的酶,包括由甲基转移酶样蛋白3(methyltransferase-like 3,METTL3)、甲基转移酶样蛋白14(methyltransferase-like 14,METTL14)和Wilms肿瘤1相关蛋白(Wilms′tumor 1-associated protein,WTAP)组成的甲基转移酶复合体和RNA结合基序蛋白15/15B(RNA-binding motif protein 15/15B,RBM15/15B);“erasers”是催化去甲基化过程的酶,目前已鉴定的该类酶主要包括脂肪量和肥胖相关蛋白(fat mass and obesity-associated protein,FTO)和alkB同源蛋白5(alkB homologue 5,ALKBH5);“readers”负责识别m6A甲基化修饰的酶,目前鉴定明确的主要为YT521-B同源性(YT521-B homology,YTH)结构域家族的酶,如YTH结构域家族蛋白-1/-2/-3(YTH domain family proteins,Ythdf-1/-2/-3),胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA binding protein,IGF2BP)、真核起始因子3(eukaryotic initiation factor 3,eIF)和异质核核糖核蛋白(heterogeneous nuclear ribonucleoprotein,HNRNP)等[24]。在后续研究中将着力鉴定应答电离辐射刺激的关键m6A修饰酶,并寻找其下游靶点以明确其中的分子机制。
作为一种遗传信息的精细调控模式,内稳态失衡(如高血压、糖尿病、精神紊乱等)和外部应激压力(如紫外线照射、光生物学、缺氧及热激作用等)均可触发RNA m6A修饰谱的灵敏波动[25-27]。而电离辐射是一种可诱发有机体应激反应的典型外部刺激,且已有报道揭示RNA的m6A修饰与肿瘤放射生物学之间存在密切关联[28]。本研究结果显示,4 Gy 137Cs γ射线全身照射可引起小鼠骨髓细胞中circRNA的m6A修饰谱的迅速(5 min)改变,差异修饰的circRNA的来源基因涉及多种造血系统放射生物学相关的功能通路,如染色质相关调控、血小板激活和B细胞受体信号通路等。该研究丰富了放射性造血系统损伤的知识体系,为后续研究提供了实验依据。
目前,氨磷汀是临床上唯一治疗放射损伤的成熟药物,但由于其注射给药不便和副作用大等原因而限制了其应用[29]。因此,新型损伤机制的鉴定对于辐射损伤防治至关重要。该研究为从表观遗传水平揭示造血系统辐射损伤的分子机制和开发新型辐射防护药物提供科学依据和思路。
在今后的研究中,将进一步揭示电离辐射诱导的多器官、多系统的RNA表观遗传修饰精确图谱,明确RNA m6A修饰与放射性造血系统损伤之间的关系并鉴定在其中发挥关键作用的具体m6A修饰circRNA及阐明其调节机制,力求开发针对于表观遗传学层面的辐射损伤阻断方法,对辐射防护研究提供更多的参考和思路。
利益冲突 本研究由署名作者按以下贡献声明独立开展,未因进行该研究而接受任何不正当的职务或财务利益,不涉及任何利益冲突
作者贡献声明 张书琴负责实验的开展实施、数据整理和论文的撰写;崔明负责实验的指导、协助实验操作及论文的修改;张梦然负责协助实验操作和数据分析;李源、肖惠文、董佳丽和尚悦负责协助实验操作和论文校对;樊赛军负责论文的设计、实验的指导和论文的审阅
| [1] |
谢光耀, 王锋, 张婷, 等. 电离辐射对小鼠骨髓造血干/祖细胞中CD47表达的影响[J]. 吉林大学学报(医学版), 2020, 46(6): 1169-1176, 1348. Xie GY, Wang F, Zhang T, et al. Effect of ionizing radiation on the expression of CD47 in bone marrow hematopoietic stem/progenitor cells of mice[J]. J Jilin Univ Med Edit, 2020, 46(6): 1169-1176, 1348. DOI:10.13481/j.1671-587x.20200610 |
| [2] |
宋曼, 关华, 王豫, 等. 丁香酸对小鼠骨髓造血系统辐射损伤的防护作用[J]. 中华放射医学与防护杂志, 2013, 33(4): 384-387. Song M, Guan H, Wang Y, et al. Protective effect of syringic acid on radiation injury of bone marrow hematopoietic system in mice[J]. Chin J Radiol Med Prot, 2013, 33(4): 384-387. DOI:10.3760/cma.j.issn.0254-5098.2013.04.012 |
| [3] |
Goodall GJ, Wickramasinghe VO. RNA in cancer[J]. Nat Rev Cancer, 2021, 21(1): 22-36. DOI:10.1038/s41568-020-00306-0 |
| [4] |
郭新园, 王蕊, 衣峻萱, 等. 环状RNA作为肿瘤标志物及其在肿瘤放疗中应用的研究进展[J]. 国际放射医学核医学杂志, 2020, 44(6): 374-380. Guo XY, Wang R, Yi JX, et al. Progress in the research on circRNAs as tumor markers and their applications in radiotherapy[J]. Int J Radiat Med Nucl Med, 2020, 44(6): 374-380. DOI:10.3760/cma.j.cn121381-201903043-00033 |
| [5] |
Li B, Zhu L, Lu C, et al. circNDUFB2 inhibits non-small cell lung cancer progression via destabilizing IGF2BPs and activating anti-tumor immunity[J]. Nat Commun, 2021, 12(1): 295. DOI:10.1038/s41467-020-20527-z |
| [6] |
Zimmerman AJ, Hafez AK, Amoah SK, et al. A psychiatric disease-related circular RNA controls synaptic gene expression and cognition[J]. Mol Psychiatr, 2020, 25(11): 2712-2727. DOI:10.1038/s41380-020-0653-4 |
| [7] |
Zeng Z, Xia L, Fan S, et al. Circular RNA circMAP3K5 acts as a microRNA-22-3p sponge to promote resolution of intimal hyperplasia via TET2-mediated smooth muscle cell differentiation[J]. Circulation, 2021, 143(4): 354-371. DOI:10.1161/CIRCULATIONAHA.120.049715 |
| [8] |
Tao X, Shao Y, Yan J, et al. Biological roles and potential clinical values of circular RNAs in gastrointestinal malignancies[J]. Cancer Biol Med, 2021, 18(2): 437-457. DOI:10.20892/j.issn.2095-3941.2020.0348 |
| [9] |
Zhang L, Hou C, Chen C, et al. The role of N(6)-methyladenosine (m6A) modification in the regulation of circRNAs[J]. Mol Cancer, 2020, 19(1): 105. DOI:10.1186/s12943-020-01224-3 |
| [10] |
Zhang S, Dong J, Li Y, et al. Gamma-irradiation fluctuates the mRNA N(6)-methyladenosine (m6A) spectrum of bone marrow in hematopoietic injury[J]. Environ Pollut, 2021, 285: 117509. DOI:10.1016/j.envpol.2021.117509 |
| [11] |
Meyer KD, Saletore Y, Zumbo P, et al. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons[J]. Cell, 2012, 149(7): 1635-1646. DOI:10.1016/j.cell.2012.05.003 |
| [12] |
Kechin A, Boyarskikh U, Kel A, et al. cutPrimers: A new tool for accurate cutting of primers from reads of targeted next generation sequencing[J]. J Comput Biol, 2017, 24(11): 1138-1143. DOI:10.1089/cmb.2017.0096 |
| [13] |
Dobin A, Davis CA, Schlesinger F, et al. STAR: ultrafast universal RNA-seq aligner[J]. Bioinformatics, 2013, 29(1): 15-21. DOI:10.1093/bioinformatics/bts635 |
| [14] |
Cheng J, Metge F, Dieterich C. Specific identification and quantification of circular RNAs from sequencing data[J]. Bioinformatics, 2016, 32(7): 1094-1096. DOI:10.1093/bioinformatics/btv656 |
| [15] |
Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements[J]. Nat Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 |
| [16] |
Zhang Y, Liu T, Meyer CA, et al. Model-based analysis of ChIP-Seq (MACS)[J]. Genome Biol, 2008, 9(9): R137. DOI:10.1186/gb-2008-9-9-r137 |
| [17] |
Shen L, Shao NY, Liu X, et al. diffReps: detecting differential chromatin modification sites from ChIP-seq data with biological replicates[J]. PLoS One, 2013, 8(6): e65598. DOI:10.1371/journal.pone.0065598 |
| [18] |
张俊伶, 李德冠, 路璐, 等. Vam3对辐射所致小鼠骨髓单个核细胞损伤的保护作用[J]. 中华放射医学与防护杂志, 2014, 34(9): 676-677. Zhang JL, Li DG, Lu L, et al. Protective effect of vam3 on radiation-induced injury of bone marrow mononuclear cells in mice[J]. Chin J Radiol Med Prot, 2014, 34(9): 676-677. DOI:10.3760/cma.j.issn.0254-5098.2014.09.008 |
| [19] |
Leibowitz BJ, Wei L, Zhang L, et al. Ionizing irradiation induces acute haematopoietic syndrome and gastrointestinal syndrome independently in mice[J]. Nat Commun, 2014, 5: 3494. DOI:10.1038/ncomms4494 |
| [20] |
Djebali S, Davis CA, Merkel A, et al. Landscape of transcription in human cells[J]. Nature, 2012, 489(7414): 101-108. DOI:10.1038/nature11233 |
| [21] |
曾晓辉, 卢帅, 汪朝晖. 环状RNA在急性心肌梗死心脏损伤与修复中作用的研究进展[J]. 山东医药, 2021, 61(4): 97-100. Zeng XH, Lu S, Wang ZH. Research progress on the role of circular RNA in the heart injury and repair of acute myocardial infarction[J]. Shandong Med J, 2021, 61(4): 97-100. DOI:10.3969/j.issn.1002-266X.2021.04.025 |
| [22] |
付海璐, 迟鑫明, 邵淑娟. 环状RNA研究进展[J]. 中国生化药物杂志, 2016, 36(5): 8-11. Fu HL, Chi XM, Shao SJ. Research progress of circular RNAs[J]. Chin J Biol Pharms, 2016, 36(5): 8-11. DOI:10.3969/j.issn.1005-1678.2016.05.03 |
| [23] |
Fu Y, Dominissini D, Rechavi G, et al. Gene expression regulation mediated through reversible m6A RNA methylation[J]. Nat Rev Genet, 2014, 15(5): 293-306. DOI:10.1038/nrg3724 |
| [24] |
Livneh I, Moshitch-Moshkovitz S, Amariglio N, et al. The m6A epitranscriptome: transcriptome plasticity in brain development and function[J]. Nat Rev Neurosci, 2020, 21(1): 36-51. DOI:10.1038/s41583-019-0244-z |
| [25] |
Dorn LE, Lasman L, Chen J, et al. The N6-methyladenosine mRNA methylase METTL3 controls cardiac homeostasis and hypertrophy[J]. Circulation, 2019, 139(4): 533-545. DOI:10.1161/CIRCULATIONAHA.118.036146 |
| [26] |
Wu J, Zhao Y, Wang X, et al. Dietary nutrients shape gut microbes and intestinal mucosa via epigenetic modifications[J]. Crit Rev Food Sci Nutr, 2020, 1-15. DOI:10.1080/10408398.2020.1828813 |
| [27] |
Zhao BS, Roundtree IA, He C. Post-transcriptional gene regulation by mRNA modifications[J]. Nat Rev Mol Cell Biol, 2017, 18(1): 31-42. DOI:10.1038/nrm.2016.132 |
| [28] |
Xiang M, Liu W, Tian W, et al. RNA N-6-methyladenosine enzymes and resistance of cancer cells to chemotherapy and radiotherapy[J]. Epigenomics, 2020, 12(9): 801-809. DOI:10.2217/epi-2019-0358 |
| [29] |
Yu X, Li M, Zhu L, et al. Amifostine-loaded armored dissolving microneedles for long-term prevention of ionizing radiation-induced injury[J]. Acta Biomater, 2020, 112: 87-100. DOI:10.1016/j.actbio.2020.05.025 |
 2021, Vol. 41
2021, Vol. 41


