2. 中国人民解放军军事科学院军事医学研究院辐射医学研究所, 北京 100850
2. The Institute of Radiation Medicine, The Academy of Military Medical Science, Beijing 100850, China
辐射诱发癌症是电离辐射生物效应的远期健康危害,其启动和促发的关键内在因素与细胞核DNA损伤及引发的基因组不稳定性有很大关联[1-2],但是其机制目前并不明确。在受照细胞不断增殖、癌变及肿瘤形成过程中,会发生一系列的分子、代谢和表观遗传调控机制的改变[3-6],其中包括非编码RNA及其调控的细胞增殖和代谢网络的变化[5]。核仁小分子RNA(small nucleolar RNA, snoRNA)是一类小分子非编码RNA,多富集于核仁,根据其结构的不同,主要分为C/D box snoRNAs与H/ACA box snoRNAs,C/D box snoRNAs (SNORDs)是一种丰富表达的短的非编码RNA类,长期以来人们已经公认它们可以进行核糖体RNA (rRNA)的2′-O-甲基化,在rRNA前体的剪接加工和转录后修饰过程中起重要作用。大约有一半的人类SNORDs没有可预测的rRNA靶标[7],并且许多SNORDs与无rRNA缺陷的疾病有关[8],其中包括帕魏二氏综合征(PWS, Prader-Willi syndrome)[9]、Duplication 15q综合征和癌症[10]。近年来,snoRNA越来越多的功能相继被发现,有文献报道,snoRNA及其衍生的小RNA的表达及机制与肿瘤密切相关[11]。但是迄今为止,尚少有报道snoRNA与辐射致癌的关系,这方面的研究将有助于揭示snoRNA更多的功能,进一步了解放射生物学效应的机制。本研究利用高传能线密度(LET)α粒子辐射诱发人支气管上皮细胞BEP2D癌变的细胞模型,鉴定辐射致癌相关的snoRNA分子,对其功能进行生物信息学分析预测。
材料与方法1.细胞培养及样品收集:正常人支气管上皮细胞BEP2D由美国Harry博士赠送,本实验室保存。1.5 Gy α粒子照射诱发BEP2D癌变并形成肿瘤细胞系BERP35T-4,由本实验室建立。细胞在无血清培养基LHC-8(美国GIBCO公司)、饱和湿度、37℃恒温、5% CO2无菌培养箱中培养。
2.细胞RNA的提取:细胞加适量TRIzol(美国Sigma-Aldrich公司),收集于1.5 ml无RNase EP管中,加入200 μl的氯仿,剧烈振荡15 s,室温静置15 min;4℃、9 660 ×g,离心15 min,转移上清于新的1.5 ml无RNase EP管中,避免吸取到白色薄膜层和红色有机相;加入0.5 ml异丙醇,颠倒混匀,室温下静置10 min;4℃、9 660 ×g,离心10 min,离心后EP管底部肉眼可见少量白色沉淀;弃上清,加入0.5 ml 75%乙醇(DEPC水配制);剧烈振荡,4℃、4 300× g,离心5 min,吸去乙醇,室温晾干,直至EP管底部白色沉淀变为透明无色;加入15~30 μl焦碳酸二乙酯(DEPC)水,37℃或55℃孵育10 min;混匀RNA溶液,NanoDrop2000/2000c软件测量提取的RNA浓度,置于-70℃保存。
3.全基因转录组芯片分析:采用Affymetrix人类转录组基因芯片Array 2.0,试剂包括Ambion WT表达Kit(RNA扩增试剂盒)、GeneChipTM WT末端标记和控制试剂盒等,由上海其明信息技术有限公司进行芯片杂交分析。具体流程为:样品总RNA质量检测,总RNA扩增、cDNA标记,并使用RNase mini试剂盒进行纯化。在预定的杂交时间后,洗涤并扫描微阵列载玻片。用RVM修正的两独立样本t检验,对实验组和对照组进行差异筛选,计算P值和错误发现率(FDR),以评估RNA差异表达的重要性,变化倍数> 2,P<0.05为差异表达RNA。
4.反转录与实时定量PCR:使用日本TOYOBO公司的反转录试剂盒(Code No.FSQ-301),将提取的总RNA按照37℃ 15 min,50℃ 5 min,98℃ 5 min,4℃停止的反应程序来合成cDNA模板,之后用日本TOYOBO公司的实时定量荧光试剂盒测量RNA表达量,每个样品的每种RNA进行3次平行实验,反应程序为95℃ 20 s;95℃ 15 s,60℃ 40 s;后面两步重复39个以上循环。实时定量PCR反应完成后,保存实验数据进行分析,按照2-△△Ct法处理数据,计算RNA在不同样品中的表达量变化。实验中所用到的snoRNA,snoRNA衍生的sdRNA,mRNA的引物序列如表 1~3所示(sdRNA采用茎环法检测)。
|
|
表 1 sno116-14衍生的sdRNA分子cDNA合成的反转录引物 Table 1 sno116-14 derived sdRNA molecule cDNA synthesis reverse transcription primer |
|
|
表 2 snoRNA及其衍生的sdRNA的分子实时定量PCR引物 Table 2 Real-time quantitative PCR primers for snoRNA and its derived sdRNA |
|
|
表 3 芯片分析中与sno116-14相关的mRNA分子实时定量PCR引物 Table 3 Real-time quantitative PCR primers for mRNA molecules related to sno116-14 in chip analysis |
|
|
表 4 预测的sno116-14的靶mRNA分子实时定量PCR引物 Table 4 Predicted sno116-14 target mRNA molecule real-time quantitative PCR primers |
5.生物信息学预测靶标:采用3种方法对snoRNA的功能区进行预测。第1种方法是用PLEXY软件预测其功能区片段及作用的靶标片段,限制条件为:snoRNA与其靶标相互作用的碱基序列长度至少应为7 nt,中间没有插入或缺失碱基,核心双链体区域最多包含1个错配,且甲基化残基形成沃森-克里克碱基配对[12];第2种方法是用RNA Structure软件对snoRNA结构进行预测,根据预测的二级结构图,推测其可能的功能区片段,之后在美国国家生物技术信息中心(NCBI)数据库中,按照碱基互补匹配的方式预测靶RNA;第3种方法是推测snoRNA可能以其衍生的sdRNA发挥功能,根据文献中报道的sdRNA的碱基序列[13],在NCBI数据库中以碱基互补配对的方式搜索sdRNA的靶RNA。本课题组前期研究发现,与BEP2D相比,BERP35T-4细胞具有失去接触抑制、呈密集复层生长、细胞骨架排列紊乱、细胞增大且出现异形性、核质比例增大、核仁突出且异形、细胞器发达、体外增殖能力增强、可在软琼脂中形成克隆等特征,因此本实验在可能的靶标中筛选出与细胞增殖、细胞骨架结构相关的mRNA进行验证。
6.过表达载体的构建与转染:选用PLKO.1质粒构建过表达载体,上海生工生物工程公司合成带酶切位点的正义反义链,如表 5所示。之后将合成好的片段退火形成双链,并将PLKO.1载体双酶切,将酶切好的载体与合成的片段在T4DNA连接酶的作用下按1:4的比例连接,经过转化摇菌提取质粒,测序正确,表明过表达载体构建成功。提前12~24 h铺板,待细胞汇合度为90%时,用polyplus的jetPRIME转染试剂(PT-114-15)按说明书进行转染,4 h后换液。
|
|
表 5 sno116-14过表达载体构建的正义反义链的碱基序列 Table 5 Base sequence of sense antisense strand constructed by sno116-14 overexpression vector |
7.统计学处理:采用SPSS 21.0软件进行统计分析,每个实验均重复3次。在样品数据正态分布和方差齐的条件下,计量资料表示为 x±s。t检验用于两组间的差异比较,方差分析用于多组之间的数据比较,秩和检验用于方差不齐的数据。P<0.05为差异有统计学意义。
结果1. snoRNA表达谱的变化:芯片分析结果显示,相比较正常人支气管上皮细胞BEP2D,α粒子辐射诱导癌变的BERP35T-4细胞中,snoRNA分子表达谱中有的降低,有的升高。图 1所示为BEP2D和BERP35T-4(T35-4)细胞中部分snoRNA表达谱聚类分析的分子变化结果,癌变细胞中更多的snoRNA分子的表达降低。
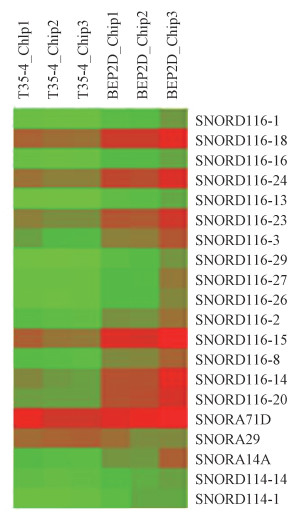
|
图 1 芯片分析snoRNA表达聚类分析 Figure 1 chip assay of snoRNA expressions and their cluster analysis |
2.表达下调的snoRNA的qRT\|PCR验证:qRT-PCR对sno116家族成员的表达变化进行验证,结果显示,sno116家族在BERP35T-4中表达量是BEP2D的0.105%,差异有统计学意义(t=26.60, P<0.01);BEP2D、A549、H460、H1299中sno116家族的表达量分别是BEAS-2B的193%、52%、104%、0.898%,差异具有统计学意义(t=11.85、20.17、10.40、36.11, P<0.01)。表明相比正常人支气管上皮细胞,sno116家族不仅在BERP35T-4细胞中表达量下降,在其他肺癌细胞中也普遍呈下调趋势,尤其在H1299细胞中下调最为明显。
3. sdRNA(snoRNA衍生的小分子RNA)的qRT\|PCR验证:上面已经验证了sno116家族在癌细胞中普遍下调,之后选取sno116-14作为后续研究对象。有文献报道,snoRNA会衍生为sdRNA,且部分H/ACA box snoRNA衍生的sdRNA有类miRNA作用[13]。本实验对sno116-14衍生的sdRNA进行qRT\|PCR的验证,结果显示,在BERP35T-4细胞中,由sno116-14衍生的sd116-14-1、sd116-14-3、sd116-14-4的表达量分别是sd116-14-2的4.07、3.39、1.59倍,差异具有统计学意义(t=6.80、5.50、6.14,P<0.05),表明衍生的sdRNA各自的表达量存在明显差异,推测表达量少的sd116-14-2和(或)sd116-14-4通过与靶分子的结合进而被降解。
4.生物信息学预测sno116-14的功能区域:用PLEXY软件限定条件后预测sno116-14的功能片段为5′ ATACCGTCATTCTCGTCGAA 3′,作用的靶标序列为GGAmCGGGAGCGGCGGG&ACCGTCATTCTCGTCG。用RNA Structure软件对snoRNA结构进行预测,根据预测的二级结构图,推测其可能的功能区片段为5′ TATATACATTCCT 3′、5′ AAGCTGAACAAAAT 3′(图 2)。以sno116-14衍生的sdRNA为功能区发挥作用预测,sd116-14-2:ATACATTCCTTGGAAAGCT,sd116-14-4:CTCGTCGAACTGAGGTCC为功能区片段。在NCBI RNA数据库中检索与预测功能区片段碱基完全互补或包含PLEXY软件预测的靶标片段的RNA,从中筛选出与细胞增殖、细胞骨架结构相关的mRNA进行验证,本实验筛选的mRNA有ZNF280D、TFDP1、CCDC28B、RPS6KA3、CANX、RUNX1、KALRN。
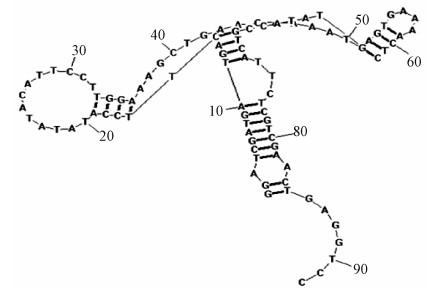
|
图 2 RNA structure预测的sno116-14的二级结构图 Figure 2 The secondary structure of sno116-14 predicted by RNA structure |
5. sno116家族的mRNA-lncRNA共表达网络:通过生物信息学分析sno116-14的mRNA和lncRNA的共表达网络如图 3所示。图中所示与sno116-14共表达能力较强的mRNA有CANX、ATP2A2、DROSHA,且这3条mRNA与sno116-14均表现为负调控。sno116-14-mRNA-lncRNA共表达网络指示的这些lncRNA和mRNA分子信息,为下一步探索snoRNA的功能提供了重要线索。
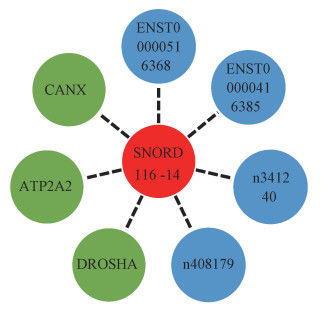
|
sno116-14的mRNA-lncRNA共表达网络图 图 3 sno116-14的mRNA-lncRNA共表达网络图 Figure 3 The co-expression network diagram of sno116-14-mRNA-lncRNA |
6. sno116-14靶标及芯片分析中与sno116-14共表达性强的mRNA分子的qRT\|PCR验证:将构建好的sno116-14过表达载体转染到BERP35T-4细胞中,同时以空载作为对照,转染48 h后收集细胞,验证相应共表达mRNA及预测的靶标的表达水平变化(图 4)。结果显示,sno116-14过表达后,与芯片一致的是mRNA ATP2A2、DROSHA呈现下降趋势,CANX无变化,预测的靶标ZNF280D、TFDP1、CCDC28B、RPS6KA3都出现上升或下调趋势,RUNX1、KALRN无变化;除TFDP1外,其余差异具有统计学意义(t=4.66、11.17、17.09、8.73、9.32,P<0.05)。
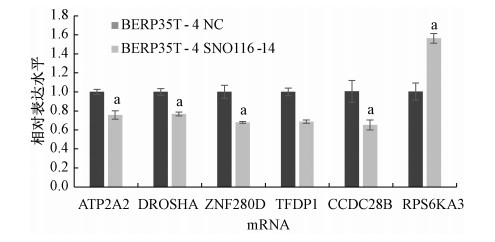
|
a同组比较, t = 4.66, 11.17, 17.09, 8.73, 9. 32, P<0. 05 图 4 过表达sno116-14后相关mRNA的表达变化 Figure 4 Changes of mRNA expressions after overexpression of sno116-14 |
讨论
研究表明,α射线暴露与肺癌之间联系十分密切,Park等[15]报道了长时间暴露在α射线的居民会增加肺癌的发病率;Grellier等[16]报道了长时间暴露在氡环境中的居民会增加肺癌的发病率。α粒子引发肺癌的作用机制中,原初始动环节中包括α粒子诱导的DNA损伤、基因组稳定性改变和DNA修复抑制导致的突变[17]。近年来,肺癌与snoRNA间的关系相继被报道,如SNORD78通过诱导上皮间质转化(EMT)促进NSCLC细胞的侵袭[18];小核仁RNA 71 A通过MAPK/ERK途径促进肺癌细胞的增殖、迁移和侵袭[19];SNORD42被证明具有致癌作用并促进肺癌的发生[20]。然而,辐射致癌与snoRNA间的关系及作用机制仍然是个未解之谜。
本研究以BEP2D细胞及其衍生的α粒子诱发癌变细胞BERP35T-4为模型进行转录组表达芯片分析,发现SNORD116家族在BERP35T-4细胞中表达量普遍下降,SNORD116属于box C/D snoRNA家族,有29个序列相近的家族成员,是父系印迹表达单元IC-SNTRF-SNRPN基因的下游内含子编码的snoRNA[21],其缺失是Prader Williz综合征发病的主要遗传因素[22]。迄今为止,sno116家族找不到能够与之相匹配的rRNA或者snRNA的互补序列,因此又被称为孤儿snoRNA(orphan snoRNA)。虽然有预测出潜在的作用靶点,但是至今还未确切证实[23]。有研究称,SNORD116家族中有一些成员参与组成具有5′和3′ snoRNA结构的lncRNA,SNORD116在两端的加尾增强了lncRNA的稳定性,同时这种sno-lncRNAs在Prader Williz综合征中作用机制与剪接因子RBFOX2有关[24]。同时,也有报道称SNORD116的表达与疾病之间的联系,在嗜酒的孕妇中引起胎儿大脑发育异常的过程中,SNORD116表达下调[25]。在小非编码RNA与肺癌相关性的研究中,报道了SNORD116-26在非小细胞肺癌中表达降低[26]。神经胶质瘤细胞在低氧刺激下分泌的外泌体中SNORD116-21表达明显上调[27]。关于snoRNA衍生的sdRNA的研究中,有报道称SNORD116家族由于其序列中含有非典型的box C′或者box D′,其家族成员可以衍生出有高度保守序列的3~4条sdRNA[13],而sdRNA的表达丰度与癌症的分化程度可能有一定联系。遗憾的是,到目前为止,SNORD116家族成员本身的作用机制少见任何报道。
本研究通过验证sno116家族及其衍生的sdRNA的表达量,发现不仅在BERP35T-4细胞中,其他的肺癌细胞A549、H1299中表达量也出现下调,尤其是在H1299细胞中,下调比例尤为明显。有趣的是,从美国模式培养物集存库(ATCC)得知,H1299细胞是从先前接受过放射治疗的患者的肺部转移淋巴结建立的细胞系,这可能从某种角度说明了sno116家族与电离辐射致癌的密切关系。并且本研究还发现sno116-14衍生的各sdRNA在同一细胞中的表达量亦有所区别,表达量最少的sd116-14-2和sd116-14-4的序列分别位于sno116-14 D′ box和D box上游,且其碱基序列分别包含D′ box和D box上游第5个碱基,snoRNA经典的反义互补靶标即为D或D′box上游第5碱基处[28],因此,推测snoRNA以sdRNA形式发挥类miRNA功能,进一步被降解。
近期已有研究发现,snoRNA不仅靶向rRNA及snRNA,对其他RNA如mRNA、tRNA等皆可靶向发挥作用。Nostramo和Hopper[29]发现tRNA可作为snoRNA 2′-O-甲基化的靶标,介导tRF的生成。小核仁RNA U32a、U33和U35a是代谢应激的关键介导因子,通过2′-O甲基化调节mRNA发挥作用[30]。在美国国家生物技术信息中心(NCBI)中对sno116-14的靶标进行预测时,发现与预测的功能区互补的RNA普遍为mRNA。在这些mRNA中,本研究挑选了与细胞骨架结构或细胞增殖相关的mRNA进行验证,结果表明在BERP35T-4细胞中,对sno116-14过表达后,部分预测的靶mRNA及在芯片分析中共表达能力强的mRNA的表达量发生了变化。通过对这些mRNA进行分析,初步显示sno116-14可能通过MAPK信号通路、mTOR信号通路、选择性剪切、细胞黏附、细胞增殖等发挥作用。
综上所述,本研究报道了α粒子辐射致细胞癌变过程中发生明显表达变化的snoRNA,对表达差异显著的snoRNA的靶标和生物学功能进行了预测,为深入探讨电离辐射致癌的机制提供了更多的线索。
利益冲突 无
作者贡献声明 岳小乔负责课题设计、实验操作和论文撰写;白琛俊负责课题思路的探讨、生信分析;谢达菲负责生信分析;刘晓丹负责实验操作;周平坤负责提出研究思路、结果分析讨论,指导论文写作和修改
| [1] |
Suzuki K, Ojima M, Kodama S, et al. Radiation-induced DNA damage and delayed induced genomic instability[J]. Oncogene, 2003, 22(45): 6988-6993. DOI:10.1038/sj.onc.1206881 |
| [2] |
Farhood B, Goradel NH, Mortezaee K, et al. Melatonin and cancer:from the promotion of genomic stability to use in cancer treatment[J]. J Cell Physiol, 2019, 234(5): 5613-5627. DOI:10.1002/jcp.27391 |
| [3] |
Sun JF, Sui JL, Zhou PK, et al. Decreased efficiency of γ-ray-induced DNA double-strand break rejoining in malignant transformants of human bronchial epithelial cells generated by alpha-particle exposure[J]. Int J Radiat Biol, 2002, 78(9): 773-780. DOI:10.1080/09553000210141441 |
| [4] |
Sui JL, An J, Sun JF, et al. Spindle checkpint and apoptotic response in α particles-transformed human bronchial epithelial cells[J]. Radiat Environ Biophys, 2004, 43(4): 257-263. DOI:10.1007/s00411-004-0268-1 |
| [5] |
Liu XD, Xie DF, Wang YL, et al. Integrated analysis of lncRNA-mRNA co-expression networks in the α-particle induced carcinogenesis of human branchial epithelial cells[J]. Int J Radiat Biol, 2019, 95(2): 144-155. DOI:10.1080/09553002.2019.1539880 |
| [6] |
Weaver DA, Hei TK, Hukku B, et al. Cytogenetic and molecular genetic analysis of tumorigenic human bronchial epithelial cells induced by radon alpha particles[J]. Carcinogenesis, 1997, 18(6): 1251-1257. DOI:10.1093/carcin/18.6.1251 |
| [7] |
Falaleeva M, Welden JR, Duncan MJ, et al. C/D-box snoRNAs form methylating and non-methylating ribonucleoprotein complexes:Old dogs show new tricks[J]. Bioessays, 2017, 39(6): 1600264. DOI:10.1002/bies.201600264 |
| [8] |
Dupuis-Sandoval F, Poirier M, Scott MS. The emerging landscape of small nucleolar RNAs in cell biology[J]. Wiley Interdiscip Rev RNA, 2015, 6(4): 381-397. DOI:10.1002/wrna.1284 |
| [9] |
Cavaillé J. Box C/D small nucleolar RNA genes and the Prader-Willi syndrome:a complex interplay[J]. Wiley Interdiscip Rev RNA, 2017, 8(4). DOI:10.1002/wrna.1417 |
| [10] |
KISS T. Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs[J]. Embo J, 2001, 20(14): 3617-3622. DOI:10.1093/emboj/20.14.3617 |
| [11] |
Chow RD, Chen S. Sno-derived RNAs are prevalent molecular markers of cancer immunity[J]. Oncogene, 2018, 37(50): 6442-6462. DOI:10.1038/s41388-018-0420-z |
| [12] |
Kehr S, Bartschat S, Stadler PF, et al. PLEXY:efficient target prediction for box C/D snoRNAs[J]. Bioinformatics, 2011, 27(2): 279-280. DOI:10.1093/bioinformatics/btq642 |
| [13] |
Martens-uzunova ES, Hoogstrate Y, Kalsbeek A, et al. C/D-box snoRNA-derived RNA production is associated with malignant transformation and metastatic progression in prostate cancer[J]. Oncotarget, 2015, 6(19): 17430-17444. DOI:10.18632/oncotarget.4172 |
| [14] |
Cheng TY, Cramb SM, Baade PD, et al. The international epidemiology of lung cancer:latest trends, disparities, and tumor characteristics[J]. J Thorac Oncol, 2016, 11(10): 1653-1671. DOI:10.1016/j.jtho.2016.05.021 |
| [15] |
Park EJ, Lee H, Kim HC, et al. Residential radon exposure and cigarette smoking in association with lung cancer:a matched case-control study in Korea[J]. Int J Environ Res Public Health, 2020, 17(8). DOI:10.3390/ijerph17082946 |
| [16] |
Grellier J, Atkinson W, Berard P, et al. Risk of lung cancer mortality in nuclear workers from internal exposure to alpha particle-emitting radionuclides[J]. Epidemiology, 2017, 28(5): 675-684. DOI:10.1097/EDE.0000000000000684 |
| [17] |
Minina VI, Sinitsky MY, Druzhinin VG, et al. Chromosome aberrations in peripheral blood lymphocytes of lung cancer patients exposed to radon and air pollution[J]. Eur J Cancer Prev, 2018, 27(1): 6-12. DOI:10.1097/CEJ.0000000000000270 |
| [18] |
Zheng D, Zhang J, Ni J, et al. Small nucleolar RNA 78 promotes the tumorigenesis in non-small cell lung cancer[J]. J Exp Clin Cancer Res, 2015, 34(1): 49. DOI:10.1186/s13046-015-0170-5 |
| [19] |
Tang G, Zeng Z, Sun W, et al. Small nucleolar RNA 71A promotes lung cancer cell proliferation, migration and invasion via MAPK/ERK pathway[J]. J Cancer, 2019, 10(10): 2261-2275. DOI:10.7150/jca.31077 |
| [20] |
Mei Y, Liao J, Shen J, et al. Small nucleolar RNA 42 acts as an oncogene in lung tumorigenesis[J]. Oncogene, 2012, 31(22): 2794-2804. DOI:10.1038/onc.2011.449 |
| [21] |
Runte M, Huttenhofer A, Gross S, et al. The IC-SNURF-SNRPN transcript serves as a host for multiple small nucleolar RNA species and as an antisense RNA for UBE3A[J]. Hum Mol Genet, 2001, 10(23): 2687-2700. DOI:10.1093/hmg/10.23.2687 |
| [22] |
Rodriguez JA, Zigman JM. Hypothalamic loss of Snord116 and Prader-willi syndrome hyperphagia:the buck stops here?[J]. J Clin Invest, 2018, 128(3): 900-902. DOI:10.1172/JCI99725 |
| [23] |
Cao P, Yang A, Wang R, et al. Germline duplication of SNORA18L5 increases risk for HBV-related hepatocellular carcinoma by altering localization of ribosomal proteins and decreasing levels of p53[J]. Gastroenterology, 2018, 155(2): 542-556. DOI:10.1053/j.gastro.2018.04.020 |
| [24] |
Yin QF, Yang L, Zhang Y, et al. Long noncoding RNAs with snoRNA ends[J]. Mol Cell, 2012, 48(2): 219-230. DOI:10.1016/j.molcel.2012.07.033 |
| [25] |
Laufer BI, Mantha K, Kleiber ML, et al. Long-lasting alterations to DNA methylation and ncRNAs could underlie the effects of fetal alcohol exposure in mice[J]. Dis Model Mech, 2013, 6(4): 977-992. DOI:10.1242/dmm.010975 |
| [26] |
Mannoor K, Shen J, Liao J, et al. Small nucleolar RNA signatures of lung tumor-initiating cells[J]. Mol Cancer, 2014, 13: 104. DOI:10.1186/1476-4598-13-104 |
| [27] |
Kore RA, Edmondson JL, Jenkins SV, et al. Hypoxia-derived exosomes induce putative altered pathways in biosynthesis and ion regulatory channels in glioblastoma cells[J]. Biochem Biophys Rep, 2018, 14: 104-113. DOI:10.1016/j.bbrep.2018.03.008 |
| [28] |
Gumienny R, Jedlinski DJ, Schmidt A, et al. High-throughput identification of C/D box snoRNA targets with CLIP and RiboMeth-seq[J]. Nucleic Acids Res, 2017, 45(5): 2341-2353. DOI:10.1093/nar/gkw1321 |
| [29] |
Nostramo RT, Hopper AK. Beyond rRNA and snRNA:tRNA as a 2'-O-methylation target for nucleolar and Cajal body box C/D RNPs[J]. Genes Dev, 2019, 33(13-14): 739-740. DOI:10.1101/gad.328443.119 |
| [30] |
Elliott BA, Ho HT, Ranganathan SV, et al. Modification of messenger RNA by 2'-O-methylation regulates gene expression in vivo[J]. Nat Commun, 2019, 10(1): 3401. DOI:10.1038/s41467-019-11375-7 |
 2020, Vol. 40
2020, Vol. 40


