2. 北京连心医疗科技有限公司 100085 张旭阳现在首都医科大学附属北京潞河医院肿瘤中心, 北京 101100
2. Beijing Linking Medical Technology Co., Ltd, Beijing 100085, China
近年来,放疗技术发展迅速,图像引导的调强放射治疗、容积旋转调强放射治疗等高精度放射治疗方法被广泛用于肿瘤治疗。准确勾画危及器官(organs at risk, OARs)是避免放疗过程中发生较为严重的不良反应的关键步骤[1]。通常一例患者有几十到上百张CT图像,每张CT图像都需要勾画多个OARs,该过程耗费临床医生大量时间和精力[2],并且勾画结果受临床医生业务水平影响而参差不齐[3-4]。自动勾画作为一种辅助工具,可以帮助医生自动勾画一些结构,并且降低勾画者之间的差异,提高勾画一致性。基于深度学习[5-6]和图谱[7-8]的自动勾画方法是目前两种主要的自动勾画方法。本研究设计了基于深度学习的自动勾画模型(由勾画模型和分类模型组成),用于勾画头颈部OARs,并将结果与基于图谱方法的Smart segmentation软件勾画结果比较,探讨不同方法对头颈部OARs勾画的差异。
资料与方法1.病例数据准备:回顾性选取2012年4月至2018年10月,在北京大学第三医院接受放射治疗的头颈部肿瘤患者170例。患者均采取仰卧位、热塑膜固定,采用荷兰飞利浦Brilliance Big Bore CT模拟定位机采集图像,扫描层厚1~3 mm。本研究涉及的OARs(脑干、眼球、视神经、颞叶、下颌骨和腮腺)均由高年资临床医生根据相关勾画指南[9]对勾画结果进行二次核对,确保勾画准确一致。同时,由临床医生对每例患者的每层CT图像进行分类标注,用于后续分类模型训练和测试。
将170例患者按照就诊ID号码从小到大按照随机数表法分为2组,前150例用于自动勾画模型训练(其中120例作为训练集,30例作为验证集),同时也用于Smart segmentation图谱库建立;后20例作为测试集,分别使用两种方法进行自动勾画并比较结果差异。
2.基于深度学习的自动勾画模型建立
本研究设计的基于深度学习的自动勾画模型由两部分组成:分类模型和勾画模型。分类模型在头脚方向上对CT图像进行分类,并将危及器官所在分类的CT图像提取出来,输入到对应危及器官的勾画模型中。勾画模型仅对分类模型分类提取出的CT图像进行自动勾画。
(1) 数据预处理:为便于模型训练,使用双线性插值法将CT图像的平面分辨率从原始的0.69 mm×0.69 mm至1.34 mm×1.34 mm之间,重新采样为1 mm×1 mm。同时,为了提高自动勾画模型的泛化能力和适用性,对CT图像进行数据增强处理以增加训练图像数据的多样性,如对CT图像在20°以内进行随机顺时针或逆时针旋转,或对CT图像在20个像素以内进行随机平移。
(2) 分类模型:分类模型由深度可分离卷积层(depth-wise separable convolution)[10]、连接层(concatenate)、最大池化层(max pooling)和全局平均池化层(global average pooling)组成(图 1)。根据危及器官的解剖特征和位置特征,分类模型利用5条分界线将每例患者的CT图像在头脚方向分为6类(图 2)。第Ⅰ类和第Ⅱ类分界线为颅骨出现层面,第Ⅱ类和第Ⅲ类分界线为眼球上层出现层面,第Ⅲ类和第Ⅳ类分界线为眼球下层出现层面,第Ⅳ类和第Ⅴ类分界线为小脑下层出现层面,第Ⅴ类和第Ⅵ类分界线为下颌骨下层出现层面。经过分类模型分类后,每张CT图像均对应一个分类。然后,根据每个OARs所属的分类,将对应分类的CT图像提取出来并输入到勾画模型进行自动勾画。本研究中涉及的危及器官分类结果如下:脑干(第Ⅱ、Ⅲ和Ⅳ类),眼球(第Ⅲ类),视神经(第Ⅲ类),颞叶(第Ⅱ、Ⅲ和Ⅳ类),下颌骨(第Ⅳ和Ⅴ类),腮腺(第Ⅳ和Ⅴ类)。例如,勾画眼球时仅将属于第Ⅲ类的CT图像提取出来输入勾画模型;勾画下颌骨时将属于第Ⅳ和Ⅴ类的CT图像提取出来输入勾画模型。为避免分类结果的误差,在提取CT图像输入勾画模型时,会额外提取上下边界各5层CT图像[11],保证对应分类完整输入到勾画模型。
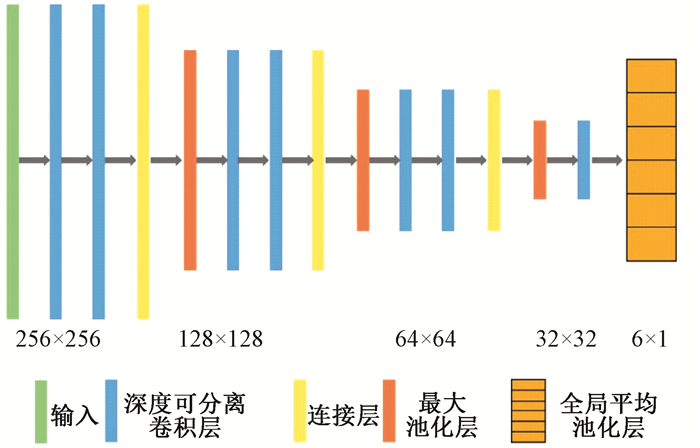
|
图 1 分类模型网络结构示意图 Figure 1 The architecture of the classification model |
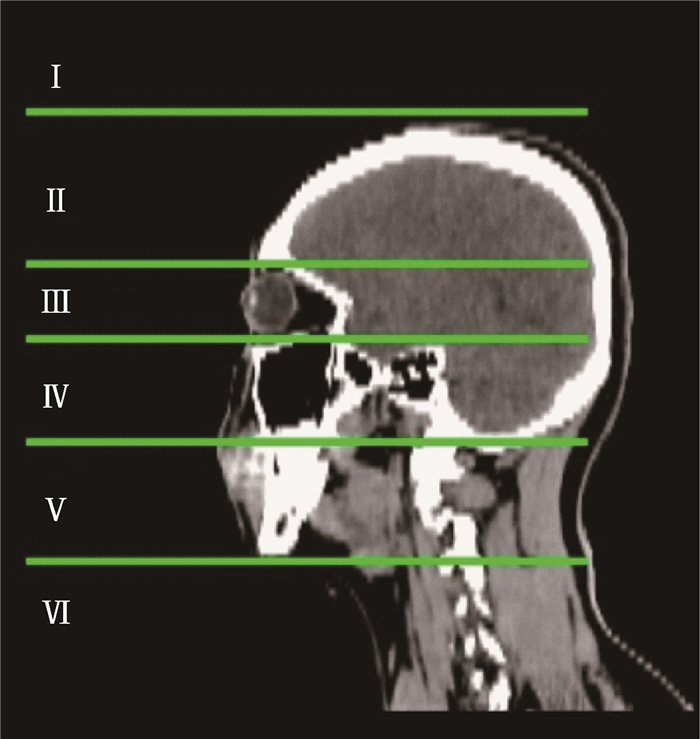
|
注:Ⅰ和Ⅱ分界线为颅骨出现层面;Ⅱ和Ⅲ分界线为眼球上层出现层面;Ⅲ和Ⅳ分界线为眼球下层出现层面;Ⅳ和Ⅴ分界线为小脑下层出现层面;Ⅴ和Ⅵ分界线为下颌骨下层出现层面 图 2 分类模型分类示意图 Figure 2 The illustration of the categories in classification model |
(3) 勾画模型:勾画模型选用的是3D U-Net模型。3D U-Net模型由下采样和上采样2个部分组成(图 3)。下采样包含7个膨胀卷积层(dilated convolution)[12]和3个最大池化层。上采样包含7个膨胀卷积层和3个反卷积层(deconvolution)。在下采样过程中,池化层为1×2×2,经过3次池化操作后,图像的平面分辨率变为原始值的1/8,同时z轴方向的分辨率保持不变。上采样过程则逐步将图像的分辨率恢复到原始值。此外,膨胀卷积层采用了4种不同的扩张率(扩张率分别为1、2、3、4),能够在不增加额外参数的情况下,扩大感受野,让每个卷积输出都包含较大范围的信息,捕获多尺度上下文信息。除了固有的下采样和上采样中的连接外,还增加了相邻前后两个膨胀卷积层之间的连接,将前后提取的特征合并以增加特征提取的丰富度。所有的卷积层都连接了具有自归一化的激活函数:缩放指数线性单元(scaled exponential linear unit,SELU)。Dice损失函数[13]和Adam[14]优化器用于模型训练,初始学习率为0.001,batch为32,echo为1 000。
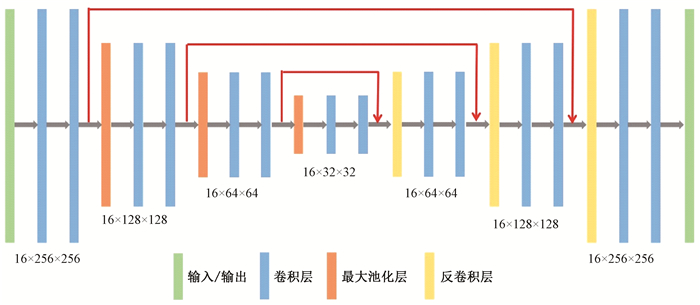
|
图 3 勾画模型(3D U-Net)网络结构示意图 Figure 3 The architecture of the segmentation model (3D U-Net) |
经过勾画模型处理后,使用二值法(阈值为0.5)得到危及器官的概率分布结果,保留最大连通域,然后利用形态学方法(如腐蚀和膨胀方法)对结果进行平滑处理,得到最终勾画结果。
3. Smart segmentation图谱库建立:本研究使用的Smart segmentation软件是Eclipse 13.6系统里内置的部分。其原理是将勾画好的患者图像导入软件建立图谱库,把每一例患者的相关临床信息(如肿瘤位置、分期等)输入软件,然后将所有器官结构与模板里器官结构一一对应。新病例输入后,软件通过图像配准,计算新病例和图谱库内所有病例之间的相似度,根据相似度结果,选择相似度最高的图谱库病例作为模板,对新病例进行自动勾画(图 4)。本研究中,将150例病例输入Smart segmentation软件用于建立自动勾画图谱库。
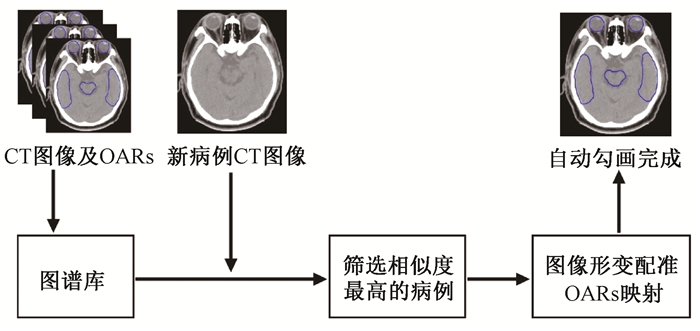
|
注:OARs.危及器官 图 4 Smart segmentation自动勾画流程图 Figure 4 The auto-segmentation flow chart of Smart segmentation |
4.结果分析:将基于深度学习的自动勾画模型的勾画结果与Smart segmentation的勾画结果进行比较,同时将结果与其他文献比较。以医生手动勾画结果为金标准,分别计算两种自动勾画方法的相似度系数(Dice similarity coefficient,DSC)[15]和豪斯多夫距离(Hausdorff distance,HD)[16]来评价自动勾画精度。
DSC计算如公式(1)所示:
| $ DSC = \frac{{2\left| {A \cap B} \right|}}{{\left| A \right| + \left| B \right|}} $ | (1) |
式中,A为金标准勾画结构体积;B为自动勾画结构体积;|A∩B|代表A和B的交集。DSC的取值范围是0~1,越接近1表示自动勾画和金标准重合的部分越多,勾画结果越好。
HD计算如公式(2)和(3)所示:
| $ HD\left( {A, B} \right) = {\rm{max}}\left[ {h\left( {A, B} \right), h\left( {B, A} \right)} \right] $ | (2) |
| $ h\left( {A, B} \right) = \mathop {{\rm{max}}}\limits_{a \in A} \mathop {{\rm{min}}}\limits_{b \in B} \left\| {a - b} \right\| $ | (3) |
式中,h(A, B)表示集合A中的点与集合B的距离最大值。HD的值越小,表示自动勾画和金标准越接近,勾画结果越好。
同时记录两种自动勾画方法勾画每例病例花费时间,比较不同勾画方法之间的效率差异。
5.统计学处理:采用SPSS 24.0软件进行统计学分析,使用Shapiro-Wilk方法检验数据是否满足正态分布。对正态分布数据用x±s表示,组间比较采用配对t检验;非正态分布数据用中位数和四分位数表示,组间比较采用Wilcoxon符号秩和检验。P < 0.05为差异有统计学意义。
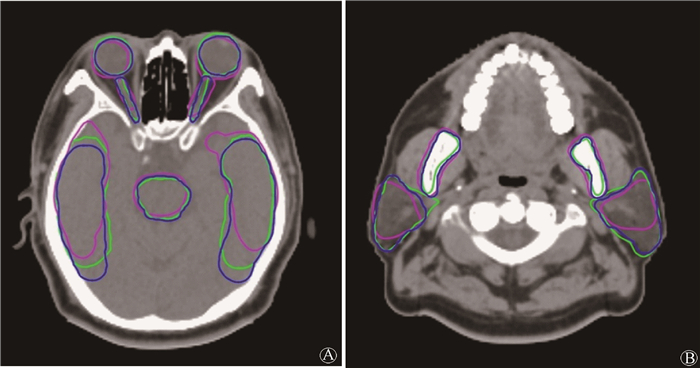
结果1.勾画精度:表 1列出两种自动勾画方法的DSC和HD结果。自动勾画模型的DSC范围为0.70~0.89,Smart segmentation的DSC范围为0.44~0.81。本研究中的10个结构中,自动勾画模型的DSC值均大于Smart segmentation,除左侧腮腺外,差异均有统计学意义(t=3.115~7.915,Z=-1.352~-3.783,P < 0.05)。自动勾画模型的HD范围为2.00~11.92 mm,Smart segmentation的HD范围为3.16~11.44 mm,除两侧腮腺外,自动勾画模型的HD值均小于Smart segmentation,差异均有统计学意义(Z=-1.521~-3.921,P < 0.05)。图 5展示了一例患者人工勾画以及使用两种方法自动勾画的结果。
|
|
表 1 两种自动勾画方法的相似度系数(DSC)和豪斯多夫距离(HD)结果 Table 1 The results of DSC and HD for the two auto-segmentation methods |
|
注:蓝色为人工勾画结果,绿色为基于深度学习的自动勾画模型勾画结果,紫色为Smart segmentation勾画结果 图 5 1例患者勾画结果 A.脑干、左眼球、右眼球、左视神经、右视神经、左颞叶和右颞叶;B.下颌骨、左腮腺和右腮腺 Figure 5 The auto-segmentation results of one patient A. brainstem, left eye, center eye, left optic nerve, center optic nerve, left temporal lobe and center temporal lobe; B. mandible, left parotid and center parotid |
2.勾画时间:基于深度学习的自动勾画模型勾画一例病例上述10个危及器官平均花费41.95 s,Smart segmentation软件平均花费86.10 s,差异有统计学意义(Z=-3.920,P < 0.05)。而医生人工勾画花费时间远远大于上述两种自动勾画方法,平均每例花费时间在20 min以上。
3.DSC比较:基于深度学习的自动勾画模型的勾画结果与其他文献比较结果见表 2。
|
|
表 2 相似度系数(DSC)结果比较 Table 2 The comparison of DSC results |
讨论
放疗过程中,OARs勾画是非常关键的步骤,该过程耗费临床医生大量时间和精力,并且勾画结果受临床医生业务水平影响而参差不齐。头颈部危及器官数量众多,特征各异,本研究选取部分危及器官作为研究对象(其他危及器官,如眼晶状体等,由于体积小且出现层面少,人工勾画较为容易,所以暂未纳入本研究),设计了基于深度学习的自动勾画模型对其进行自动勾画,并与自动勾画软件Smart segmentation进行比较。自动勾画模型是由分类模型和勾画模型共同组成,其中分类模型用于将CT图像从头脚方向分为6类,然后将每个结构对应的类别输入到勾画模型进行勾画。以眼球为例,仅需要将第Ⅲ类的CT图像输入到勾画模型中进行眼球勾画,这样能减少输入到勾画模型中的CT图像数量,避免冗余图像增加运算负担,同时也能避免其他层面出现的假阳性。
本研究的主要创新点是设计了分类模型,用于对CT图像分类。分类模型的分类数量为6类,共涉及5条分类界线。每条分类界线的选取均考虑了本研究涉及的危及器官解剖特点和位置特点。第Ⅰ类和第Ⅱ类以颅骨出现第一层作为分界线,可以将未包含任何患者解剖信息的第Ⅰ类所在位置的CT图像提取出去,且颅骨在CT图像上显示清晰,易于模型训练学习。第Ⅴ类和第Ⅵ类以下颌骨最下层面作为分界线,在该位置以下不包含本研究涉及的危及器官,可以将该分界线以下即第Ⅵ类包含的CT图像提取出去,且下颌骨在CT图像上显示清晰,易于模型训练学习。第Ⅳ类和第Ⅴ类以小脑最下层面作为分界线,将CT图像分为颅内和颅外,便于确定颅内和颅外危及器官的上下界。考虑到从颅顶到小脑最下层范围较大,选取眼球最上和最下层面分别作为第Ⅱ类和第Ⅲ类、第Ⅲ类和第Ⅳ类之间的分界线,以此将头部最为关键的视觉相关危及器官(眼球、视神经等)所在CT图像层面提取出来,便于后续勾画。分类结果发现,第Ⅰ类不包含任何器官,第Ⅱ类有脑干和颞叶的上缘出现,第Ⅲ类有脑干、眼球、视神经和颞叶出现,第Ⅳ类有脑干、颞叶、下颌骨和腮腺出现,第Ⅴ类有下颌骨和腮腺出现,第Ⅵ类不包含本研究涉及的危及器官。
Zijdenbos等[21]提出,当DSC>0.70时,表明两个结构重复度高,也就表明自动勾画效果较好。从本研究结果可以看出,本研究设计的自动勾画模型对脑干、眼球、视神经、颞叶、下颌骨和腮腺均能较好勾画,DSC值均≥0.70。除左视神经和双侧腮腺外,其余结构的DSC值均≥0.80。这是由于视神经体积比较小,在CT图像上显示不够清晰;腮腺是软组织,边界不易分辨[22]。其中,眼球和下颌骨的DSC值高达0.89,这是由于眼球形状较为规则便于识别,而下颌骨是骨性结构,在CT图像上便于识别[22]。HD的结果同样表明,眼球、下颌骨和脑干这些规则或骨性结构的结果,要优于腮腺和颞叶这些不规则的软组织结构。从整体上看,基于深度学习的自动勾画模型的勾画结果,明显优于Smart segmentation。除腮腺外,其余结构的DSC和HD结果,基于深度学习的自动勾画模型均优于基于图谱方法的Smart segmentation,且差异有统计学意义。两种自动勾画方法的结果,均与人工勾画有一定差异,需要临床医生进行修改后使用。同时,基于深度学习的自动勾画模型勾画速度比Smart segmentation软件快,效率提高了51.28%。
本研究与其他文献比较显示,视神经的勾画结果优于其他文献结果,脑干、眼球、颞叶和下颌骨的勾画结果与其他文献结果相近,而腮腺勾画结果比其他文献结果略差。分析主要原因是腮腺为软组织,边界不易分辨。本研究的不足之处在于,自动勾画是在CT图像上进行的,对一些软组织结构的勾画精度较低,后续研究可以和MR图像结合,提高勾画的准确性[23]。此外,也可以继续增加训练集数量,纳入更多特异性病例,从而增加深度学习模型的稳定性[20]。
本研究设计了基于深度学习的自动勾画模型,能够对头颈部OARs较为精准的勾画,勾画精度和速度优于基于图谱方法的Smart segmentation软件,能够减轻临床医生工作负担,但仍需要临床医生进行修改后使用。
志谢 本研究获得北京大学医-信交叉学科种子基金(BMU20160585)和北京大学第三医院临床重点项目(BYSY2018013)的资助
利益冲突 无
作者贡献声明 张书铭负责数据收集、数据准备、统计分析和论文撰写;杨瑞杰负责研究设计与指导,论文修改;朱森华负责模型训练指导;王皓和田素青负责结构勾画指导;张旭阳、李佳奇和雷润宏参与数据准备
| [1] |
Hawkins PG, Kadam AS, Jackson WC, et al. Organ-sparing in radiotherapy for head-and-neck cancer:improving quality of life[J]. Semin Radiat Oncol, 2018, 28(1): 46-52. DOI:10.1016/j.semradonc.2017.08.002 |
| [2] |
Harari PM, Song S, Tomé WA. Emphasizing conformal avoidance versus target definition for IMRT planning in head-and-neck cancer[J]. Int J Radiat Oncol Biol Phys, 2010, 77(3): 950-958. DOI:10.1016/j.ijrobp.2009.09.062 |
| [3] |
Lorenzen EL, Taylor CW, Maraldo M, et al. Inter-observer variation in delineation of the heart and left anterior descending coronary artery in radiotherapy for breast cancer:a multi-centre study from Denmark and the UK[J]. Radiother Oncol, 2013, 108(2): 254-258. DOI:10.1016/j.radonc.2013.06.025 |
| [4] |
Sharp G, Fritscher KD, Pekar V, et al. Vision 20/20:perspectives on automated image segmentation for radiotherapy[J]. Med Phys, 2014, 41(5): 50902. DOI:10.1118/1.4871620 |
| [5] |
Ibragimov B, Xing L. Segmentation of organs-at-risks in head and neck CT images using convolutional neural networks[J]. Med Phys, 2017, 44(2): 547-557. DOI:10.1002/mp.12045 |
| [6] |
门阔, 戴建荣. 利用深度反卷积神经网络自动勾画放疗危及器官[J]. 中国医学物理学杂志, 2018, 35(3): 256-259. Men K, Dai JR. Automatic segmentation of organs at risk in radiotherapy using deep deconvolutional neural network[J]. Chin J Med Phys, 2018, 35(3): 256-259. DOI:10.3969/j.issn.1005-202X.2018.03.002 |
| [7] |
La Macchia M, Fellin F, Amichetti M, et al. Systematic evaluation of three different commercial software solutions for automatic segmentation for adaptive therapy in head-and-neck, prostate and pleural cancer[J]. Radiat Oncol, 2012, 7: 160. DOI:10.1186/1748-717X-7-160 |
| [8] |
葛迦, 宁丽华, 严森祥, 等. 两种软件Smart Segmentation与MIM Atlas自动勾画鼻咽癌危及器官的准确性研究[J]. 中华放射医学与防护杂志, 2019, 39(9): 668-672. Ge J, Ning LH, Yan SX, et al. Automatic segmentation of organs at risk for nasopharyngeal carcinoma with Smart Segmentation and MIM Atlas[J]. Chin J Radiol Med Prot, 2019, 39(9): 668-672. DOI:10.3760/cma.jissn.0254-5098.2019.09.006 |
| [9] |
Brouwer CL, Steenbakkers RJ, Bourhis J, et al. CT-based delineation of organs at risk in the head and neck region:DAHANCA, EORTC, GORTEC, HKNPCSG, NCIC CTG, NCRI, NRG Oncology and TROG consensus guidelines[J]. Radiother Oncol, 2015, 117(1): 83-90. DOI:10.1016/j.radonc.2015.07.041 |
| [10] |
Chollet F. Xception: deep learning with depthwise separable convolutions[C]. Honolulu: Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 2017.
|
| [11] |
Men K, Geng H, Cheng C, et al. Technical note:more accurate and efficient segmentation of organs-at-risk in radiotherapy with convolutional neural networks cascades[J]. Med Phys, 2018, 46: 289-292. DOI:10.1002/mp.13296 |
| [12] |
Yu F, Koltun V. Multi-scale context aggregation by dilated convolutions[J]. 2015, arXiv: 1511.07122v2.
|
| [13] |
Sudre CH, Li W, Vercauteren T, et al. Generalised Dice overlap as a deep learning loss function for highly unbalanced segmentations[C].Quebec: Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support, 2017.
|
| [14] |
Kingma DP, Ba JL. Adam a method for stochastic optimization. 2014, arXiv: 1412.6980v9.
|
| [15] |
Crum WR, Camara O, Hill DLG. Generalized overlap measures for evaluation and validation in medical image analysis[J]. IEEE Trans Med Imaging, 2006, 25(11): 1451-1461. DOI:10.1109/TMI.2006.880587 |
| [16] |
Huttenlocher DP, Klanderman GA, Rucklidge WJ. Comparing images using the Hausdorff distance[J]. IEEE Trans Pattern Anal Mach Intell, 1993, 15: 850-863. DOI:10.1109/34.232073 |
| [17] |
Liang S, Tang F, Huang X, et al. Deep-learning-based detection and segmentation of organs at risk in nasopharyngeal carcinoma computed tomographic images for radiotherapy planning[J]. Eur Radiol, 2019, 29(4): 1961-1967. DOI:10.1007/s00330-018-5748-9 |
| [18] |
Tappeiner E, Pröll S, Hönig M, et al. Multi-organ segmentation of the head and neck area:an efficient hierarchical neural networks approach[J]. Int J Comput Assist Radiol Surg, 2019, 14(5): 745-754. DOI:10.1007/s11548-019-01922-4 |
| [19] |
Tong N, Gou S, Yang S, et al. Fully automatic multi-organ segmentation for head and neck cancer radiotherapy using shape representation model constrained fully convolutional neural networks[J]. Med Phys, 2018, 45(10): 4558-4567. DOI:10.1002/mp.13147 |
| [20] |
Zhu W, Huang Y, Zeng L, et al. AnatomyNet:deep learning for fast and fully automated whole-volume segmentation of head and neck anatomy[J]. Med Phys, 2019, 46: 576-589. DOI:10.1002/mp.13300 |
| [21] |
Zijdenbos AP, Dawant BM, Margolin RA, et al. Morphometric analysis of white matter lesions in MR images:method and validation[J]. IEEE Trans Med Imaging, 1994, 13(4): 716-724. DOI:10.1109/42.363096 |
| [22] |
Raudaschl PF, Zaffino P, Sharp GC, et al. Evaluation of segmentation methods on head and neck CT:auto-segmentation challenge 2015[J]. Med Phys, 2017, 44(5): 2020-2036. DOI:10.1002/mp.12197 |
| [23] |
Lin L, Dou Q, Jin Y, et al. Deep learning for automated contouring of primary tumor volumes by MRI for nasopharyngeal carcinoma[J]. Radiology, 2019, 291(3): 677-686. DOI:10.1148/radiol.2019182012 |
 2020, Vol. 40
2020, Vol. 40


