2. 常州工学院电子信息工程学院电子科学与技术系 213032;
3. 常州大学微电子与控制工程学院自动化系, 213164
2. Department of Electronic Science and Technology School of Electrical Information Engineering, Changzhou Institute of Technology, 213032, China;
3. Department of Automation, School of Microelectronics and Control Engineering, Changzhou University, 213164, China
医学图像在临床上有着重要的作用,可以辅助医生进行临床诊断和手术计划。医学图像具有多模态的优势,临床上主要采取的手段有:计算机体层成像(CT)图像、超声(ultrasound, US)图像、磁共振(magnetic resonance, MR)图像、正电子发射计算机断层显像(positron emission tomography, PET)等。CT图像是利用精确准直的X射线束对人体进行一定厚度的层面扫描得到的图像,具有高密度分辨率的特点,对人体软组织能够成像[1]。超声图像是利用超声探头向人体发射超声波束并按一定的方向进行扫描,通过监测反射信号的延迟时间及强弱,判断脏器距离体表的距离及性质,经过计算机处理后获得超声图像。超声成像对活动界面能作动态的实时显示,对小病灶有良好的显示能力,且超声图像的信息量丰富,具有灰阶的切面图像,层次清楚[2]。MR图像是根据强磁场中放射波和氢核的相互作用获得的,对于软组织的对比度更高,然而成像速度过慢且受患者活动影响较大; PET图像利用正电子核素标记葡萄糖等人体代谢物作为显像剂,可以提供疾病的生物代谢信息[1]。
放疗是治疗恶性肿瘤的重要手段之一,在放射治疗中,需要物理师根据CT图像进行治疗靶区的定位和勾画。然而,由于CT图像软组织分辨率不高且电子密度不准确,不利于靶区的精准定位与勾画。为了保证治疗的安全性和准确性,需要结合其他模态的图像如超声、MR和PET等,利用其呈现出的组织特点进行互补,以帮助医生更加准确的完成勾画[3]。然而,多模态数据的采集面临着很大的挑战。首先,多次检查会产生昂贵的检查费用;其次,多次图像采集过程中无法保证患者数据的一致性。因此,基于CT图像的跨模态转换得到了广泛的关注。
本文将回顾近年来基于CT图像的超声图像、MR图像、PET图像的跨模态转换研究,对研究方法进行总结,旨在提出未来的研究方向。
一、CT图像与超声图像的跨模态转换经过多年的努力,国内外学者提出了多种超声图像与CT图像的跨模态转换方法。CT图像与超声图像的跨模态转换主要采用基于人体组织声抗信息的转换方法,根据CT图像获得探查的人体内部组织的信息,利用这些组织的声抗信息建立分布模型,而后加入描述超声图像的特征信息以期获得超声图像。CT图像与超声图像的跨模态转换为基于数学模型的传统方法见表 1。
|
|
表 1 不同文献报道CT图像与超声图像的跨模态转换方法 Table 1 CT and ultrasounic image translation reported in literatures. |
人体组织的声抗信息为超声图像转换提供了理论依据。Bamber和Dickinson[4]提出了一种预测B超图像表现和性质的方法,利用频域特征生成组织模型,利用超声脉冲与波形进行B超图像的仿真。该模型实现简单,为CT图像与超声图像的跨模态转换提供了思路。基于Bamber&Dickinson模型,Dillenseger等[5]利用点扩散函数实现了CT图像到超声图像的跨模态转换。首先,利用CT扫描信息构建组织模型,然后在生成的超声图像中加入散斑特性。该模型计算效率较高,可以定性地反映超声图像内部换能器数目和频率的影响、散斑位置和形状、组织间的声学界面等特征。为了更好地表现超声图像的散斑特征,Tsui和Chang[6]等提出一种不受系统和操作者影响的定量方法来提供与生物组织性质相关的散射体信息,通过建立一个基于Nakagami参数图描述局部散射体浓度图像。该模型一方面生成不同系统增益和动态范围下的B超图像,另一方面确定满足局部Nakagami参数的稳定估计的窗口的最佳尺寸,以用于构造体内组织的分布图像。该模型可以与传统的B超扫描结合起来,同时显示组织中的结构和散射特性。然而,Nakagami的参数对转换超声图像影响很大,Larrue和Noble[7]发现在乳房上直接应用该模型生成的模拟超声图像与实际超声图像存在很大的偏差。这是因为不同组织内部的Nakagami分布的均值并不相同。他们还提出一种从肿瘤超声图像中提取模型参数的方法,通过分离位于肿瘤的3个区域:肿瘤与周围组织的边界、肿瘤内部的微观结构和剩余区域对Nakagami参数值进行确定。首先,对于每个区域计算形态学和定性参数,进而分析和量化肿瘤的形态参数和结构对参数统计值的影响。然后,对由混合分布引起的参数低估进行分析,并给出了这种现象的解析描述和数值例子。参数估计应考虑对肿瘤的各种参数和特征进行广泛的统计分析,以期获得更加准确的参数值。进一步地,Kutter等[8]基于射线模型进行CT图像到超声图像转换过程,同时基于图像处理单元(graphics processing unit, GPU)对生成过程进行了加速。该方法选取适合超声训练的CT图像中获得真实的超声模拟,提出了一种基于GPU的超声射线模拟框架。该框架能够适应不同复杂程度的超声模拟,并且速度足够快,能够生成用于训练目的的交互式模拟。同时,实现了图像转换过程的可视化。与基于中央处理单元(center processing unit, CPU)的算法相比,该方法在超声仿真以及CT和超声图像跨模态转换两个主要应用中的具有显著的加速效果(高达200倍)。Bürger等[9]利用卷积增强射线可追溯的方法基于可变网格模型实现了实时的CT到超声图像的转换过程,可以精确的对超声特有的伪影进行仿真。该方法基于Bamber & Dickinson模型,利用光线跟踪和卷积模型的组合的方法,实现了具有真实感的快速超声图像生成方法。该方法对超声特定的伪影,如折射和混响等,都可以进行正确的模拟,也可以适应超声图像中发生的组织变形。同时,该方法通过使用物理引擎的定义良好的接口,可以很容易地开展应用及图像实时传输。
得益于GPU的发展,Idzenga等[10]针对2D图像合成过程中存在的归一化交叉相关(normalized cross correlation, NCC)计算量大的问题,提出基于GPU进行超声图像的转换过程,对两种并行化方法进行了研究:多CPU的OpenMP接口和GPU的计算统一设备体系结构(compute unified device architecture, CUDA),对于两种方法的NCC计算进行了性能评估,两种并行化方法均未造成弹性图像质量的显著损失,GPU在计算NCC方面优于CPU方法。基于GPU可以减少计算NCC所消耗的时间,提高了运动估计的帧速率,进而实现一定范围内NCC的变形实时估计计算。
基于GPU平台设计的射线追踪技术可以对超声的多界面和多反射进行仿真,却忽略了界面间反射、折射的弥散效应。Mattausch和Goksel[11]提出利用Monte-Carlo采样技术应对射线追踪的不确定性对超声图像内复杂的信号进行仿真,仿真结果可以很好的体现超声图像中的伪影和模糊反射。为了满足所需的帧率,Szostek和Piókowski[12]提出一个考虑折射效果的波阵面构建的方法减弱超声波的散射和反射影响。波阵面构建利用了射线追踪和速率平均等信息。该方法可以实时实现CT图像到超声图像的跨模态转换。
基于人体组织声抗信息的超声图像跨模态转换已经取得了一些成果,然而目前的方法均是通过利用统计分布、射线追踪等人为制造散斑、伪影等信息,会导致转换的超声图像与真实采集的超声图像相比信噪比很低,且组织内部细节信息缺失等。同时,目前尚未开展基于深度学习的CT图像与超声图像的跨模态研究。
二、CT图像与MR图像的跨模态转换CT图像与MR图像作为疾病诊断的金标准,两个模态图像转换的研究至关重要。CT图像与MR图像的跨模态研究主要有两类方法:基于传统图像处理的方法与基于深度学习的方法。CT图像与MR图像的跨模态转换是利用MR图像自身的特征,如灰度、纹理、结构等信息,结果针对MR不同成像序列并无区别[13]。现从这两个方面进行介绍,CT图像与MR图像的跨模态转换方法见表 2。
|
|
表 2 不同文献报道CT图像与MR图像的跨模态转换方法 Table 2 CT and MRI translation reported in literatures |
1.基于传统的方法:在放疗的过程中,需要在计划阶段利用CT图像进行剂量勾画,治疗阶段利用术中的CT图像指导剂量。为了避免CT图像采集过程对患者造成额外的辐射,需利用MR图像跨模态转换生成CT图像。然而,MR图像内的灰度级与电子密度信息不是直接相关的。Berker等[14]提出将组织分成4类,并结合超短回声时间/Dixon进行衰减矫正。为了提高组织分割的精度,Zaidi等[15]提出对MR图像内组织进行分割细化。基于图谱的方法首先对图谱进行配准,然后对配准图谱的衰减图进行变形。基于学习的方法的核心是建立MR图像到CT图像的非线性映射模型。Jog等[16]利用随机森林(random forest, RF)的方法提高了MR图像的分辨率。Huynh等[17]将MR图像分成若干图像块,针对每个图像块应用结构随机森林的方法,实现了大脑、前列腺部位的MR图像到CT图像的跨模态转换。Andreasen等[18]提出基于局部纹理、边缘及空间特征的随机森林法,实现了骨盆MR图像与CT图像的跨模态转换。Yang等[19]在图像块随机森林的基础上提出,提取配准后的训练图像内部的解剖特征作为标志,在脑部MR图像上实现了CT图像的跨模态转换。进一步地,Lei等[20]提出迭代细化模型提高CT图像转换的精度,脑部MR图像与CT图像跨模态转换的平均绝对误差、峰值信噪比、归一化交叉指数分别可以达到(60.87±15.10)HU、(24.63±1.73)dB、0.954±0.0013;盆骨MR图像与CT图像的平均绝对误差、峰值信噪比、归一化交叉指数分别可以达到(29.86±10.4)HU、(34.18±3.31)dB、0.980±0.025。Largent等[21]对基于图像块的、基于图谱的以及基于容积密度的MR图像与CT图像的跨模态的方法在39名前列腺癌患者数据上进行了比较,结果表明基于图像块的方法优于其他两种,具有更好的临床显著性。Wang等[22]在14例患者的数据上进行了MR图像与CT图像的跨模态转换,并进一步实现了剂量预测。
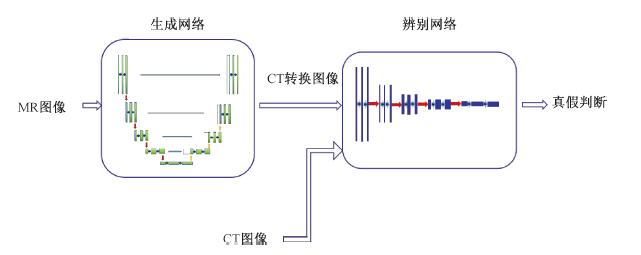
2.基于深度学习的方法:随着深度学习的火热发展,利用深度学习的跨模态图像转换得到了越来越多的关注。Arabi等[32]对比了6种转换方法,实验结果证明基于深度学习的转换方法具有更高的精度和敏感性。生成对抗网络生成对抗网络(generative adversarial networks, GANs)作为一种典型的网络结构,在医学图像跨模态转换上得到了广泛的应用,网络结构图如图 1所示[31]。由于生成对抗网络提出以来,引起了国内研究学者的极大重视[34-35]。基于GANs网络进行跨模态图像转换有两种途径:基于图像对和非图像对[23-36]。
|
图 1 MR图像与CT图像转换的生成对抗网络结构示意图 Figure 1 Schematic of generative adversarial networks for MRI and CT image translation |
(1) 基于图像对的跨模态图像转换方法:基于图像对的跨模态图像依赖于同一状态下的配准后的两种模态图像进行,在脑部、心脏等器官取得了一些研究成果。张杰等[35]对利用生成对抗时网络技术合成医学图像进行了总结和展望。Nie等[23]在GANs框架下提出cascade GANs,利用全卷积网络(fully convolutional network, FCN)实现了核磁图像(magnetic resonance, MR)图像到CT图像的转换,为了去除转换图像内的模糊边界现象,在损失函数内加入图像梯度差,同时利用长时残差单元帮助生成网络的训练;判别网络则使用自动上下文模型进行实现。该网络不仅可以实现脑部、骨盆的MR图像到CT图像的转换,而且实现了脑部3T MR图像到7T MR图像的转换。
Nie等[23]的方法需要依赖于同器官的MR图像和CT图像,考虑到只采集MR图像的情况。而Emami等[24]使用条件生成对抗网络(conditional generative adversarial networks, cGANs), 残差网络(residual network, ResNet)作为生成网络,使用5层卷积神经网络(convolution neural network, CNN)作为判别网络,利用15个患者的数据,实现了脑部MR图像到CT图像的转换,以满足只使用MR图像做手术计划的情况。实验结果表明,该方法在仿真CT(synthetic computerized tomography, synCTs)上的性能优于CNN,为支持MR-only工作流提供了强大的潜力。同样的,考虑MR进行计划的情况下,Maspero等[25]利用cGAN网络进行训练,利用91个患者的前列腺、18个患者的直肠及14个患者的子宫颈MR图像进行了验证,转换得到的CT图像与实际采集的CT图像之间的剂量分布点误差可以达到±2.5%。然而,在采集多模态图像对的过程中,需要保证患者在同一体位下,这给采集过程带来了很大的困难。同时,基于图像对的方法依赖于医生的标注,标注的精度和偏差对转换效果影响很大。为克服以上问题,基于非图像对的方法引起了学者们的关注。
(2) 基于非图像对跨模态图像转换方法:为了克服图像对方法中存在的问题,基于非图像对的跨模态转换应运而生。Chartsias等[26]基于循环生成对抗网络(cycle generative adversarial networks, cycleGAN)架构实现了心脏2D MR图像与CT图像的跨模态转换,然而由于实验没有金标准图像的存在,所以导致无法对转换图像的质量进行评价。进一步地,Zhang等[27]基于cycleGAN架构实现了心脏的3D MRI图像和CT图像的转换过程,生成器由端到端的3D CNN组成,损失函数分别由对抗误差、循环一次性误差和形状一致性误差构成。该网络不仅能够实现图像转换,同时可以实现医学分割。Wolterink等[28]基于cycleGAN在核磁引导的放疗过程的核磁图像和CT图像的配准,利用非配对的核磁和CT图像进行图像合成,与实际采集的图像相比,MAE可以达到73.7%,信噪比达到32.3%。为了考虑组织结构的相似性,Yang等[29]在cycleGAN网络上额外加入一个损失函数用来评估图像之间组织结构的一致性,同时利用基于位置的选择机制进行训练图像的选择,实现了2D MR图像到CT图像的转换。该网络提出了一种结构约束的脑部MR-To-CT合成循环,该循环利用未配对数据定义额外的结构一致性损失,并基于与模态无关的邻域描述符来约束结构一致性。同时,使用基于位置的选择策略来选择训练图像,而不是完全随机的选择方案, 其结果表明,所提出的选择策略产生了更高的结构相似性(structural similarity, SSIM)得分(P<0.05)。Huo等[30]提出了一个端到端的综合分割网络(synthetic segmentation network, SynSeg-Net)进行脾脏MR图像至CT图像的转换,利用了cycleGAN和DCNN的优势,可以直接利用MR图像的标记完成对CT图像的分割。该网络不需要在目标成像模式上进行人工标注。该网络由未配对的源和目标模态图像训练,仅对源模态进行手动分割。该方法利用跨模态图像综合学习的优点,减轻了医学图像分析中人工分割的工作量。Yu等[31]基于cGAN在生成网络加入组织的边缘信息、在判别网络中加入边缘判别项,以丰富合成CT图像内部的组织结构信息。该网络利用了分层特征,可以用于3D CT图像的合成,提出了边缘感知的生成性对抗网络(edge aware generative adversarial networks, Ea-GAN),以进一步克服现有医学图像合成cGAN模型中存在的切片不连续和合成不太尖锐的问题。该网络可以提取体素强度和图像结构信息。为了获取图像结构,该网络提取包含关键纹理信息的边缘进行视觉识别,并将边缘映射与cGAN模型进行集成以提高转换质量。网络由两个框架组成,生成器诱导gEa-GAN(generative edge aware generative adversarial networks, gEa-GAN)和更高级的判别器诱导dEa-GAN(discriminative edge aware generative adversarial networks, dEa-GAN)。gEa-GAN模型用来提取合成和真实目标形态图像的Sobel边缘图,并在生成器训练过程中使距离最小化;dEa-GAN通过生成器和判别器之间的对抗学习进一步加强了这种边缘相似性。实验结果充分证明了图像跨模态转换过程中感知边缘细节的重要性。
三、CT图像与PET图像的跨模态转换CT图像与PET图像的跨模态转换研究得到了研究者的关注,研究热点主要是利用深度学习网络进行实现。为了丰富训练集,Bi等[36]提出多频段生成对抗网络(Multi-channel Generative Adversarial Networks, M-GANs)利用50幅肺部CT图像进行了PET图像的跨模态转换。与传统的GAN方法相比,该模型具有基于对抗学习概念的高层语义信息的特征表示能力。Ben-Cohen等[37]利用FCN与cGANs作为生成网络,使用23幅图像做训练集,实现了肝脏部位的CT图像到PET图像的跨模态转换。该方法将平均错误率从2.9降低到2.1。Armanious等[38]提出医学图像转换网络(Medical Image translation using Generative Adversarial Networks, MedGAN)实现PET图像到CT图像的转换,在损失函数中加入高频/低频的成分,利用编码-解码架构提高物体边界的尖锐度的堆叠网络(cascade network, CasNet)作为生成网络,利用判别网络作为特征提取器用来衡量转换图像与期望图像的像素和感知差异。
综上所述,CT图像与超声图像、MR图像、PET图像的跨模态转换已经取得了一些进展,但仍有待进一步开展研究。CT图像与超声图像的跨模态转换领域多利用传统算法进行实现,但基于深度学习的方法仍是空白。CT图像与MR图像的跨模态转换在传统算法上和深度学习算法上都取得了很好的成果。基于GANs网络进行跨模态转换更是发展迅速,不仅实现了跨模态之间的单向转换,而且实现了跨模态之间的双向转换。但是,目前的应用部位多为脑部和盆骨,针对其他部位的研究,如肝脏、肺部,仍有待进一步充实。
利益冲突 无
志谢 感谢常州市医学物理重点实验室项目(CM20193005)对本研究的赞助
作者贡献声明 毕卉负责文献阅读、分析及论文撰写;姜一波指导论文写作、参与论文修改;张琦、眭建峰、陆正大负责文献收集;倪昕晔负责论文审阅
| [1] |
张晓康, 张卫萍. 医学影像成像原理[M]. 北京: 人民卫生出版社, 2014. Zhang XK, Zhang WP. Principles of medical imaging[M]. Beijing: People's Medical Publishing House, 2014. |
| [2] |
万明习. 生物医学超声学[M]. 北京: 科学出版社, 2010. Wan MX. Biomedical ultrasound[M]. Beijing: Science Press, 2010. |
| [3] |
Jiao D, Li WS, Lu K, et al. An overview of multi-modal medical image fusion[J]. Neurocomputing, 2016, 215: 3-20. DOI:10.1016/j.neucom.2015.07.160 |
| [4] |
Bamber JC, Dickinson RJ. Ultrasonic B-scanning:a computer simulation[J]. Phys Med Biol, 1980, 25(3): 463. DOI:10.1007/978-3-540-72907-5_47 |
| [5] |
Dillenseger JL, Laguitton S, Delabrousse É. Fast simulation of ultrasound images from a CT volume[J]. Comput Biol Med, 2009, 39(2): 180-186. DOI:10.1016/j.compbiomed.2008.12.009 |
| [6] |
Tsui PH, Chang CC. Imaging local scatterer concentrations by the Nakagami statistical model[J]. Ultrasound Med Biol, 2007, 33(4): 608-619. DOI:10.1016/j.ultrasmedbio.2006.10.005 |
| [7] |
Larrue A, Noble J A. Modeling of errors in Nakagami imaging:illustration on breast mass characterization[J]. Ultrasound Med Biol, 2014, 40(5): 917-930. DOI:10.1016/j.ultrasmedbio.2013.11.018 |
| [8] |
Kutter O, Shams R, Navab N. Visualization and GPU-accelerated simulation of medical ultrasound from CT images[J]. Comput Meth Prog Bio, 2009, 94(3): 250-266. DOI:10.1016/j.cmpb.2008.12.011 |
| [9] |
Bürger B, Bettinghausen S, Rädle M, et al. Real-time GPU-based ultrasound simulation using deformable mesh models[J]. IEEE Trans Med Imaging, 2012, 32(3): 609-618. DOI:10.1109/TMI.2012.2234474 |
| [10] |
Idzenga T, Gaburov E, Vermin W, et al. Fast 2-D ultrasound strain imaging:The benefits of using a GPU[J]. IEEE Trans Ultrason Ferr, 2014, 61(1): 207-213. DOI:10.1109/TUFFC.2014.6689790 |
| [11] |
Mattausch O, Goksel O. Monte-Carlo ray-tracing for realistic interactive ultrasound simulation[C]. Eurographics Workshop on Visual Computing for Biology and Medicine, Bergen, NORWAY, Eurographics Association, 2016: 173-181. DOI: 10.2312/vcbm.20161285.
|
| [12] |
Szostek K, Piórkowski A. Real-time simulation of ultrasound refraction phenomena using ray-trace based wavefront construction method[J]. Comput Meth Prog Bio, 2016, 135: 187-197. DOI:10.1016/j.cmpb.2016.07.034 |
| [13] |
Edmund J M, Nyholm T. A review of substitute CT generation for MRI-only radiation therapy[J]. Radiat Oncol, 2017, 12(1): 28. DOI:10.1186/s13014-016-0747-y |
| [14] |
Berker Y, Franke J, Salomon A, et al. MRI-based attenuation correction for hybrid PET/MRI systems:a 4-class tissue segmentation technique using a combined ultrashort-echo-time/Dixon MRI sequence[J]. J Nucl Med, 2012, 53(5): 796-804. DOI:10.2967/jnumed.111.092577 |
| [15] |
Zaidi H, Montandon ML, Slosman DO. Magnetic resonance imaging-guided attenuation and scatter corrections in three-dimensional brain positron emission tomography[J]. Med Phys, 2003, 30(5): 937-948. DOI:10.1118/1.1569270 |
| [16] |
Jog A, Carass A, Prince JL. Improving magnetic resonance resolution with supervised learning[C]. 2014 IEEE 11th International Symposium on Biomedical Imaging, Beijing, China, IEEE, 2014, 2014: 987-990. DOI: 10.1109/ISBI.2014.6868038.
|
| [17] |
Huynh T, Gao Y, Kang J, et al. Estimating CT image from MRI data using structured random forest and auto-context model[J]. IEEE Trans Med Imaging, 2015, 35(1): 174-183. DOI:10.1109/TMI.2015.2461533 |
| [18] |
Andreasen D, Edmund JM, Zografos V, et al. Computed tomography synthesis from magnetic resonance images in the pelvis using multiple random forests and auto-context features[C]. Medical Imaging 2016: Image Processing, San Diego, CA, SPIE, 2016, 9784: 978417. DOI: 10.1117/12.2216924.
|
| [19] |
Yang X, Lei Y, Shu H K, et al. Pseudo CT estimation from MRI using patch-based random forest[C]. Medical Imaging 2017: Image Processing, Orlando, FL, United states, Proc SPIE Int Soc Opt Eng, 2017, 10133: 101332Q. DOI: 10.1117/12.2253936.
|
| [20] |
Lei Y, Jeong J J, Wang T, et al. MRI-based pseudo CT synthesis using anatomical signature and alternating random forest with iterative refinement model[J]. J Med Imaging, 2018, 5(4): 043504. DOI:10.1117/1.JMI.5.4.043504 |
| [21] |
Largent A, Barateau A, Nunes J, et al. Pseudo-CT generation for MRI-only radiation therapy treatment planning:comparison among patch-based, atlas-based, and bulk density methods[J]. Int J Radiat Oncol Biol Phys, 103(2): 479-490. DOI:10.1016/j.ijrobp.2018.10.002 |
| [22] |
Wang T, Manohar N, Lei Y, et al. MRI-based treatment planning for brain stereotactic radiosurgery:dosimetric validation of a learning-based pseudo-CT generation method[J]. Med Dosim, 2019, 44(3): 199-204. DOI:10.1016/j.meddos.2018.06.008 |
| [23] |
Nie D, Trullo R, Lian J, et al. Medical image synthesis with context-aware generative adversarial networks[C]. 20th International Conference on Medical Image Computing and Computer-Assisted Intervention, Quebec City, QC, Canada, Springer, 2017: 417-425. DOI: 10.1007/978-3-319-66179-7_48.
|
| [24] |
Emami H, Dong M, Nejad-Davarani SP, et al. Generating synthetic CTs from magnetic resonance images using generative adversarial networks[J]. Med Phys, 2018, 45(8): 3627-3636. DOI:10.1002/mp.13047 |
| [25] |
Maspero M, Savenije M, Dinkla A M, et al. Dose evaluation of fast synthetic-CT generation using a generative adversarial network for general pelvis MR-only radiotherapy[J]. Phys Med Biol, 2018, 63(18): 185001. DOI:10.1088/1361-6560/aada6d |
| [26] |
Chartsias A, Joyce T, Dharmakumar R, et al. Adversarial image synthesis for unpaired multi-modal cardiac data[C]. 2nd International Workshop on Simulation and Synthesis in Medical Imaging. Quebec City, Canada: Springer, 2017: 3-13. DOI: 10.1007/978-3-319-68127-6_1.
|
| [27] |
Zhang Z, Yang L, Zheng Y. Translating and segmenting multimodal medical volumes with cycle-and shape-consistency generative adversarial network[C]. 31th IEEE Conference on CVPR. Salt Lake City, UT: IEEE, 2018: 9242-9251. DOI: 10.1109/CVPR.2018.00963.
|
| [28] |
Wolterink JM, Dinkla AM, Savenije MHF, et al. Deep MR to CT synthesis using unpaired data[C]. 2nd International Workshop on Simulation and Synthesis in Medical Imaging. Quebec City, Canada: Springer, 2017: 14-23. DOI: 10.1007/978-3-319-68127-6_2.
|
| [29] |
Yang H, Sun J, Carass A, et al. Unpaired brain MR-to-CT synthesis using a structure-constrained CycleGAN[C]. 4th International Workshop on DLMIA. Granada, Spain: Springer, 2018: 174-182. DOI: 10.1007/978-3-030-00889-5_20.
|
| [30] |
Huo Y, Xu Z, Moon H, et al. Synseg-net:Synthetic segmentation without target modality ground truth[J]. IEEE Trans Med Imaging, 2018, 38(4): 1016-1025. DOI:10.1109/TMI.2018.2876633 |
| [31] |
Yu B, Zhou L, Wang L, et al. Ea-GANs:edge-aware generative adversarial networks for cross-modality MR image synthesis[J]. IEEE Trans Med Imaging, 2019, 38(7): 1750-1762. DOI:10.1109/TMI.2019.2895894 |
| [32] |
Arabi H, Dowling JA, Burgos N, et al. Comparative study of algorithms for synthetic CT generation from MRI:Consequences for MRI-guided radiation planning in the pelvic region[J]. Med Phys, 2018, 45(11): 5218-5233. DOI:10.1002/mp.13187 |
| [33] |
Yi X, Walia E, Babyn P. Generative adversarial network in medical imaging:a review[J]. Med Image Anal, 2019, 58: 101552. DOI:10.1016/j.media.2019.101552 |
| [34] |
王坤峰, 苟超, 段艳杰, 等. 生成式对抗网络GAN的研究进展与展望[J]. 自动化学报, 2017, 43(3): 321-332. Wang KF, Gou C, Duan YJ, et al. Generative adversarial networks:the state of the art and beyond[J]. Acta Automatica Sinica, 2017, 43(3): 321-332. DOI:10.16383/j.aas.2017.y000003 |
| [35] |
张杰, 赵惠军, 李贤威, 等. 基于生成式对抗网络技术合成医学图像的研究进展与展望[J]. 医疗卫生装备, 2019, 40(10): 93-98. Zhang J, Zhao HJ, Li XW, et al. Progress and prospects of medical images synthesis based on generative adversarial networks[J]. Chin Med Equip J, 2019, 40(10): 93-98. DOI:10.19745/j.1003-8868.2019257 |
| [36] |
Bi L, Kim J, Kumar A, et al. Synthesis of positron emission tomography (PET) images via multi-channel generative adversarial networks (GANs)[C]. 1st Int Workshop on CMMI. Quebec City, Canada: Springer, 2017: 43-51. DOI: 10.1007/978-3-319-67564-0_5.
|
| [37] |
Ben-Cohen A, Klang E, Raskin SP, et al. Cross-modality synthesis from CT to PET using FCN and GAN networks for improved automated lesion detection[J]. Eng Appl Artif Intel, 2019, 78: 186-194. DOI:10.1016/j.engappai.2018.11.013 |
| [38] |
Armanious K, Jiang C, Fischer M, et al. MedGAN:medical image translation using GANs[J]. Comput Med Imaging Graph, 2020, 79: 101684. DOI:10.1016/j.compmedimag.2019.101684 |
 2020, Vol. 40
2020, Vol. 40


