2. 军事科学院军事医学研究院辐射医学研究所, 北京 100850
2. Institute of Radiation Medicine, Academy of Military Medical Sciences, Academy of Military Sciences, Beijing 100850, China
电离辐射靶向损伤细胞核DNA,严重可造成细胞死亡[1]。电离辐射能诱导大量分子表达,这些分子在细胞周期调控、细胞凋亡、DNA损伤修复等过程发挥重要作用。现已有辐射诱导mRNA (ATM,Egr-1)、miRNA、snoRNA参与辐射损伤修复过程的报道,尚缺乏长链非编码RNA(long non-coding RNA, lncRNA)相关研究。lncRNA是一类长度>200 nt且无蛋白编码功能的RNA,具有表达丰度低,物种及细胞类型保守性低等特点[2],已被发现参与细胞周期、凋亡、免疫应答等重要生物学过程[3]。基因芯片作为高通量测序手段之一,可用于筛选辐射诱导新分子,为探索辐射诱导分子在辐射相应过程的作用机制提供新的策略[4]。本研究运用基因芯片检测辐射诱导LOC102606465下游靶基因变化情况,通过生物信息学分析靶基因潜在生物学功能,为揭示LOC102606465生物学功能提供理论依据。
材料与方法1.细胞株及实验材料:人宫颈癌HeLa细胞系购自中国医学科学院基础医学研究所。RPMI1640培养基(美国GIBCO公司),胎牛血清和细胞培养抗生素(美国Hyclone公司),TRIzol(美国Sigma公司),Lipofectamine® 2000(美国Invitrogen公司)cDNA反转录试剂盒(日本TaKaRa公司),Prime ViewTM基因芯片(美国Affymetrix公司),qRT-PCR试剂(美国Bio-Rad公司),实验所用引物由北京擎科生物有限公司合成,序列见表 1。
|
|
表 1 基因实时荧光定量PCR引物 Table 1 The primers for qRT-PCR analysis of the genes |
2.细胞培养:HeLa细胞于含10%胎牛血清、1%双抗的RPMI 1640培养基培养,放置于37℃、5%CO2培养箱。
3.细胞转染:细胞转染前1天铺匀至6孔板,待细胞融合度达到70%~80%开始转染。用无血清RPMI 1640培养基分别稀释siRNA及Lipofectamine®2000,室温静置5 min后将稀释的Lipofectamine®2000逐滴加入siRNA,混匀后室温静置15 min,最后将转染混合液逐孔加入,转染后8 h更换新鲜完全培养基。
4. RNA提取及芯片检测:1 ml TRIzol裂解细胞,按照试剂说明书提取细胞总RNA。无DNA/RNAse纯水溶解稀释RNA,紫外分光光度计测量RNA浓度,为确保样品符合PrimerviewTM芯片实验要求,RNA总量>5 μg,A280/260值>2.0。基因芯片检测委托北京普瑞麦迪生物公司辅助完成,主要包括芯片制备、样品制备、杂交反应、信号检测等步骤。
5. cDNA反转录及qRT-PCR验证:按照cDNA反转录试剂盒(TaKaRa)操作使用说明书,反转录RNA为cDNA,采用iTaqTM Universal SYBR® Green Supermix (美国Bio-Rad公司)试剂盒,qRT-PCR扩增仪(美国Bio-Rad公司,CFX96)扩增cDNA,β-肌动蛋白作为内参基因,2-ΔΔCt法计算基因相对表达量。
6.生物信息学分析:Affy包读取芯片原始数据,稳健的多阵列均值(RMA)方法对原始数据进行背景矫正、标准化以及表达量计算预处理。R语言的Limma函数包[5]对基因芯片进行差异表达分析,若基因变化倍数(fold change,FC)>1.1,P < 0.05认为是DEGs。通过维恩图的方法[6]合并两组共同变化的DEGs,用于后续研究。采用Clusterprofilter函数包[7]及DAVID数据库(https://david.ncifcrf.gov/)对共变化的DEGs分别进行GO和KEGG富集分析,ggplot2函数包[8]进行可视化,P < 0.05作为筛选显著性GO terms和信号通路的阈值。STRING数据库(https://string-db.org/)用以构建蛋白互作网络(protein protein interaction network,PPI network)并查找潜在的基因间关系,数据导入Cytoscape软件进行可视化。通过Cytoscape中MCODE程序[9]寻找PPI网络重要的功能模块,CytoHubba程序[10]用于识别PPI网络中枢纽基因,采用Closeness、Betweeness以及Degree算法获得关键枢纽基因。
7.统计学处理:运用SPSS 20.0软件进行统计分析,数据用x±s表示,若数据服从正态分布且方差齐性,两组间采用两独立样本t检验;若方差不齐,则采用两独立样本矫正t检验。P < 0.05为差异有统计学意义。
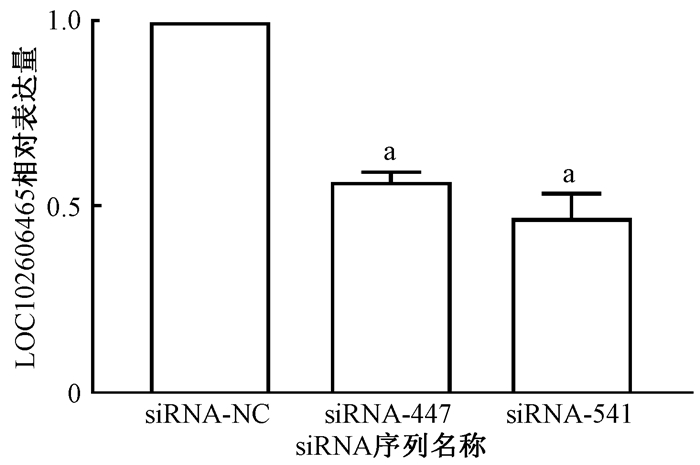
结果1. siRNA敲低效率的验证:采用siRNA-447和siRNA-541敲低LOC102606465,以siRNA-NC为对照,qRT-PCR检测表达量变化情况,结果发现siRNA-447、siRNA-541的LOC102606465表达量显著低于siRNA-NC(t=29.095、13.751,P < 0.01),结果见图 1。
|
注:与siRNA-NC比较,at=29.095、13.751,P < 0.01 图 1 siRNA敲低效率的验证 Figure 1 qRT-PCR Validation of siRNA knockdown efficiency |
2.靶基因表达谱变化:通过芯片筛选LOC102606465靶基因,结果发现siRNA-447、siRNA-541下调LOC102606465表达,均能引起靶基因表达谱发生变化,见图 2。同时发现siRNA-447、siRNA-541有374个共同变化的DEGs,其中有112个上调DEGs,262个下调DEGs。

|
注:红色为上调,蓝色为下调 图 2 差异表达基因热图A.siRNA-447与siRNA-NC相比;B.siRNA-541与siRNA-NC相比 Figure 2 The heatmap of DEGs A.siRNA-447 compare to siRNA-NC; B.siRNA-541 compare to siRNA-NC |
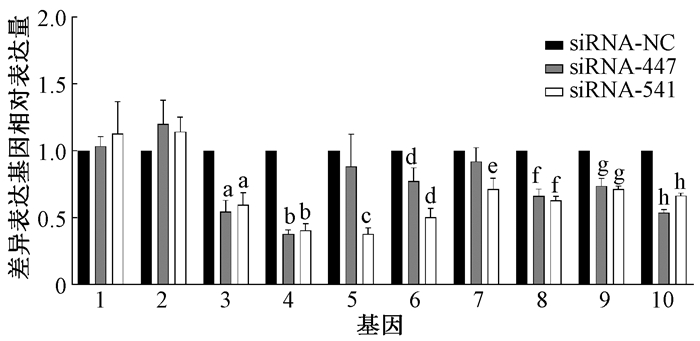
3. DEGs的qRT-PCR验证:随机选择10个DEGs进行qRT-PCR验证。结果发现,DEGs表达情况与基因芯片结果变化趋势基本一致,且AKR1C4、DPEP1、CDKN1A、SERPIN35、AKR1C1、SPINK1在siRNA-447、siRNA-S41的表达量低于siRNA-NC, 差异有统计学意义(t=3.943~34.166,P < 0.05),见图 3。
|
注:1. RAC1;2. PLCB4;3. AKR1C4;4. DPEP1;5.STEAP3;6.CDKN1A;7.GADD45B;8.SERPIN35;9.AKR1C1;10.SPINK1。与同基因siRNA-NC组相比,at=9.491、9.706,P < 0.05;bt=34.181、21.019,P < 0.01;ct=24.057,P < 0.05;dt=3.943、13.312,P < 0.05;et=6.143,P < 0.01;ft=11.847、21.88,P < 0.01;gt=8.039、21.477,P < 0.01;ht=34.166、29.436,P < 0.01 图 3 qRT-PCR验证LOC102606465表达下调后DEGs的表达变化 Figure 3 qRT-PCR confirmation of the expression of DEGs after downregulation of LOC102606465 |
4.GO功能富集分析:GO功能富集分析发现,下调DEGs显著富集于“氧化还原酶活性作用集群”(分子功能),“基底层”(细胞组分),“铵离子代谢过程”(生物过程);上调DEGs显著富集于“蛋白磷酸酶抑制剂活性”(分子功能),“SNARE复合体”(细胞组分),“纤维蛋白溶解负调节”(生物过程)。GO分析结果见表 2。
|
|
表 2 差异表达基因的GO分析(前15位) Table 2 GO enrichment analysis of DEGs(TOP 15) |
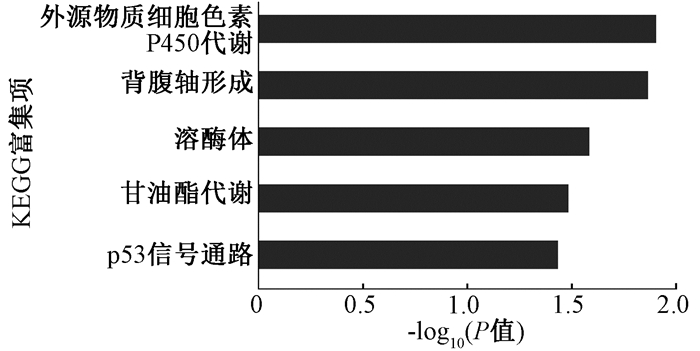
5.KEGG信号通路富集分析:通过DAVID数据库对DEGs进行KEGG分析。结果发现,共同变化DEGs显著富集于外源物质细胞色素P450代谢通路,背腹轴形成信号通路,溶酶体信号通路,甘油酯代谢通路和P53信号通路,见图 4。
|
图 4 KEGG分析差异表达基因的功能信号通路 Figure 4 KEGG analysis of the DEGs functional pathways |
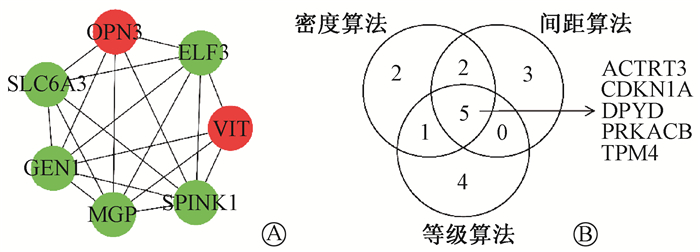
6.PPI蛋白间互作关系网络图及关键模块、枢纽基因的筛选:为发现DEGs相互作用关系,基于STRING数据库构建蛋白互作网络,所获PPI网络由194个节点与268个边缘组成。通过Cytoscape软件中的MCODE程序从PPI网络筛选出重要的功能模块(图 5), CytoHubba程序的3种算法筛选出排名前十的关键枢纽基因,数据取交集。结果发现,ACTRT3、CDKN1A、DPYD、PRKACB、TPM4为稳定的枢纽基因,见图 5。
|
注:红色表示表达上调,绿色表示表达下调 图 5 PPI网络关键功能模块与枢纽基因的筛选A.显著性功能模块;B.关键枢纽基因 Figure 5 Construction of key functional module and hub genes of PPI network A. The top functional module; B. The key hub genes |
讨论
电离辐射能靶向细胞中的DNA并通过多种形式造成其不同程度的损伤[11]。为维持基因组稳定性,防止异常遗传信息的传递,细胞可以响应电离辐射并激活DNA损伤修复过程,主要包括DNA损伤位点识别、DNA损伤阶段性评估以及DNA损伤修复等[12]。有研究表明,电离辐射通过辐解细胞内水分子产生的大量自由基或损伤细胞核DNA[13],并以此为信号,通过转录、转录后调控机制,诱导大量基因表达后参与细胞周期、细胞凋亡等重要生理过程[14]。所以,深入研究辐射诱导分子表达变化和规律,将为放射生物学领域提供新的研究思路。近年来,lncRNA作为非编码RNA研究重点,已被证实参与人类众多疾病的发生发展过程,如肿瘤、免疫和感染、自身免疫性疾病等,具有重要的生物学功能。前期研究发现,LOC102606465是辐射诱导表达的lncRNA,其功能少见报道,本研究基于基因芯片探索其潜在生物学功能。
基因芯片检测LOC102606465敲低后相关基因变化情况,结果发现,siRNA-447和siRNA-541干扰序列有374个共同变化的DEGs,其中有112个上调DEGs,262个下调DEGs。为进一步发现这些DEGs的相关功能及可能参与的信号通路,本研究用GO、KEGG富集分析进行功能注释,GO富集分析发现,下调DEGs显著富集于“氧化还原酶活性作用集群”(分子功能),“基底层”(细胞组分),“铵离子代谢过程”(生物过程);上调DEGs显著富集于“蛋白磷酸酶抑制剂活性”(分子功能),“SNARE复合体”(细胞组分),“纤维蛋白溶解负调节”(生物过程);KEGG信号通路富集分析发现,DEGs显著富集于外源物质细胞色素P450代谢通路,背腹轴形成信号通路,溶酶体信号通路,甘油酯代谢通路和P53信号通路,提示LOC102606465可能通过代谢信号通路、P53相关信号通路发挥其作用。
本研究构建PPI网络并筛选出关键枢纽基因(TPM4、CDKN1A、ACTRT3、DPYD、PRKACB)。TPM4主要参与骨骼肌和平滑肌细胞的收缩或维持细胞骨架在非肌肉细胞中的稳定性[15],其异常表达对乳腺癌、肺癌、前列腺癌等癌症的发生、发展起着重要作用[16],并且可作为结直肠癌患者预后指标[17]。CDKN1A能在细胞周期过程中调节细胞增殖以维持基因组稳定性,从而响应DNA损伤效应[18]。有研究发现,lncRNA PANDAR能通过与p53蛋白竞争结合的方式调控CDKN1A转录,对胃癌细胞生长进行调节[19]。ACTRT3可保护遗传物质免受物理和化学损害[20]。Lin等[21]研究发现,LINC00261通过介导甲基化依赖抑制DPYD以增加人食管癌细胞对5-FU的敏感性,发挥其抗肿瘤的能力。PRKACB编码蛋白是cAMP依赖性蛋白激酶的催化亚基,可通过cAMP介导参与细胞增殖、分化等过程[22]。目前,鲜有LOC102606465与TPM4、CDKN1A、ACTRT3、DPYD、PRKACB相互作用的文献报道,由于lncRNA可通过多种方式与基因结合并发挥生物学功能,故推测LOC102606465可能与枢纽基因相互作用,并在电离辐射响应过程中发挥重要作用。
采用qRT-PCR验证随机选择DEGs表达情况。结果发现,DEGs与基因芯片变化趋势基本一致,提示基因芯片结果具有可靠性。此外,筛选验证的DEGs将为后续研究LOC102606465的功能打下坚实基础。
综上所述,本研究采用基因芯片及生物信息学的方法对LOC102606465相关DEGs进行分析,发现ACTRT3、CDKN1A、DPYD、PRKACB、TPM4枢纽基因与LOC102606465潜在相互作用关系,对明确LOC102606465在细胞电离辐射响应过程中的分子作用机制具有重要意义。本研究仅通过生物信息学分析LOC102606465相关DEGs的功能作用,后续将采用体内、体外实验进一步验证LOC102606465在电离辐射响应过程中的作用及其分子机制。
利益冲突 无作者贡献声明 余昶负责实验操作、分析数据及论文撰写;王琪、王治东、周美娟在实验方案设计、结果分析及论文修改过程中予以指导;刘瑞雪、黄金凤负责采集数据、参与实验研究
| [1] |
Santivasi WL, Xia F. Ionizing radiation-induced DNA damage, response, and repair[J]. Antioxid Redox Signal, 2014, 21(2): 251-259. DOI:10.1089/ars.2013.5668 |
| [2] |
Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641. DOI:10.1016/j.cell.2009.02.006 |
| [3] |
Engreitz JM, Haines JE, Perez EM, et al. Local regulation of gene expression by lncRNA promoters, transcription and splicing[J]. Nature, 2016, 539(7629): 452-455. DOI:10.1038/nature20149 |
| [4] |
Ding LH, Shingyoji M, Chen F, et al. Gene expression profiles of normal human fibroblasts after exposure to ionizing radiation:a comparative study of low and high doses[J]. Radiat Res, 2005, 164(1): 17-26. DOI:10.1667/RR3354 |
| [5] |
Smyth GK, Gentleman R, Carey V, et al. Limma:linear models for microarray data, bioinformatics and computational biology solutions using R and bioconductor[M]. New York: Springer, 2005: 397-420. DOI:10.1007/0-387-29362-0_23
|
| [6] |
Chen H, Boutros PC. VennDiagram:a package for the generation of highly-customizable Venn and Euler diagrams in R[J]. BMC Bioinformatics, 2011, 12: 35. DOI:10.1186/1471-2105-12-35 |
| [7] |
Yu G, Wang LG, Han Y, et al. ClusterProfiler:an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
| [8] |
Wickham H. Ggplot2:elegant graphics for data analysis[M]. New York: Springer, 2016. DOI:10.1007/978-0-387-98141-3
|
| [9] |
Bader GD, Hogue CW. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4(1): 2. DOI:10.1186/1471-2105-4-2 |
| [10] |
Chin CH, Chen SH, Wu HH, et al. CytoHubba:identifying hub objects and sub-networks from complex interactome[J]. BMC Syst Biol, 2014, 8(Suppl 4): S11. DOI:10.1186/1752-0509-8-S4-S11 |
| [11] |
Kai M. Roles of RNA-binding proteins in DNA damage response[J]. Int J Mol Sci, 2016, 17(3): 310. DOI:10.3390/ijms17030310 |
| [12] |
Sage E, Shikazono N. Radiation-induced clustered DNA lesions:repair and mutagenesis[J]. Free Radic Biol Med, 2017, 107: 125-135. DOI:10.1016/j.freeradbiomed.2016.12.008 |
| [13] |
Slupphaug G, Kavli B, Krokan HE. The interacting pathways for prevention and repair of oxidative DNA damage[J]. Mutat Res, 2003, 531(1-2): 231-251. DOI:10.1016/j.mrfmmm.2003.06.002 |
| [14] |
Otani K, Naito Y, Sakaguchi Y, et al. Cell-cycle-controlled radiation therapy was effective for treating a murine malignant melanoma cell line in vitro and in vivo[J]. Sci Rep, 2016, 6: 30689. DOI:10.1038/srep30689 |
| [15] |
Geeves MA, Hitchcock-DeGregori SE, Gunning PW. A systematic nomenclature for mammalian tropomyosin isoforms[J]. J Muscle Res Cell Motil, 2015, 36(2): 147-153. DOI:10.1007/s10974-014-9389-6 |
| [16] |
Pendharkar N, Gajbhiye A, Taunk K, et al. Quantitative tissue proteomic investigation of invasive ductal carcinoma of breast with luminal B HER2 positive and HER2 enriched subtypes towards potential diagnostic and therapeutic biomarkers[J]. J Proteomics, 2016, 132: 112-130. DOI:10.1016/j.jprot.2015.11.024 |
| [17] |
Yang R, Zheng G, Ren D, et al. The clinical significance and biological function of tropomyosin 4 in colon cancer[J]. Biomed Pharmacother, 2018, 101: 1-7. DOI:10.1016/j.biopha.2018.01.166 |
| [18] |
Barr AR, Cooper S, Heldt FS, et al. DNA damage during S-phase mediates the proliferation-quiescence decision in the subsequent G1 via p21 expression[J]. Nat Commun, 2017, 8: 14728. DOI:10.1038/ncomms14728 |
| [19] |
Liu J, Ben Q, Lu E, et al. Long noncoding RNA PANDAR blocks CDKN1A gene transcription by competitive interaction with p53 protein in gastric cancer[J]. Cell Death Dis, 2018, 9(2): 168. DOI:10.1038/s41419-017-0246-6 |
| [20] |
Braun RE. Packaging paternal chromosomes with protamine[J]. Nat Genet, 2001, 28(1): 10-12. DOI:10.1056/NEJMct0803525 |
| [21] |
Lin K, Jiang H, Zhuang SS, et al. Long noncoding RNA LINC00261 induces chemosensitization to 5-fluorouracil by mediating methylation-dependent repression of DPYD in human esophageal cancer[J]. FASEB J, 2019, 33(2): 1972-1988. DOI:10.1096/fj.201800759R |
| [22] |
Massimi M, Cardarelli S, Galli F, et al. Increase of intracellular cyclic AMP by PDE4 inhibitors affects HepG2 cell cycle progression and survival[J]. J Cell Biochem, 2017, 118(6): 1401-1411. DOI:10.1002/jcb.25798 |
 2019, Vol. 39
2019, Vol. 39


