核与辐射事故发生后,对伤员进行快速准确的生物剂量评估是后续正确处理的关键。广泛用于辐射生物剂量估算的染色体畸变分析因实验周期长、分析技术要求高等缺点,难以满足较大人群受照后快速检测的要求。电离辐射诱导基因表达变化作为潜在生物剂量计,因其快速、高通量的特点成为目前研究的热点[1-3]。研究表明,与单一基因相比,应用一组基因通过统计学方法建立的用于估算生物剂量的评价模型,在剂量估算的准确度方面有了较大提高[4-5]。但如何优化模型,应用较少数量的基因建立估算效果最好的基因组合表达模型仍然是一项重要课题。
本实验在前期研究的基础上,通过检测人外周血在受到电离辐射作用后一组辐射敏感基因的相对表达量,利用逐步回归方法建立由几个基因组成的基因组合表达模型,并对其估算剂量的准确性进行初步验证,探讨其作为辐射生物剂量计的可行性。
材料与方法1.实验材料和仪器:RNAprep Pure血液总RNA提取试剂盒购于天根生化科技(北京)有限公司,高容量cDNA反转录试剂盒购于美国Thermo Fisher公司,Power SYBR Green PCR Master Mix购于美国ABI公司,Taq聚合酶,DEPC水购于北京鼎国昌盛生物技术有限公司,7500 Fast实时荧光定量PCR仪购自美国ABI公司,NanoDrop 2000紫外分光光度仪购自美国Thermo Fisher公司。
2.研究对象:在签署知情同意书的前提下,选择非放射工作者、无急慢性疾病、半年内无射线和化学毒物接触史的8名健康志愿者,男女各4人,年龄30~39岁,平均年龄为(33.13±2.30)岁。每名志愿者采集静脉血25 ml,平均分装至8个4 ml肝素锂抗凝真空采血管内备用。另外收集3名健康人外周血8 ml,平均分装至2个4 ml肝素锂抗凝真空采血管内,用于基因组合表达模型的离体验证。
3.照射条件和细胞培养:将已分装的血样于北京市辐照中心在室温下进行照射。吸收剂量为1 Gy/min,源靶距为73.5 cm,平均照射野为30 cm× 30 cm。剂量点分别0、0.5、1、2、3、4、6、8 Gy。照后将每个剂量点的血样平均接种于两个含有8 ml 10%胎牛血清的RPMI 1640培养瓶中(每瓶约含1.5 ml全血),置于37℃恒温培养箱中培养6和12 h。在离体验证实验中,双盲法照射人外周血,照射条件同上,剂量点为1和5 Gy,照后置于37℃恒温培养箱中培养6和12 h。
4.细胞总RNA提取与cDNA合成:照后预定时间点收集细胞,按照RNAprep Pure血液总RNA提取试剂盒产品说明书提取总RNA,然后通过NanoDrop 2000紫外分光光度仪测定纯度(A260/A280为1.8~2.0)并定量。然后应用高容量反转录试剂盒合成cDNA, 取100~200 ng RNA,加入20 μl的反应体系中,经25℃ 10 min,37℃ 120 min,85℃ 5 min后终止反应,-20℃冰箱保存。
5.实时定量PCR引物合成:根据GeneBank数据库的基因信息,应用Primer Premier 5.0软件设计用于实时定量PCR的辐射敏感基因以及内参基因β-肌动蛋白和B2M的引物序列。引物由上海生工生物技术有限公司合成,序列见表 1。
|
|
表 1 引物序列 Table 1 Primer sequences |
6.实时定量PCR检测:反应体系为20 μl:10 μl 2×SYBR green I,2 μl cDNA模板,上下游引物共0.5 μl和7.5 μl双蒸水。反应条件:95℃预变性10 min后进行下述40个循环:95℃变性15 s,60℃退火和延伸共1 min。每个样品设置3个平行样,在每次实时定量PCR反应后进行熔解曲线分析,以排除引物二聚体的影响,同时设立无引物空白对照组,重复3次实验。
7. PCR检测数据分析:利用Applied Biosystems 7500 Sequence Detection Software(SDS)进行结果分析。用2-ΔΔCt法,以β-肌动蛋白和B2M为内参基因,相对定量2个时间点和8个剂量点各基因的表达变化水平。其中,内参基因Ct值=
8.统计学处理:采用SPSS 25.0软件进行数据分析,基因相对表达量以x±s表示。组间比较经正态性检验符合正态分布,采用双侧t检验。剂量-效应关系分析采用一元线性回归分析。基因组合表达模型的构建采用多元线性逐步回归方法分析。P<0.05为差异有统计学意义。
结果1. 60Co γ射线照射后各基因表达的剂量-效应关系和时间-效应关系:结果见图 1。在0~8 Gy 60Co γ射线照射后,相同时间点、不同受照剂量之间,10个辐射敏感基因的相对表达量均随着受照剂量的增加而明显增加,并且照后两个时间点的各基因表达的变化趋势相一致,均呈现良好的剂量-效应关系(R2=0.61~0.97,P<0.05);相同受照剂量,不同照射时间点之间,PHPT1和PCNA两个基因在照后6 h的相对表达量高于照后12 h(图 1 B,E);TNFSF4、GADD45A、CCNG1、MDM2和TNFRSF10B 5个基因的相对表达量均随着照后时间的增加而明显升高(图 1 A,C,F,G,J);FDXR、XPC和GDF15 3个基因在照后6和12 h的基因表达量没有明显差异(图 1D,H,I)。
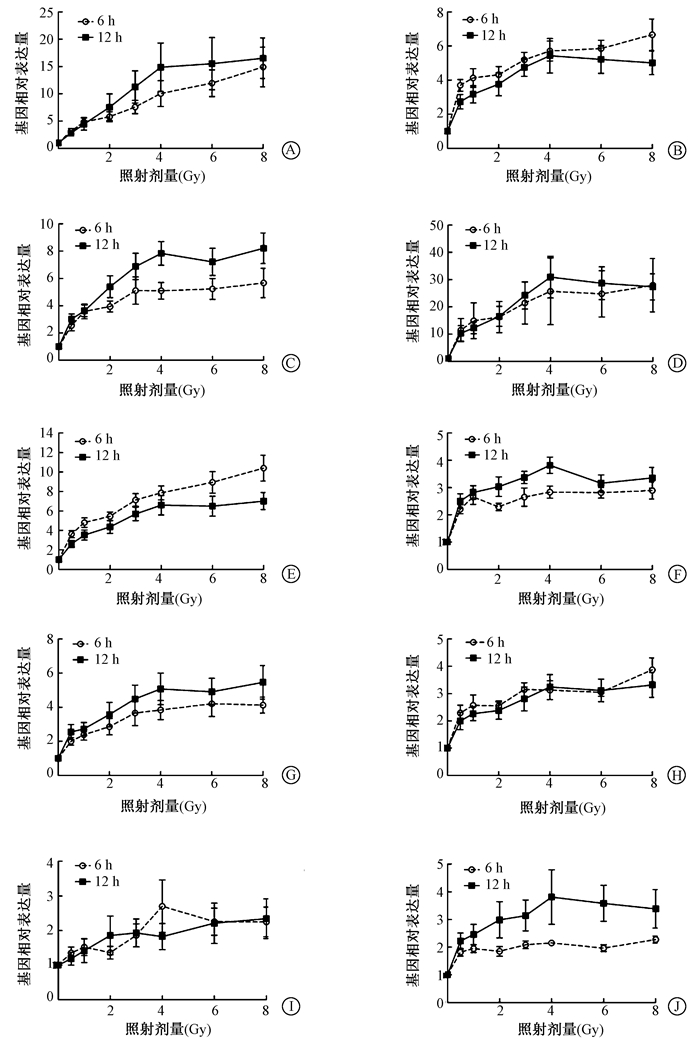
|
图 1 不同剂量60Co γ射线照射后不同时间各基因的相对表达水平 A. TNFSF4;B. PHPT1;C. GADD45A;D. FDXR;E. PCNA;F. CCNG1;G. MDM2;H. XPC;I. GDF15;J. TNFRSF10B Figure 1 Gene expression levels in ex vivo irradiated blood cells detected at different times after irradiation with different doses A. TNFSF4; B. PHPT1; C. GADD45A; D. FDXR; E. PCNA; F. CCNG1; G. MDM2; H. XPC; I. GDF15; J. TNFRSF10B |
2.基因表达的个体差异性:结果见图 2。在0~8 Gy 60Co γ射线照射8名志愿者外周血12 h后,对各基因的相对表达量进行分析。结果显示,在相同剂量点,GADD45A、PCNA和CCNG1 3个基因相对表达量的个体间差异均小于3倍(图 2 C,E,F);而TNFSF4、PHPT1和FDXR 3个基因表达水平在相同剂量点,不同个体间的差异可高达5倍以上(图 2 A,B,D)。
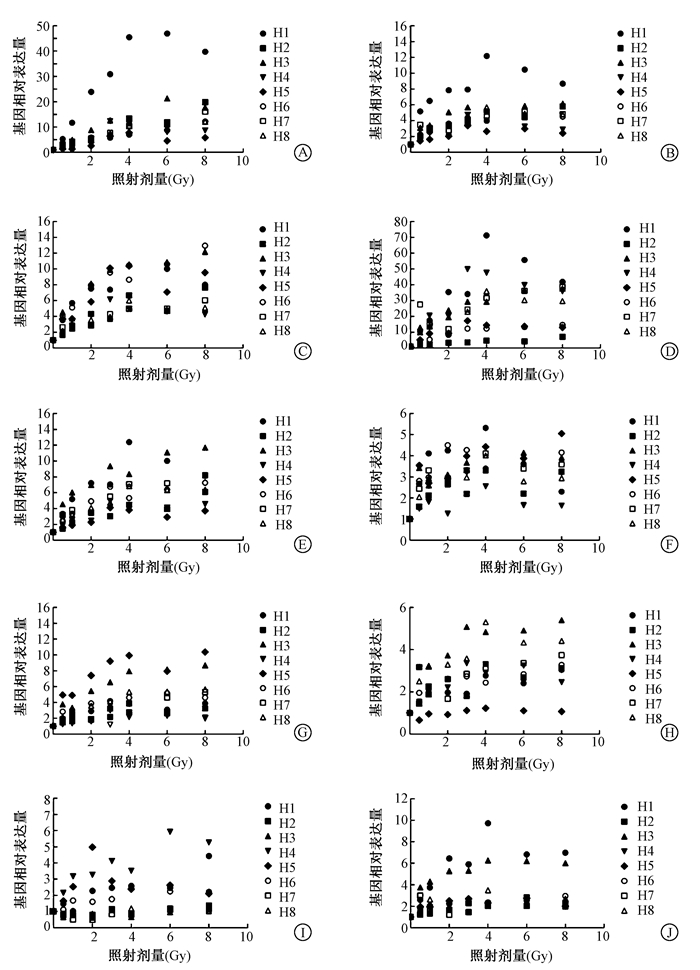
|
图 2 不同剂量60Co γ射线照射后12 h各基因相对表达量的个体差异性 A. TNFSF4;B. PHPT1;C. GADD45A;D. FDXR;E. PCNA;F. CCNG1;G. MDM2;H. XPC;I. GDF15;J. TNFRSF10B Figure 2 Individual variations of gene expression levels in ex vivo irradiated blood cells at 12 h after irradiation wiht different doses. A. TNFSF4; B. PHPT1; C. GADD45A; D. FDXR; E. PCNA; F. CCNG1; G. MDM2; H. XPC; I. GDF15; J. TNFRSF10B |
3.基因组合表达模型的建立:由图 1可知,各基因在照后6和12 h的基因相对表达量与受照剂量均呈现线性相关性,采用多元线性逐步回归方法,得到生物剂量回归方程(表 2)6 h:y=0.213TNFSF4-0.604PHPT1+0.542GADD45A-0.06FDXR+0.91PCNA-1.407CCNG1(R2=0.88,F=54.8,P<0.001);12 h:y=0.575MDM2+0.916PCNA+0.504GD15-1.539TNFRSF10B+0.167TNFSF4-0.667CCNG1(R2=0.82,F=42.767,P<0.001),其中y为照射剂量,Gy; 基因名称为照后不同时间点该基因的相对表达量。
|
|
表 2 基因组合表达模型逐步回归分析 Table 2 Stepwise regression analysis of multiple gene expression-based models |
4.基因组合表达模型的初步验证:1和5 Gy 60Co γ射线照射3名健康人外周血,照后6和12 h检测纳入模型的基因相对表达量,应用相应的基因组合表达模型估算受照剂量。照后6 h,2个剂量点的平均估算剂量分别为(1.05±0.43)和(5.10±0.15)Gy; 照射12 h,2个剂量点的平均估算剂量分别为(1.07±0.21)和(4.87±0.28)Gy,各剂量点的相对偏差(|估算剂量-真实照射剂量|)≤0.5 Gy。
讨论在大范围核与辐射恐怖袭击事件中,快速、有效、准确的生物剂量估算对伤员的分类诊断与医学救治至关重要。近年来应用由不同数量基因组合建立的生物剂量评价模型已经成为研究的热点,它的优点是联合多个基因的表达化对受照剂量进行估算,可以较大程度地提高剂量估算的准确度,提示可能是辐射生物剂量计的重要方向[7-9]。本研究在前期实验的基础上,相对定量检测10个辐射敏感基因的表达水平,同时分析每个基因在不同个体间的表达差异,通过多元逐步回归的方法建立用于生物剂量估算的基因组合表达模型。
TNFSF4、PHPT1、GADD45A、FDXR、PCNA、CCNG1、MDM2、XPC、GDF15和TNFRSF10B是本实验室前期对近20年已发表的国外相关研究,应用文献计量学和生物信息学等方法筛选并验证后确定的辐射敏感基因[10]。有研究表明,不同剂量的电离辐射也会引起一些内参基因的表达发生改变,为保证实验结果的准确性与稳定性,本研究应用β-肌动蛋白和B2M共同作为内参基因[6, 11]。通过检测8名正常人外周血在受到0~8 Gy 60Co γ射线照射后上述基因的表达变化,发现10个基因的表达水平均呈现随着照射剂量增加而增加,呈现较好的剂量-效应关系;同时TNFSF4、GADD45A、CCNG1、MDM2和TNFRSF10B 5个基因的相对表达量随着受照时间的增加而升高,这与之前已报道的研究结果基本相一致[12]。在个体间差异方面,TNFSF4、PHPT1和FDXR 3个基因对电离辐射具有较强的辐射敏感性,但同时存在明显的个体差异性,如4 Gy 60Co γ射线照射12 h后,TNFSF4基因的相对表达量在不同个体间的差异可高达9倍,这说明基因表达量的改变不只是由电离辐射所诱导的,同时可能也会受到吸烟、性别、慢性照射和炎症反应等潜在混杂因素的影响[3, 12]。因此,建立多基因组合表达模型可以较大程度地减少相关干扰因素对生物剂量估算的准确度和模型适用范围的影响。
目前,用于基因组合表达模型的建立方法主要包括两大类:一类是基于芯片技术,通常由几十或几百个差异表达基因组成,其优点是简便、快速、高通量,但其成本高,只能对受照剂量进行分类诊断[4, 13-15];另一类是基于统计学方法,将几个或十几个差异表达基因进行组合,建立生物剂量预测模型,其优点是统计方法简单、易掌握[11, 16-17]。本研究主要采用第二类方法,即多元逐步回归方法分别建立了由6个基因组成的受照后6和12 h的回归方程,其R2值均分别为0.88和0.82,说明大部分的基因表达变化是由电离辐射所诱导的。在纳入模型的基因中,PCNA、CCNG1和TNFSF4 3个基因共同存在于两个回归方程中,其中PCNA和CCNG1是个体间差异较小的两个基因,对方程的稳定性具有重要作用。尽管已有研究表明在纳入3~4个基因的条件下,利用逐步回归方法建立的剂量回归模型,在0.15~6 Gy照射剂量范围内对用于建立模型的样本进行剂量估算的准确率达80%以上,但该研究的不足之处是缺乏离体与整体实验的验证[15]。因此,本研究应用离体实验分别对所建立的基因组合表达模型进行了初步了验证,结果发现,各受照样本的估算剂量均比较接近于真实照射剂量,提示本研究建立的基因组合表达模型作为辐射生物剂量计具有一定的可行性。
综上所述,本研究建立并初步验证了60Co γ射线照射正常人外周血不同时间点的基因组合表达模型,研究结果可用于人群受照后的生物剂量估算,因此具有一定的实际应用价值。下一步研究方向是在现有基因组合表达模型的基础上,一方面应用体内和体外实验验证其估算辐射生物剂量的准确性;另一方面,扩大样本量,研究相关干扰因素对基因组合表达模型的影响。
利益冲突 无作者贡献声明 李爽负责实验设计、实验操作、数据整理及论文撰写;封江彬和陆雪负责样本采集、参与实验研究;田梅和刘青杰在实验方案设计,结果分析和论文修改过程中给予指导
| [1] |
Kis E, Szatmári T, Keszei M, et al. Microarray analysis of radiation response genes in primary human fibroblasts[J]. Int J Radiat Oncol Biol Phys, 2006, 66(5): 1506-1514. DOI:10.1016/j.ijrobp.2006.08.004 |
| [2] |
Wang HP, Long XH, Sun ZZ, et al. Identification of differentially transcribed genes in human lymphoblastoid cells irradiated with 0.5 Gy of gamma-ray and the involvement of low dose radiation inducible CHD6 gene in cell proliferation and radiosensitivity[J]. Int J Radiat Biol, 2006, 82(3): 181-190. DOI:10.1080/09553000600632261 |
| [3] |
Manning G, Macaeva E, Majewski M, et al. Comparable dose estimates of blinded whole blood samples are obtained independently of culture conditions and analytical approaches Second RENEB gene expression study[J]. Int J Radiat Biol, 2017, 93(1): 87-98. DOI:10.1080/09553002.2016.1227105 |
| [4] |
Paul S, Amundson SA. Gene expression signatures of radiation exposure in peripheral white blood cells of smokers and non-smokers[J]. Int J Radiat Biol, 2011, 87(8): 791-801. DOI:10.3109/09553002.2011.568574 |
| [5] |
Paul S, Smilenov LB, Amundson SA. Widespread decreased expression of immune function genes in human peripheral blood following radiation exposure[J]. Radiat Res, 2013, 180(6): 575-583. DOI:10.1667/RR13343.1 |
| [6] |
Brzóska K, Kruszewski M. Toward the development of transcriptional biodosimetry for the identification of irradiated individuals and assessment of absorbed radiation dose[J]. Radiat Environ Biophys, 2015, 54(3): 353-363. DOI:10.1007/s00411-015-0603-8 |
| [7] |
Broustas CG, Xu Y, Harken AD, et al. Comparison of gene expression response to neutron and x-ray irradiation using mouse blood[J]. BMC Genomics, 2017, 18(1): 2. DOI:10.1186/s12864-016-3436-1 |
| [8] |
Broustas CG, Xu Y, Harken AD, et al. Impact of neutron exposure on global gene expression in a human peripheral blood mode[J]. Radiat Res, 2017, 187(4): 433-440. DOI:10.1667/RR0005.1 |
| [9] |
Sproull M, Camphausen K. State-of-the-art advances in radiation biodosimetry for mass casualty events involving radiation exposure[J]. Radiat Res, 2016, 186(5): 423-435. DOI:10.1667/RR14452.1 |
| [10] |
Li S, Lu X, Feng JB, et al. Identification and validation of candidate radiation-responsive genes for human biodosimetry[J]. Biomed Environ Sci, 2017, 30(11): 834-840. DOI:10.3967/bes2017.112 |
| [11] |
Tucker JD, Joiner MC, Thomas RA, et al. Accurate gene expression-based biodosimetry using a minimal set of human gene transcripts[J]. Int J Radiat Oncol Biol Phys, 2014, 88(4): 933-939. DOI:10.1016/j.ijrobp.2013.11.248 |
| [12] |
Manning G, Kabacik S, Finnon P, et al. High and low dose responses of transcriptional biomarkers in ex vivo X-irradiated human blood[J]. Int J Radiat Biol, 2013, 89(7): 512-522. DOI:10.3109/09553002.2013.769694 |
| [13] |
Boldt S, Knops K, Kriehuber R, et al. A frequency-based gene selection method to identify robust biomarkers for radiation dose prediction[J]. Int J Radiat Biol, 2012, 88(3): 267-276. DOI:10.3109/09553002.2012.638358 |
| [14] |
Knops K, Boldt S, Wolkenhauer O, et al. Gene expression in low-and high-dose-irradiated human peripheral blood lymphocytes:possible applications for biodosimetry[J]. Radiat Res, 2012, 178(4): 304-312. DOI:10.1667/RR2913.1 |
| [15] |
Riecke A, Rufa CG, Cordes M, et al. Gene expression comparisons performed for biodosimetry purposes on in vitro peripheral blood cellular subsets and irradiated individuals[J]. Radiat Res, 2012, 178(3): 234-243. DOI:10.1667/RR2738.1 |
| [16] |
Pogosova-Agadjanyan EL, Fan W, Georges GE, et al. Identification of radiation-induced expression changes in nonimmortalized human T cells[J]. Radiat Res, 2011, 175(2): 172-184. DOI:10.1667/RR1977.1 |
| [17] |
Joiner MC, Thomas RA, Grever WE, et al. Developing point of care and high-throughput biological assays for determining absorbed radiation dose[J]. Radiother Oncol, 2011, 101(1): 233-236. DOI:10.1016/j.radonc.2011.05.068 |
 2018, Vol. 38
2018, Vol. 38


