放射性肠损伤是腹腔局部放疗、盆腔放疗和造血干细胞移植预处理中常见的不良反应之一[1]。放射性肠损伤不仅严重影响患者的生活质量,而且在某些情况下可能危及生命,目前仍缺乏有效的防治手段控制放射性肠损伤[2]。在以往的研究中,大多关注腹部照射后肠道形态学的变化,但对于其病理的分子生物学变化的发生发展规律还尚未深入[3]。尽管受照后引起p53信号通路激活早已成为共识,但具体是哪些p53信号通路中的信号仍是未知。
本研究为了探究受照后小鼠肠道的分子生物学变化规律,采用对小鼠腹部照射致死剂量的急性放射性肠损伤模型,提取照射后不同时间小鼠肠道组织中RNA,通过转录组测序,高通量获得不同时间后表达差异的基因,进而揭示基因表达与放射损伤之间的信号通路关系,对急性放射性肠损伤中的发展规律研究提供参考。
材料与方法1.照射条件:由中国医学科学院放射医学研究所提供的137Cs γ射线照射源进行照射,在室温下进行全腹照射(剑突以上、耻骨联合以下使用铅板),剂量率为1.01 Gy/min。
2.动物分组及处理:小鼠购自北京维通利华实验动物技术有限公司,6~8周龄,雄性C57BL6/J小鼠,合格证号:11400700255030。动物研究方案经中国医学科学院放射医学研究所伦理委员会审查和批准。在30 d存活率实验中分为2组:腹部照射12 Gy组和14 Gy组,每组40只。转录组测序实验中分为4组,分别为对照组、照射后6 h组、照射后3.5 d组和照射后5 d组,每组3只,照射剂量为14 Gy。对照组小鼠在相同条件下,接受腹部的假照射(0 Gy)。
3.小鼠肠道样品采集和RNA的提取:照射后不同时间脱颈处死小鼠,剪开腹膜,充分暴露腹部,取空肠组织于-80℃冻存,用于后续RNA的提取。取小鼠肠道样品长度约5 mm组织于1.5 ml离心管内,加入200 μl TRIzol后用研磨机打碎,后添加TRIzol至1 ml,采用TRIzol法提取总RNA,检验吸光度A260/A280的比值, 范围在1.8~2.1则RNA纯度合格。提取的RNA于-80℃保存备用,用于测序送样待检。
4.转录组测序:RNA文库制备及测序由上海云序生物科技有限公司完成。肠道样本中的RNA使用Ribo-Zero rRNA Removal Kits移除总RNA中的rRNAs。采用TruSeq Stranded Total RNA Library Prep Kit预处理RNA,构建测序文库。通过美国安捷伦公司BioAnalyzer 2100生物分析仪进行文库质量控制和定量。在美国Illumina公司HiSeq测序仪上采用双端模式进行150 cycle测序。基因表达差异在2倍以上,且P < 0.05则认为是显著差异基因。
5.信号通路分析:在主成分分析(principal components analysis,PCA)中,使用全部基因和外显子的表达值进行标准化后,校正后的数据以散点图表示。标准化后的各组基因信号值进行校正后,再用基因芯片显著性分析(significance analysis of microarrays, SAM)软件进行分析。变化倍数在2倍以上(即上调2倍以上或下调至对照50%以下)认为是显著差异表达基因。将差异基因输入到HemI软件中进行热图绘制,并通过Funrich软件进行基因间相互作用分析。随后使用IPA (ingenuity pathway analysis)软件对差异基因所涉及的信号通路按重要性排序,进一步筛选P < 0.05的细胞信号通路进行后续分析。
6.统计学处理:采用SPSS 19.0软件进行分析。组间比较经正态性检验符合正态分布,采用双侧t检验。P < 0.05为差异有统计学意义。生存分析使用Kaplan-Meier法计算生存组和死亡组30 d生存率。
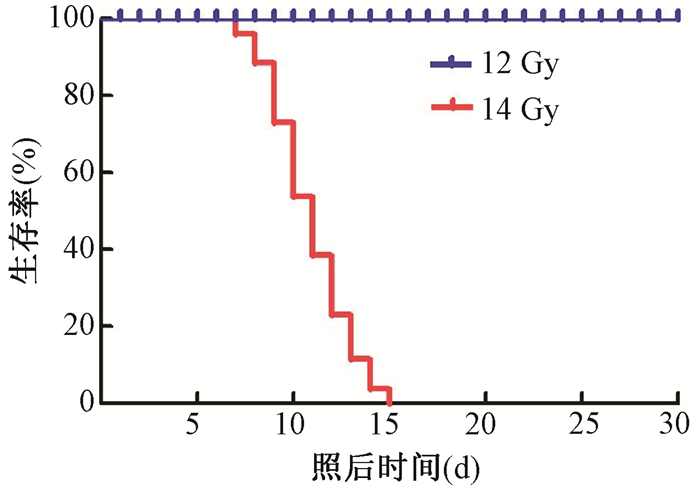
结果1.腹部照射后小鼠30 d存活率:结果如图 1所示。腹部照射12 Gy组小鼠全部存活。14 Gy组中,小鼠在照射后第11天开始死亡,照射后第15天全部死亡。
|
图 1 小鼠经腹部照射12和14 Gy后30 d存活曲线 Figure 1 Survival curve of C57BL/6 mice treated with PBS or MSCs after 12 or 14 Gy ABI |
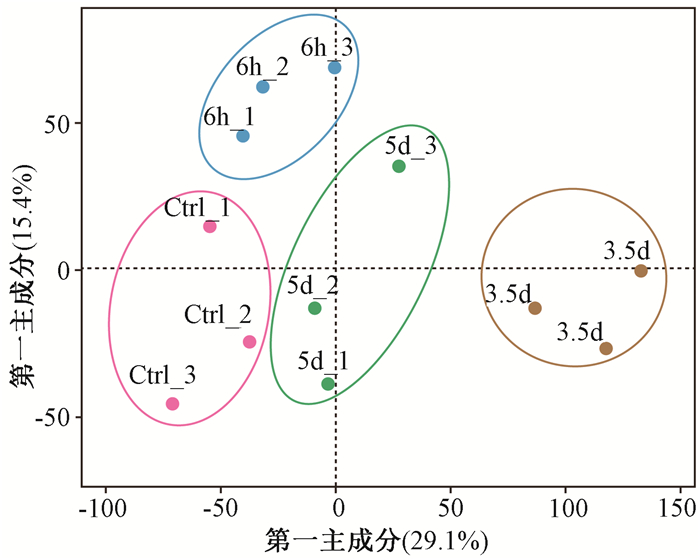
2.腹部照射14 Gy后不同时间小鼠空肠样本基因表达主成分分析:结果表明,照射后6 h至第5天,不同组之间呈现出了显著的分离。如图 2所示,腹部照射后3.5 d与对照组距离最远,表明照后3.5 d与对照组中的基因表达差异最为显著。在转录层面上,照射后6 h,3.5与5 d后分别与对照组相比,上调和下调的差异基因数目如表 1所示。
|
图 2 照射后不同时间小鼠空肠样本的差异基因主成分分析 Figure 2 2D principal components analysis (PCA) of jejunum samples collected at the indicated time points after ABI |
|
|
表 1 腹部照射后差异基因数目分析 Table 1 The number of differentially expressed genes after ABI |
3.腹部照射后3.5 d空肠样本中上调及下调基因分析:由于照射后3.5 d与对照组差异基因数目最多,因此本研究根据差异基因中上调和下调的基因做IPA分析,观察差异基因所涉及的细胞信号通路。
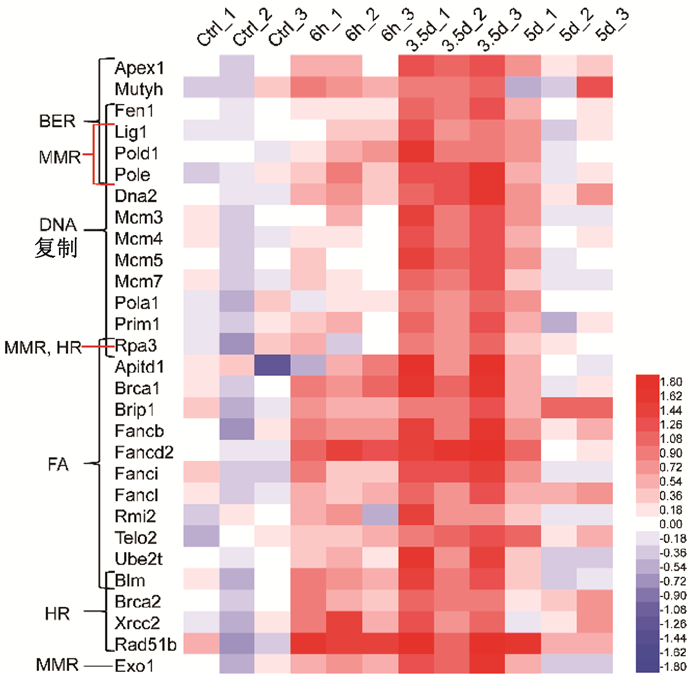
照射后上调的差异基因经过IPA分析后表明,电离辐射造成的DNA损伤后,主要激活了以下DNA损伤修复信号通路包括碱基切除修复,错配修复和同源重组等。但某些基因包如Fen1等基因涉及多条DNA损伤修复信号通路,如图 3所示。
|
图 3 小鼠在腹部照射后第3.5天后空肠样本中DNA损伤修复相关的基因上调 注:BER.碱基切除修复;MMR.错配修复;HR.同源重组;FA.范可尼贫血症途径 Figure 3 The gene expression profiles showed the upregulation of DNA damage repair pathways in mouse jejunum after 3.5 d of ABI |
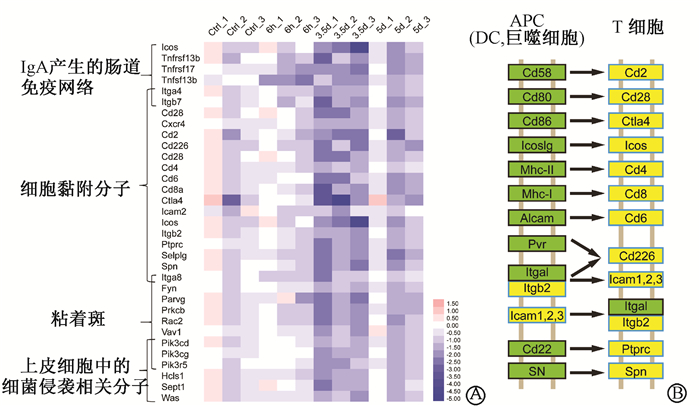
在照射后第3.5天下调的基因中发现了更多的关于细胞黏附分子,粘着斑的基因的下调,以及抵抗细菌侵袭的上皮细胞下调和肠道黏膜免疫中IgA分泌途径信号的下调(图 4)。下调的细胞黏附分子,主要集中在T细胞受体信号通路中(图 4),如涉及抗原呈递细胞或DC细胞与T细胞之间的黏附分子Cd2、Cd4、Cd6、Cd8、Cd28、Cd266、Ctla、Icos、Itgb2等在照射后3.5 d显著下调。这一结果与结果4中下调的Lck和Fyn基因涉及了T细胞受体信号通路所一致。
|
图 4 腹部照射后3.5 d小鼠空肠样本中下调基因分析 A.肠道样本中IgA产生的肠道免疫网络,细胞黏附分子、黏着斑和上皮细胞中的细菌侵袭相关分子热图;B.KEGG分析下调的细胞黏附分子所涉及抗原呈递细胞与T细胞之间的相互作用 注:黄色为下调的基因,绿色为未呈显著性差异的基因 Figure 4 Analysis of down-regulated genes in mouse jejunum at 3.5 days after ABI A. The gene expression profiles showed upregulation of DNA damage repair pathways and down-regulation of cell adhesion molecules, focal adhesion, bacterial invasion of epithelial cells and IgA production pathways; B. KEGG map visualization of differentially expressed genes involved in immune system signaling pathway |
4.腹部照射3.5 d后差异基因相互作用分析:腹部照射后3.5 d与对照组差异基因数目最多,本实验室利用Funrich软件对差异基因进行Pathway分析和相互作用分析,发现差异基因中Lck、Cdk1和Fyn处于关键位置(图 5)。Lck、Cdk1和Fyn涉及的信号通路如表 2所示,其中Lck和Fyn为腹部照射后3.5 d空肠样本中下调的基因,并且均为酪氨酸激酶Src家族成员,Pathway分析表明Lck和Fyn涉及的信号通路多为免疫相关的信号通路,如T细胞受体,自然杀伤性细胞介导的细胞毒等免疫相关的信号通路受到了抑制。Cdk1为腹部照射后3.5 d空肠样本中上调的基因,Pathway分析表明照射后3.5 d空肠样本中,细胞周期和p53信号通路被激活。

|
图 5 小鼠腹部照射14 Gy后3.5 d空肠差异基因的相互作用网络图 Figure 5 Gene interaction network analysis of differentially expressed genes in mouse jejunum samples at 3.5 d after 14 Gy ABI |
|
|
表 2 Lck、Cdk1、Fyn 3个基因涉及的信号通路 Table 2 The signal pathways involved in Lck, Cdk1 and Fyn genes |
5.腹部照射后6 h与3.5 d共同激活p53信号通路:将照射后6 h,3.5和5 d中表达差异的基因输入到IPA软件中进行细胞信号通路分析。其中照射后6 h与3.5 d的两组样本中p53信号通路都受到激活,涉及的p53信号通路的基因包括Bax、Bbc3、Ccng1、Ppm1d、Sesn2、Zmat3、Ccnb1、Gtse1、Rrm2、Ccnb2、Ccnd1、Ccne1、Cdk1、Cdk4、Chek1和Chek2(图 6)。照射后6 h与照射后3.5 d的差异基因相比,差异的基因有所不同,并少于照射后3.5 d,其中Ccnb1、Gtse1、Rrm2为共同差异基因。表明照射后3.5 d,更多的p53信号通路中的基因被激活。尽管p53信号通路在照射后6 h和3.5 d都呈现出高表达,但pathway分析,p53信号通路并未在照射后5 d的小鼠肠道组织中被激活。

|
图 6 照射后6 h和3.5 d小鼠空肠样本中共同激活p53信号通路的热图分析 Figure 6 Heatmap of the transcript genes in p53 pathway in mouse jejunum at 6 h and 3.5 d after ABI |
讨论
随着测序技术的高速发展,二代测序技术高通量、高速度和低成本的优点,使得它越来越被科学家们所青睐。在二代测序中,转录组测序技术主要是针对转录产物mRNA进行高通量测序,获得特定组织在某一状态下的几乎所有转录本序列信息,是基因功能研究的基础。本课题为了高通量地了解放射性肠损伤的分子病理机制,采用转录组测序获得了小鼠腹部照射14 Gy后不同时间的空肠转录组信息,采用PCA分析、IPA分析和KEGG分析等方法,对不同组样本中的差异基因进行分析。
本研究采用对C57BL6/J小鼠单次腹部照射14 Gy作为急性放射性肠损伤研究方案,这与Guo等[4]和Gong等[5]的研究方案所一致。急性放射性肠损伤小鼠的肠道损伤的形态学变化结果已在本实验室前期工作中予以体现[5-6]。本研究着重在于对该模型进行基因测序分析。在单次腹部照射后(14 Gy) 6 h与3.5 d,两组样品中小鼠肠道组织中p53信号通路中涉及的多个基因上调表达。电离辐射后激活的p53信号通路,可能潜在地为肠道组织提供保护作用,这是因为在Kirsch等[7]的研究中发现,肠道上皮细胞特异性的剔除p53基因小鼠模型更容易因放射引起肠道损伤,而高表达的p53转基因小鼠的放射性肠损伤程度明显降低。Leibowitz等[8]的研究表明,照射后p53信号通路激活可能为放射性肠损伤提供了某种保护机制。但是,p53信号通路中,具体是哪些基因在照射后的肠道中发挥作用,还是需要进一步的探究。在本课题中,通过转录组测序分析,p53信号通路中的不同基因在照射后不同时相的变化,为p53信号通路的研究提供更多的候选基因,进一步提示p53信号通路在放射性肠损伤中的作用。
转录组测序结果表明,在小鼠腹部照射3.5 d后其肠道组织与未照射组差异基因数目最多。针对于腹部照射3.5 d后,通过Funrich基因相互作用分析表明,Lck、Cdk1与Fyn处在中心位置,并涉及多个信号通路,这一结果有助于针对放射性肠损伤靶点和针对特异性药物开发的研究。
此外,还对上调和下调差异基因进行IPA分析。结果表明,在上调的基因中,主要激活了DNA损伤修复信号通路。DNA损伤修复通路与放射性肠损伤中细胞类型的修复一直是科学家研究的重点,尤其是ATM、AIM2等明星分子[9-10]。在已有文献报道中,在腹部照射后引起的肠道组织中的DNA损伤,并激活DNA损伤修复途径[11]。本研究通过转录组测序的方法,发现了更多的DNA损伤修复中涉及的分子转录水平的上调,这一结果与Zheng等[12]的研究结果一致,在腹部照射后3.5 d后多种DNA损伤修复相关的信号通路被激活。但不足的是,DNA损伤修复中的分子往往涉及多种修复途径,仍需要更多的工作去了解不同分子在放射性肠损伤中的作用。在下调的基因中,针对于肠道免疫的IgA信号通路,细胞间黏附分子、黏着斑及抵抗细菌对肠道上皮细胞的侵袭通路受到了抑制。既往研究中,无论是腹部照射还是全身照射,都会引起模式动物如小鼠、恒河猴等模式动物肠道屏障的破坏[13-14]。本研究发现细胞间黏附分子和黏着斑通路中相关基因的下调,有可能与照射后肠道屏障的破坏有关。在T细胞受体信号通路中,针对靶点TLR5和TLR9的激动剂已经在小鼠和恒河猴的模型中证明具有减轻放射性肠损伤的作用[15-17],可见T细胞受体信号通路的重要作用。在下调细胞间黏附分子基因中,更多的集中在抗原呈递细胞与T细胞之间的相互作用,这与照射后3.5 d主要涉及的细胞信号通路T细胞受体信号通路密切相关。
综上所述,本研究通过对小鼠腹部照射后不同时间肠道的转录组测序结果分析,探讨了不同时相中涉及的共同信号通路p53信号通路和关键基因Lck、Cdk1与Fyn的信号交联,并针对照后3.5 d后差异基因最为显著的这组样本中,针对DNA损伤修复途径与放射性肠损伤中黏膜屏障破坏,细菌侵入和IgA的产生涉及的信号通路做了进一步的阐释。这些结果有望为急性放射性肠损伤的发展规律和关键靶点的筛选提供更多的研究方向。
利益冲突 本研究还接受中国医学科学院医学与健康科技创新工程项目(2017-I2M-1-016)、协和青年基金和中央高校基本科研业务费专项资金(3332016100)资助。由署名作者按以下贡献声明独立开展,未接受有关公司的任何赞助,不涉及各相关方的利益冲突作者贡献声明 王津晗负责分析数据、论文撰写;路倩颖负责对结果进行统计和分析;纪凯华和杜利清负责构思、指导论文的撰写;王彦和徐畅负责论文的修改;刘强指导实验设计、论文的撰写和修改
| [1] |
Hauer-Jensen M, Denham JW, Andreyev HJ. Radiation enteropathy-pathogenesis, treatment and prevention[J]. Nat Rev Gastroenterol Hepatol, 2014, 11(8): 470-479. DOI:10.1038/nrgastro.2014.46 |
| [2] |
Stacey R, Green JT. Radiation-induced small bowel disease:latest developments and clinical guidance[J]. Ther Adv Chronic Dis, 2014, 5(1): 15-29. DOI:10.1177/2040622313510730 |
| [3] |
王津晗, 徐畅, 王彦, 等. 放射性肠损伤模型及其评价研究进展[J]. 国际放射医学核医学杂志, 2017, 41(1): 33-39, 44. Wang JH, Xu C, Wang Y, et al. Progress on the evaluating and establishing methods of radiation-induced intestinal injuries animal model[J]. Int J Radiat Med Nucl Med, 2017, 41(1): 33-39, 44. DOI:10.3760/cma.j.issn.1673-4114.2017.01.007 |
| [4] |
Guo MZ, Gong W, Zhang HW, et al. Human mesenchymal stem cells promote survival and prevent intestinal damage in a mouse model of radiation injury[J]. RSC Adv, 2016, 6(69): 65105-65111. DOI:10.1039/c6ra05165k |
| [5] |
Gong W, Guo MZ, Han Z, et al. Mesenchymal stem cells stimulate intestinal stem cells to repair radiation-induced intestinal injury[J]. Cell Death Dis, 2016, 7(9): e2387. DOI:10.1038/cddis.2016.276 |
| [6] |
Wang JH, Yao M, Wang Y, et al. 6-Shogaol ameliorates injury to the intestinal mucosa and increases survival after high-dose abdominal irradiation[J]. J Funct Foods, 2017, 36: 63-71. DOI:10.1016/j.jff.2017.06.054 |
| [7] |
Kirsch DG, Santiago PM, di TE, et al. p53 controls radiation-induced gastrointestinal syndrome in mice independent of apoptosis[J]. Science, 2010, 327(5965): 593-596. DOI:10.1126/science.1166202 |
| [8] |
Leibowitz BJ, Qiu W, Liu H, et al. Uncoupling p53 functions in radiation-induced intestinal damage via PUMA and p21[J]. Mol Cancer Res, 2011, 9(5): 616-625. DOI:10.1158/1541-7786.MCR-11-0052 |
| [9] |
Chandrakesan P, May R, Weygant N, et al. Intestinal tuft cells regulate the ATM mediated DNA damage response via Dclk1 dependent mechanism for crypt restitution following radiation injury[J]. Sci Rep, 2016, 6: 37667. DOI:10.1038/srep37667 |
| [10] |
Hu B, Jin C, Li HB, et al. The DNA-sensing AIM2 inflammasome controls radiation-induced cell death and tissue injury[J]. Science, 2016, 354(6313): 765-768. DOI:10.1126/science.aaf7532 |
| [11] |
Poindexter SV, Reddy VK, Mittal MK, et al. Transcriptional corepressor MTG16 regulates small intestinal crypt proliferation and crypt regeneration after radiation-induced injury[J]. Am J Physiol Gastrointest Liver Physiol, 2015, 308(6): G562-571. DOI:10.1152/ajpgi.00253.2014 |
| [12] |
Zheng J, Garg S, Wang J, et al. Laser capture microdissected mucosa versus whole tissue specimens for assessment of radiation-induced dynamic molecular and pathway changes in the small intestine[J]. PLoS One, 2013, 8(1): e53711. DOI:10.1371/journal.pone.0053711 |
| [13] |
Garg S, Zheng J, Wang J, et al. Segmental differences in radiation-induced alterations of tight junction-related proteins in non-human primate jejunum, ileum and colon[J]. Radiat Res, 2016, 185(1): 50-59. DOI:10.1667/RR14157.1 |
| [14] |
Biju PG, Garg S, Wang W, et al. Procalcitonin as a predictive biomarker for total body irradiation-induced bacterial load and lethality in mice[J]. Shock, 2012, 38(2): 170-176. DOI:10.1097/SHK.0b013e31825b2db3 |
| [15] |
申星, 熊国林, 谢玲, 等. Toll样受体5激动剂CBLB502对7.0 Gy 60Co γ射线照射恒河猴的辐射防护作用观察[J]. 解放军医学杂志, 2015, 40(4): 285-291. Shen X, Xiong GL, Xie L, et al. Radioprotective effects of CBLB502 on γ-radiated Rhesus monkey[J]. Med J Chin PLA, 2015, 40(4): 285-291. DOI:10.11855/j.issn.0577-7402.2015.04.07 |
| [16] |
Saha S, Bhanja P, Liu L, et al. TLR9 agonist protects mice from radiation-induced gastrointestinal syndrome[J]. PLoS One, 2012, 7(1): e29357. DOI:10.1371/journal.pone.0029357 |
| [17] |
Burdelya LG, Krivokrysenko VI, Tallant TC, et al. An agonist of toll-like receptor 5 has radioprotective activity in mouse and primate models[J]. Science, 2008, 320(5873): 226-230. DOI:10.1126/science.1154986 |
 2018, Vol. 38
2018, Vol. 38


