2. 100850 北京, 军事医学科学院放射与辐射医学研究所 放射生物学北京市重点实验室
2. Institute of Radiation Medicine, Academy of Military Medical Sciences, Beijing Key Laboratory for Radiobiology, Beijing 100850, China
DNA依赖蛋白激酶的催化亚单位(DNA dependent protein kinase catalytic subunit,DNA-PKcs)是由富含亮氨酸的重复序列(leucine rich region,LRR)、磷酸化簇区域(phosphorylation cluster)、caspase 3剪切位点(caspase 3 cleavage sites)、Ku结合区域(Ku interaction domain)、FAT结构域(FAT domain)、FATC结构域(FATC domain)及羧基端激酶活性等区域组成[1],其中磷酸化簇区域是位于N端的一段具有多段磷酸化集簇的区域。早期对于DNA-PKcs蛋白的研究多集中于C端区域,对于N端的磷酸化簇区域的认识并不多,但近年来发现该区域的自磷酸化和协同磷酸化与肿瘤放射治疗敏感性密切相关[2]。在前期研究中,发现针对该区域的单链抗体anti-DPK3-scFv可以增强肿瘤细胞对放射治疗的敏感性[3]。为研究该区域参与调控细胞放射敏感性的机制,本课题组构建了针对该区域的诱饵质粒pGBKT7-DPC[4]。本研究拟通过酵母双杂交的方法筛选与DNA-PKcs磷酸化簇区域相互作用的蛋白,对筛选出的阳性酵母细胞克隆进行鉴定,最后将阳性的克隆蛋白连入真核表达载体中,通过免疫共沉淀实验检测阳性克隆蛋白与DNA-PKcs磷酸化簇区域之间的相互作用。
材料与方法1.载体、菌株和细胞系:质粒pGBKT7、pGADT7、pGBKT7-p53和pGADT7-T(美国Clontech公司),真核表达质粒PCD3-CMYC和PCD3-HA(美国Life Technologies公司),诱饵质粒pGBKT7-DPC为前次实验构建[4];酵母菌株AH109(美国Clontech公司),XL1-Blue菌由本室保存;人胚肾293T细胞购自美国ATCC公司;人肝组织酵母文库由天津赛尔生物技术有限公司提供。
2.主要试剂:限制性内切酶、LA Taq DNA聚合酶、T4 DNA连接酶(日本TaKaRa公司);RIPA细胞裂解液、蛋白上样缓冲液、兔抗CMYC和HA标签抗体、HRP标记羊抗兔IgG二抗(上海碧云天公司);酵母培养基(上海睿星基因有限公司);PCR产物纯化试剂盒、DNA凝胶回收试剂盒、质粒提取试剂盒和DNA marker(北京天根生物有限公司);脂质体Lipofectamine 2000、Opti-MEM、DMEM、磷酸盐缓冲液(PBS)、胰酶和胎牛血清(美国Life Technologies公司);蛋白酶抑制剂、硝酸纤维素膜和增强化学发光试剂盒(德国Merck Millipore公司);其他试剂均为国产分析纯。
3.细胞培养:用含10%胎牛血清的DMEM高糖培养基,在恒温37℃、5%CO2的条件下培养293T细胞。用0.25%的胰酶消化,2~3 d传代1次。
4.酵母双杂交实验:以转化入AH109酵母细胞的pGBKT7-DPC质粒为“诱饵”,按照酵母双杂交手册操作,采用酵母融合方法筛选人肝组织酵母文库。转化后的菌液先铺至SD/-Leu/-Trp/-His平板,待阳性克隆长出后,将阳性克隆复制到SD/-Leu/-Trp/-His/-Ade和SD/-Leu/-Trp平板上继续筛选,将在两块平板上均有生长的克隆再接种至另一块SD/-Leu/-Trp/-His/-Ade平板上进行第2轮筛选。
5.阳性酵母细胞克隆PCR鉴定:扩大培养含有阳性克隆的酵母细胞后,提取其中的质粒,然后转化至XL1-Blue感受态菌,挑取单克隆后分离纯化。通过采用特异性引物(pGADT7-Insert)的PCR反应鉴定质粒中所含外源性片段的大小并分析比对,引物见表 1。
|
|
表 1 引物数据汇总 Table 1 Summary of primer |
6.阳性酵母细胞克隆回转验证:将PCR鉴定阳性的质粒分别与诱饵质粒pGBKT7-DPC共转至AH109酵母细胞内,并铺于SD/-Leu/-Trp平板上,待克隆长出后,再将克隆分别接种至SD/-Leu/-Trp和SD/-Leu/-Trp/-His/-Ade平板上,以验证文库质粒编码蛋白和诱饵蛋白之间是否具有相互作用。设计阳性对照组(共转pGBKT7-p53和pGADT7-T)、阴性对照组(共转pGBKT7和pGADT7)、其他对照组(共转pGBKT7和文库质粒)以及实验组(共转pGBKT7-DPC和文库质粒)。
7.阳性克隆测序及结果分析:对回转实验验证也为阳性的质粒进行测序分析(由北京诺赛基因组研究中心有限公司完成),然后将测序结果提交在线BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)程序检索分析。
8. PCD3-CMYC-DPC、PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1真核表达载体构建及鉴定:根据之前筛选anti-DPK3-scFv所用的抗原序列设计扩增DNA-PKcs磷酸化簇区域的引物(表 1),以载体pGBKT7-DPC为模板。PCR扩增条件:预变性94℃ 3 min;94℃ 45 s,52℃ 45 s,72℃ 1 min,共30个循环;延伸72℃ 10 min。
根据GeneBank中的序列设计扩增MBNL1、SIK2和YY1AP1的引物(表 1),以cDNA为模板进行扩增。MBNL1和YY1AP1的反应条件为94℃预变性3 min;94℃ 45 s,60℃ 45 s,72℃ 2 min,共30个循环;72℃延伸10 min。SIK2的反应条件为94℃预变性3 min;94℃ 45 s,58℃ 45 s,72℃ 3 min,共30个循环;72℃延伸10 min。
PCR扩增产物两端带有酶切位点(DNA-PKcs磷酸化簇区域为KpnI和XhoI, MBLN1和SIK2均为BamHI和XhoI, YY1AP1为EcoRI和XhoI),经回收后进行双酶切,然后与同样经过双酶切的载体进行定向连接,转化至感受态菌中。经酶切鉴定,选择一个正确的阳性克隆进行测序,将测序结果正确的重组质粒命名为PCD3-CMYC-DPC、PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1。
9.蛋白质印迹:分别将PCD3-CMYC-DPC、PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1转染至293T细胞中,转染步骤参照Lipofectamine2000操作说明。转染后72 h收集蛋白,经10%十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)电泳、转膜、封闭、孵育一抗(PCD3-CMYC-DPC用兔抗CMYC标签抗体检测,PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1用兔抗HA标签抗体进行检测)和二抗,最后经发光显示在X光片上。
10.免疫共沉淀实验(Co-Immunoprecipitation,Co-IP):分别将质粒PCD3-CMYC-DPC与PCD3-HA-MBNL1、PCD3-HA-SIK2或PCD3-HA-YY1AP1共转染至293T细胞中,72 h后冰上裂解细胞(裂解液:50 mmol/L Tris HCl pH=8.0,120 mmol/L NaCl,1% Nonidet P-40,2 mmol/L EDTA,蛋白酶抑制剂)30 min,经4℃、12 000 r/min,离心半径7 cm,离心30 min后取上清。取10%的上清作为对照Input,剩余上清与经过免疫预清洗的CMYC单抗交联protein A琼脂糖珠4℃孵育4 h。接着4℃、3 000 r/min,离心半径7 cm,离心3 min后弃上清,用裂解洗琼脂糖珠3次;最后加入蛋白上样缓冲液,沸水煮5 min。用Western blot技术检测免疫沉淀复合物中的蛋白。
结果1.人肝组织酵母文库筛选及阳性酵母细胞克隆PCR鉴定:将表达pGBKT7-DPC的AH109酵母细胞与人肝细胞酵母文库融合杂交,经两轮筛选,大部分克隆仍可在四缺平板(SD/-Leu/-Trp/-His/-Ade)上生长。扩增含有阳性克隆的酵母细胞后提取其中的文库质粒转化至大肠杆菌XL1-Blue,挑取单克隆分离纯化得到12个文库质粒克隆。通过PCR鉴定文库质粒中插入基因片段的大小,经排除大小相同的插入片段后,共确定7个插入片段长度互不相同的文库克隆(C1、C2、C3、C5、C6、C9和C10,图 1)。
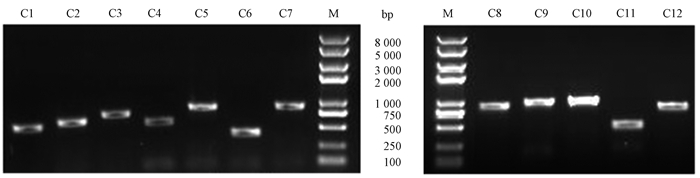
|
图 1 DNA-PKcs磷酸化簇区域筛选的阳性酵母细胞克隆PCR鉴定结果 注:M. DNA标志物; C1~12. 1~12号阳性克隆 Figure 1 PCR identification of the positive clones screened by DNA-PKcs phosphorylation cluster |
2.阳性酵母细胞克隆回转验证:将上一步筛选得到的7个文库质粒分别与pGBKT7-DPC诱饵载体共转至酵母细胞内,并铺于SD/-Leu/-Trp平板上,待克隆长出后,将克隆分别接种至SD/-Leu/-Trp和SD/-Leu/-Trp/-His/-Ade平板上,以验证文库质粒编码蛋白和诱饵蛋白之间是否具有相互作用。图 2显示所有共转染组均可以在Trp和Leu缺陷平板(SD/-Leu/-Trp)上生长,但分别共转pGBKT7和7个文库质粒的其他对照组在同时缺陷His和Ade的平板(SD/-Leu/-Trp/-His/-Ade)上不能生长,说明各文库质粒编码的蛋白均缺乏自激活作用;共转pGBKT7-DPC和pGADT7-C1、C2、C6和C10的实验组同共转pGBKT7和pGADT7的阴性对照组一样,在同时缺陷His和Ade的平板(SD/-Leu/-Trp/-His/-Ade)上不能生长,而共转pGBKT7-DPC和pGADT7-C3、C5和C9同共转pGBKT7-p53和pGADT7-T的阳性对照组一样,在SD/-Leu/-Trp/-His/-Ade平板上也能生长,提示C1、C2、C6和C10号克隆编码蛋白与DNA-PKcs磷酸化簇区域之间不存在相互作用,而C3、C5和C9号克隆存在。

|
图 2 阳性酵母细胞克隆回转验证结果 注:pGBKT7-p53+pGADT7-T(阳性对照组); pGBKT7+ pGADT7(阴性对照组); pGBKT7+文库质粒(其他对照组); pGBKT7-DPC+文库质粒(实验组) Figure 2 Identification of the positive clones by rotary validation |
3.阳性克隆测序结果:对C3、C5和C9号克隆进行测序,并采用BLAST程序进行分析,结果显示,C3为MBNL1 (Homo sapiens muscleblind like splicing regulator 1),C5为SIK2 (Homo sapiens salt inducible kinase 2),C9为YY1AP1 (Homo sapiens YY1 associated protein 1)。
4. PCD3-CMYC-DPC、PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1真核表达载体构建及鉴定:构建PCD3-CMYC-DPC、PCD3-HA-MBNL1、PCD3-HA-SIK2和PCD3-HA-YY1AP1真核表达载体,经酶切和测序鉴定均正确后将它们与相对应的空载体分别转染至293T细胞中,72 h后各组分别进行Western blot检测。如图 3所示,转染PCD3-CMYC-DPC组在约相对分子质量34 000位置、PCD3-HA-MBNL1组在约45 000位置、PCD3-HA-SIK2组在约100 000位置、PCD3-HA-YY1AP1组在约50 000位置均可检测到相应融合蛋白的表达。
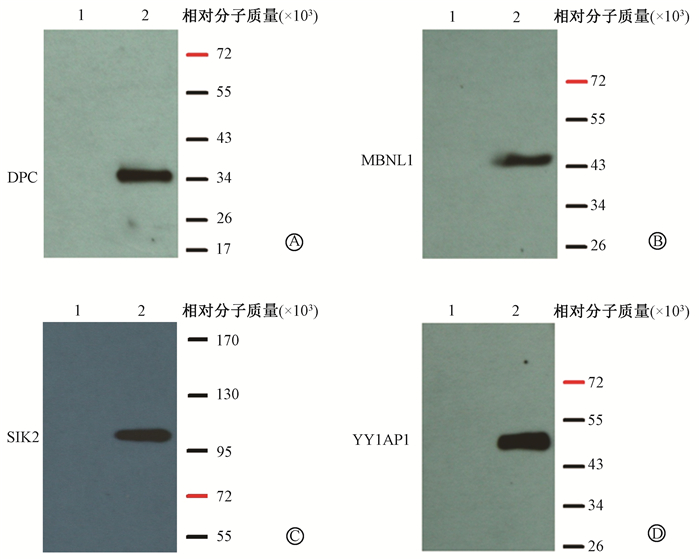
|
图 3 Western blot检测真核表达载体在293T细胞中的表达情况 注:A1. PCD3-CMYC; A2. PCD3-CMYC-DPC; B1. PCD3-HA; B2. PCD3-HA-MBNL1; C1. PCD3-HA; C2. PCD3-HA-SIK2; D1. PCD3-HA; D2. PCD3-HA-YY1AP1 Figure 3 Western blot assay of the eukaryotic expression vector in 293T cells |
5.真核细胞内候选基因与DNA-PKcs磷酸化簇相互作用的检测:将PCD3-CMYC-DPC分别与PCD3-HA-MBNL1、PCD3-HA-SIK2或PCD3-HA-YY1AP1共转至293T细胞中,通过免疫共沉淀实验检测DPC与MBNL1、SIK2或YY1AP1之间的相互作用。如图 4所示,DPC与MBNL1、YY1AP1或SIK2分别共转染293T细胞后,细胞裂解物中可检测到两种蛋白的表达(Input组),与CMYC单抗琼脂糖珠孵育后,在免疫沉淀复合物中也可检测到两种蛋白的表达(IP:CMYC组),表明在真核细胞内DNA-PKcs磷酸化簇与MBNL1、SIK2和YY1AP1之间均存在相互作用。

|
图 4 免疫共沉淀实验检测DPC与MBNL1、SIK2或YY1AP1在293T细胞中的相互作用A和B.共转PCD3-CMYC-DPC和PCD3-HA-MBNL1;C和D.共转PCD3-CMYC-DPC和PCD3-HA-SIK2;E和F.共转PCD3-CMYC-DPC和PCD3-HA-YY1AP1 注:1. Input; 2. IP: CMYC Figure 4 Interaction of DNA-PKcs phosphorylation cluster (DPC) between MBNL1, SIK2 or YY1AP1 detected by Co-IP analysis in 293T cells A and B. Cotransfection of PCD3-CMYC-DPC and PCD3-HA-MBNL1; C and D. Cotransfection of PCD3-CMYC-DPC and PCD3-HA-SIK2; E and F. Cotransfection of PCD3-CMYC-DPC and PCD3-HA-YY1AP1 |
讨论
放射疗法是肿瘤治疗最常用的方法之一,但是肿瘤细胞增强的DNA损伤修复能力常导致放射治疗的失败[5]。DNA-PKcs是DNA损伤修复最重要分子之一,属于磷脂酰肌醇3激酶样蛋白激酶(PIKK)激酶家族[6],它与由Ku70和Ku80组成的靶向DNA的异二聚体共同组成完整的DNA依赖的蛋白激酶(DNA-PK)复合物[7-8]。当发生DNA双链断裂(DSBs)时,DNA-PKcs会被激活并启动非同源性末端连接(non-homologous end joining,NHEJ)通路修复DNA损伤[8-9]。既往对DNA-PKcs的研究多集中在C端区域,但近年来发现,N端区域特别是磷酸化簇区域在DNA损伤修复过程中也发挥着重要作用,该区域的缺失或者磷酸化位点的突变会显著影响细胞的损伤修复能力以及对辐射的敏感性[10-12]。在前期研究中,将针对DNA-PKcs磷酸化簇区域的单链抗体导入HeLa细胞后也发现类似的现象[3],但是磷酸化簇区域影响细胞放射敏感性的机制目前并不清楚,因此有必要筛选该区域的相互作用分子。
前期研究成功构建了DNA-PKcs磷酸化簇区域的诱饵质粒pGBKT7-DPC,并且通过观察其对酵母细胞生长的影响和自激活活性分析证实其可用于相互作用蛋白的筛选[4]。本研究将表达pGBKT7-DPC的酵母细胞与人肝细胞酵母文库融合杂交,经过两轮筛选后提取阳性酵母细胞克隆的文库质粒,通过PCR的方法排除片段大小一致的文库克隆以及回转验证排除假阳性结果后得到3株阳性克隆,经测序鉴定分别是MBNL1、SIK2和YY1AP1。由于酵母细胞是较为简单的真核细胞,其蛋白质加工和修饰过程与高等的哺乳动物细胞存在一定的差别,进一步将筛选出来的克隆蛋白以及诱饵蛋白构建至哺乳动物细胞真核表达载体中,通过免疫共沉淀的方法对MBNL1、SIK2和YY1AP1与DNA-PKcs磷酸化簇区域的相互作用进行了验证确定它们在人胚肾293T细胞中也存在相互作用。酵母双杂交实验检测出MBNL1、SIK2和YY1AP1与DNA-PKcs磷酸化簇区域可以直接相互作用,而Co-IP实验则验证了外源性过表达的蛋白在哺乳动物细胞内也可以发生相互作用,但是内源性的相互作用是否发生,还需要结合具体的细胞类型以及不同的生理或者病理状态等进行验证和分析。
肌盲蛋白样剪接调节蛋白1(muscleblind like splicing regulator 1,MBNL1) 是一个高度保守的RNA结合蛋白(RNA binding protein,RBP),通过直接与靶RNA结合参与调控RNA可变剪接、选择性多聚腺苷酸化(alternative polyadenylation,APA)、RNA稳定性和定位等[13-14]。盐诱导激酶2(salt inducible kinase 2,SIK2) 是一种丝氨酸/苏氨酸激酶,属于钙调素依赖性蛋白激酶(calcium calmodulin kinases,CaM)和AMP激活的蛋白激酶(AMP-activated protein kinases,AMPK)家族,可作为细胞能量变化的感受器,参与调控细胞能量代谢[15]。也有研究显示,SIK2是有丝分裂的关键调节分子之一,调控有丝分裂前中期中心体的分离[16]。目前对于YY1相关蛋白1(YY1 associated protein 1,YY1AP1) 的研究相对较少,有研究报道YY1AP1是YY1的转录共激活子[17],在肝癌细胞中可以协同YY1调控染色质图谱(chromatin landscape)并激活“干性”调节蛋白的表达[18]。
本研究首次通过目前使用较成熟的酵母双杂交实验对与DNA-PKcs磷酸化簇区域相互作用的蛋白进行筛选,并通过Co-IP实验在哺乳动物细胞内进行了验证。后续课题组还对SIK2与DNA-PKcs之间的相互作用进行了进一步验证[19],目前并没有其他文献报道MBNL1、SIK2和YY1AP1与DNA-PKcs磷酸化簇区域存在相互作用,根据本研究结果推测,在某些情况下,MBNL1、SIK2和YY1AP1可能协同DNA-PKcs参与调控细胞放射敏感性,另一方面DNA-PKcs也可能参与MBNL1、SIK2和YY1AP1相关的生物学功能,提示DNA-PKcs是一个多功能的蛋白。总之,本研究为深入研究DNA-PKcs参与调控细胞放射敏感性以及其他生物学功能的机制提供了新思路。
利益冲突 作者无利益冲突,排名无争议,作者的配偶、工作伙伴或子女不存在影响研究结果的财务关系,在此对研究的独立性和科学性予以保证作者贡献声明 吴成林执行项目的研究、撰写和修改论文;刘晓丹、王欲晓、杜丽和付凯飞执行部分研究;周丽君设计研究方案,分析讨论研究结果
| [1] | Lees-Miller SP, Meek K. Repair of DNA double strand breaks by non-homologous end joining[J]. Biochimie, 2003, 85 (11): 1161-1173. DOI:10.1016/j.biochi.2003.10.011. |
| [2] | Nagasawa H, Little JB, Lin YF, et al. Differential role of DNA-PKcs phosphorylations and kinase activity in radiosensitivity and chromosomal instability[J]. Radiat Res, 2011, 175 (1): 83-89. DOI:10.1667/RR2092.1. |
| [3] | Du L, Zhou LJ, Pan XJ, et al. Radiosensitization and growth inhibition of cancer cells mediated by an scFv antibody gene against DNA-PKcs in vitro and in vivo[J]. Radiat Oncol, 2010, 5 : 70 DOI:10.1186/1748-717X-5-70. |
| [4] |
吴成林, 刘晓丹, 王欲晓, 等. 用酵母双杂交系统鉴定DNA-PKcs磷酸化簇区域和DNA-PKcs单链抗体anti-DPK3-scFv间的相互作用[J].
生物技术通讯, 2016, 27 (3): 335-339. Wu CL, Liu XD, Wang YX, et al. Identification of interaction between DNA dependent protein kinase catalytic subunit(DNA-PKcs)phosphorylation cluster and DNA-PKcs scFv antibody anti-DPK3-scFv by yeast two-hybrid assay[J]. Lett Biotechnol, 2016, 27 (3): 335-339. DOI:10.3969/j.issn.1009-0002.2016.03.008. |
| [5] | Morgan MA, Lawrence TS. Molecular pathways:overcoming radiation resistance by targeting DNA damage response pathways[J]. Clin Cancer Res, 2015, 21 (13): 2898-2904. DOI:10.1158/1078-0432.CCR-13-3229. |
| [6] | Collis SJ, DeWeese TL, Jeggo PA, et al. The life and death of DNA-PK[J]. Oncogene, 2005, 24 (6): 949-961. DOI:10.1038/sj.onc.1208332. |
| [7] | Helleday T, Petermann E, Lundin C, et al. DNA repair pathways as targets for cancer therapy[J]. Nat Rev Cancer, 2008, 8 (3): 193-204. DOI:10.1038/nrc2342. |
| [8] | Falck J, Coates J, Jackson SP. Conserved modes of recruitment of ATM, ATR and DNA-PKcs to sites of DNA damage[J]. Nature, 2005, 434 (7033): 605-611. DOI:10.1038/nature03442. |
| [9] | Gao Y, Chaudhuri J, Zhu C, et al. A targeted DNA-PKcs-null mutation reveals DNA-PK-independent functions for KU in V(D)J recombination[J]. Immunity, 1998, 9 (3): 367-376. DOI:10.1016/S1074-7613(00)80619-6. |
| [10] | Jiang W, Crowe JL, Liu X, et al. Differential phosphorylation of DNA-PKcs regulates the interplay between end-processing and end-ligation during nonhomologous end-joining[J]. Mol Cell, 2015, 58 (1): 172-185. DOI:10.1016/j.molcel.2015.02.024. |
| [11] | Cartwright IM, Bell JJ, Maeda J, et al. Effects of targeted phosphorylation site mutations in the DNA-PKcs phosphorylation domain on low and high LET radiation sensitivity[J]. Oncol Lett, 2015, 9 (4): 1621-1627. DOI:10.3892/ol.2015.2974. |
| [12] | Zou LH, Shang ZF, Tan W, et al. TNKS1BP1 functions in DNA double-strand break repair though facilitating DNA-PKcs autophosphorylation dependent on PARP-1[J]. Oncotarget, 2015, 6 (9): 7011-7022. DOI:10.18632/oncotarget.3137. |
| [13] | Pascual M, Vicente M, Monferrer L, et al. The Muscleblind family of proteins:an emerging class of regulators of developmentally programmed alternative splicing[J]. Differentiation, 2006, 74 (2-3): 65-80. DOI:10.1111/j.1432-0436.2006.00060.x. |
| [14] | Wang ET, Cody NA, Jog S, et al. Transcriptome-wide regulation of pre-mRNA splicing and mRNA localization by muscleblind proteins[J]. Cell, 2012, 150 (4): 710-724. DOI:10.1016/j.cell.2012.06.041. |
| [15] | Park J, Yoon YS, Han HS, et al. SIK2 is critical in the regulation of lipid homeostasis and adipogenesis in vivo[J]. Diabetes, 2014, 63 (11): 3659-3673. DOI:10.2337/db13-1423. |
| [16] | Ahmed AA, Lu Z, Jennings NB, et al. SIK2 is a centrosome kinase required for bipolar mitotic spindle formation that provides a potential target for therapy in ovarian cancer[J]. Cancer Cell, 2010, 18 (2): 109-121. DOI:10.1016/j.ccr.2010.06.018. |
| [17] | Wang CY, Liang YJ, Lin YS, et al. YY1AP, a novel co-activator of YY1[J]. J Biol Chem, 2004, 279 (17): 17750-17755. DOI:10.1074/jbc.M310532200. |
| [18] | Zhao X, Parpart S, Takai A, et al. Integrative genomics identifies YY1AP1 as an oncogenic driver in EpCAM(+) AFP(+) hepatocellular carcinoma[J]. Oncogene, 2015, 34 (39): 5095-5104. DOI:10.1038/onc.2014.438. |
| [19] |
王建校. 盐诱导激酶SIK2与DNA-PKcs相互作用的鉴定[D]. 石河子: 石河子大学, 2014.
Wang JX. Study of the interaction between SIK2 and DNA-PKcs[D]. Shihezi:Shihezi Univ, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10759-1015517602.htm |
 2017, Vol. 37
2017, Vol. 37



