2. 100850 北京 军事医学科学院放射与辐射医学研究所 放射生物学北京市重点实验室
2. Institute of Radiation Medicine, the Military Medical Sciences, Beijing Key Laboratory for Radiobiology, Beijing 100850, China
宫颈癌是女性第三大恶性肿瘤[1],其放射治疗中存在的放射不敏感,使宫颈癌治疗效果不令人满意,所以寻找宫颈癌放射敏感性靶标是近年来众多学者关注的热点问题。
IER5(immediate early response 5) 是早期反应基因之一,基因芯片结果发现放射损伤诱导其表达[2], 是放射损伤暴露剂量相关的DNA损伤修复蛋白生物标记物[3]。本研究组前期经实时定量PCR方法验证放射损伤后IER5基因在mRNA水平上表达发生变化[4]。有文献报道,IER5在宫颈癌组织中较正常宫颈组织表达增加了89.3倍[5]。IER5是否可作为宫颈癌治疗新靶标及其作用机制还有待进一步阐明。本研究拟在蛋白水平上验证IER5放射后变化,并筛选出IER5在放射损伤后的相互作用蛋白,为进一步研究放射损伤后的作用机制奠定基础。
材料与方法1.主要试剂与仪器:ANTI-FLAG M2 Affinity Gel、Flag抗体、0.25%胰蛋白酶 (美国Sigma-aldrich公司),Lipofectamine2000 (美国Invitrogen公司),M-PER蛋白裂解液 (美国Thermo公司),DMEM、胎牛血清 (美国Hyclone公司),IP裂解液NETN100 (本实验室配置:Tris-HCL pH=8.0, 20 mmol/L,EDTA 1 mmol/L,NaCl 100 mmol/L,NP40 0.5%),蛋白酶抑制剂混合片剂cocktail (瑞士Roche公司),IER5多克隆抗体 (美国Abcam公司),proteinA/G plus-agarose、PARP1抗体、Histone H1抗体 (美国Santa Cruz公司),GAPDH单克隆抗体、HRP标记的山羊抗鼠二抗、HRP标记的兔抗山羊二抗 (北京中杉金桥公司),细胞核蛋白与细胞质蛋白抽提试剂盒 (上海碧云天生物技术有限公司)。
2.细胞培养:HeLa细胞及人肾上皮细胞293T (军事医学科学院放射与辐射医学研究所实验室) 使用DMEM培养基培养,含10%胎牛血清,1%青霉素和链霉素,置于5%CO2,37 ℃恒温培养箱。使用0.25%胰蛋白酶消化分离,进行传代。
3.细胞转染:取对数期293T、HeLa细胞转染,密度70%~90%,转染前换为双无培养基,100 mm培养皿转染8 μg质粒3×Flag\|IER5,质粒与脂质体 (质量:体积=1:2) 分别与双无培养基作用5 min,混合后继续作用20 min,加入培养皿中,十字摇匀,6 h后换正常培养基。转染36~48 h后照射并收集细胞,两种细胞均分别设照射组,将处理后未照射细胞分别设对照组。验证实验使用HeLa细胞。
4.细胞照射:采用军事医学科学院辐射中心60Co γ辐射源进行单次照射,照射总剂量4 Gy,吸收剂量率为91.54 cGy/min,源靶距为3 m。
5.免疫共沉淀:293T细胞转染36 h后进行4 Gy照射,照后1 h收集样品。预冷PBS洗2遍,PBS吹落混匀细胞,4 ℃,3 000 r/min,离心半径7 cm, 离心3 min,弃上清,每组加入NETN100裂解液6 ml+100×cocktail 60 μl冰上裂解15 min,4 ℃ 12 000 r/min,离心半径7 cm, 离心10 min,取上清,各留100 μl做细胞总蛋白input;于proteinA/G plus-agarose预吸附混悬1.5 h,4 ℃ 2 000 r/min,离心半径7 cm, 离心1 min,取上清,于anti-flag-beads混悬4 h,4 ℃ 2 000 r/min,离心半径7 cm, 离心1 min,弃上清,NETN100混悬清洗3×8 min,最后加100 μl NETN100,与6×上样缓冲液混匀后沸水煮10 min,-80 ℃保存。
6.免疫印迹 (Western blot):HeLa细胞照后1、2、4、6、8、12、24、48 h收集细胞于离心管中,加入蛋白裂解液100 μl,冰上裂解10 min,4 ℃ 12 000 r/min,离心半径7 cm,离心10 min,取上清并测浓度;另取受照后0.5、2、8 h细胞,抽取细胞核内蛋白,方法参照试剂盒说明书。根据蛋白浓度电泳上样100 μg;质谱样品细胞总蛋白input及IP各取8 μl上样。10%SDS-PAGE胶恒压电泳,100 V电转膜90 min,5%脱脂奶粉室温封闭1 h,孵育,PARP1抗体、Flag抗体、IER5抗体、GAPDH抗体、Histone H1抗体4 ℃过夜,TBST洗一抗,10 min×3次,室温孵育相应二抗1 h,TBST洗10 min×3次,化学发光法显影。
7.数据处理:蛋白定量分析使用Image J软件进行分析,所有实验独立重复至少3次,质谱鉴定的蛋白质用在线分析软件DAVID进行功能注释和分析。
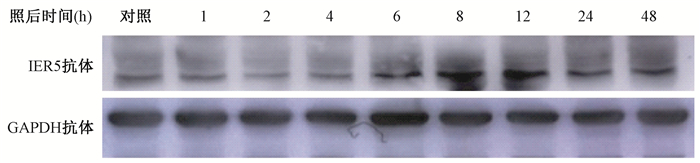
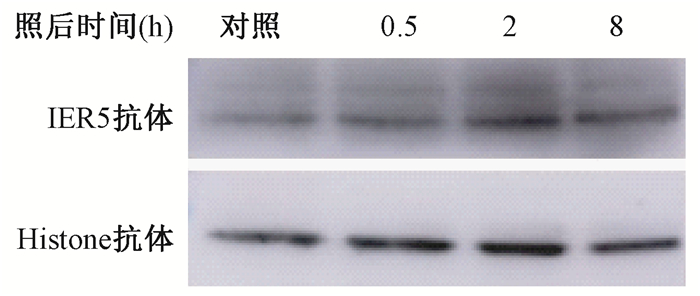
结 果1. HeLa细胞受照后IER5蛋白表达量的变化:结果显示,细胞受照后,IER5蛋白表达上调,于照后4 h蛋白水平开始增加,12 h蛋白水平至高峰,蛋白表达增至2倍,并持续至48 h,见图 1。受照后,核内IER5蛋白有增多趋势,照后0.5 h核内IER5蛋白即有升高,见图 2。
|
图 1 Western blot检测HeLa细胞4 Gy照射后不同时间点IER5蛋白的表达 Figure 1 Western blot analysis of IER5 protein expression in Hela cells after 4 Gy irradiation |
|
图 2 Western blot检测HeLa细胞4 Gy照射后不同时间点细胞核内IER5蛋白表达 Figure 2 Western blot analysis of IER5 protein in the nucleus of HeLa cells after 4 Gy irradiation |
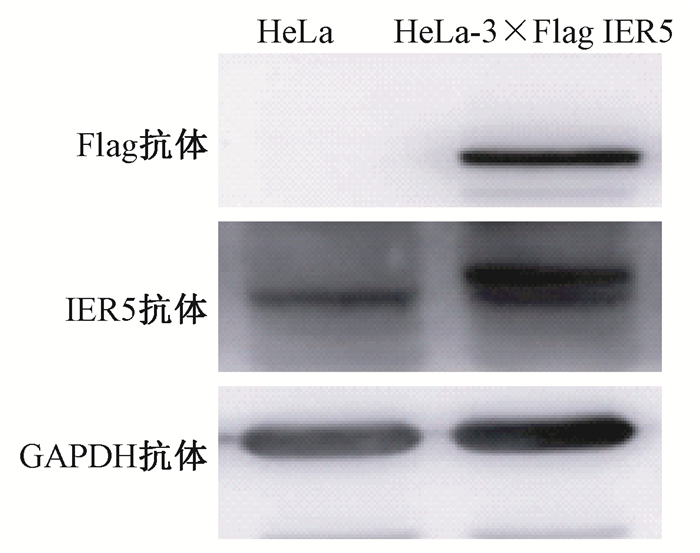
2. Western blot检测转染3×Flag-IER5融合蛋白的表达:使用p-CMV载体构建3×Flag-IER5融合表达质粒,瞬时转染至HeLa细胞,48 h后Western blot检测结果见图 3,3×Flag-IER5载体成功表达,标签抗体及目的抗体均能识别。
|
图 3 Western blot检测HeLa细胞3×Flag-IER5融合蛋白表达 Figure 3 Western blot analysis of 3×Flag-IER5 protein |
3.质谱结果及筛选:免疫共沉淀复合物SDS-PAGE分离并染色后,条带切胶送质谱分析。分析质谱得出数据,照射组检测出374个蛋白,对照组检测出256个蛋白,两组间比较,匹配肽链数≥2的差异蛋白有41个。使用DAVID在线工具进行蛋白功能分类,照射组中与DNA结合、代谢、损伤修复相关的蛋白包括10个,见表 1。
|
|
表 1 质谱分析联合DAVID在线分析软件筛选出与IER5相互作用蛋白 Table 1 MS and DAVID online datebase analysis of IER5 interacting proteins |
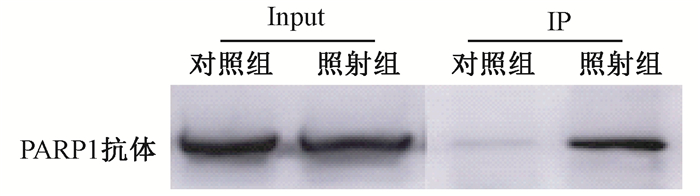
4.质谱结果验证:根据质谱分析的结果,选择PARP1蛋白进行质谱样品验证,图 4结果显示,PARP1与IER5在细胞发生放射损伤后存在明显的相互作用关系,对照组也同样检测到PARP1(考虑可能对照组PARP1蛋白量少而质谱未能检测出),照射后相互作用蛋白增多。同时, 本实验也采用HeLa细胞重复了上述实验,验证了此结果,见图 5。

|
图 4 Western blot检测IER5免疫共沉淀复合物中的PARP1蛋白 Figure 4 Western blot analysis of IER5 and PARP1 interaction |
|
图 5 HeLa细胞验证IER5免疫共沉淀复合物中的PARP1蛋白 Figure 5 Analysis of IER5 and PARP1 interaction in HeLa cells |
讨 论
早期研究提示,IER5基因可能在肿瘤增殖或患者预后、细胞代谢、细胞有丝分裂中发挥重要的作用[6-8]。近年来的研究进一步表明,IER5基因作为DNA损伤相关基因,可作为接受放射损伤后剂量评估的生物标记物,但其参与的机制却未阐明。
本研究组前期通过RT-PCR方法验证了放射损伤后IER5基因在HeLa、AHH-1细胞mRNA水平随剂量、时间变化而波动,本实验以HeLa细胞作为研究对象,验证了IER5在蛋白水平上放射损伤后表达变化,其表达于照后4 h开始增加,12 h出现高峰,并持续至48 h,结果与前期研究结果一致。目前,DNA是公认的电离辐射主要靶目标,细胞受照后的大部分生物学改变理论上是与DNA损伤相关,通过一系列信号传导而进行DNA损伤修复、周期调控或者凋亡[4]。有研究显示,IER5蛋白于细胞质和细胞核内均有分布[9],而本研究结果观察到,在放射损伤后IER5蛋白在细胞核内有增加趋势,所以推测放射损伤后细胞核内IER5蛋白发生变化,可能参与DNA损伤修复的某一过程,同时也指引了今后深入研究的方向。
本实验进一步采用免疫共沉淀方法富集到IER5相互作用蛋白,通过质谱分析及DAVID在线蛋白质功能注释,共筛选出10个与DNA代谢、损伤修复相关功能蛋白,比如B淋巴细胞瘤2相关转录因子参与Bcl-2家族蛋白转录调控[10],DNA解旋酶Ⅱ参与DNA解螺旋过程[11],核内不均一核糖核蛋白E1在DNA复制及调控转录翻译都有作用[12],X射线交叉互补修复蛋白6,即KU70蛋白,是DNA双链断裂非同源末端连接修复通路中的重要因子[13],尿嘧啶DNA糖基化酶和3-磷酸甘油醛脱氢酶在细胞核内起尿嘧啶糖基化酶的作用[14],无义转录稳定调节因子和外切核酸酶2主要参与mRNA的调控[15],组蛋白H3是维持染色体结构重要的核蛋白[16]。其中,最感兴趣的是PARP1蛋白,有研究证实,PARP1是催化多聚ADP核糖化的蛋白酶,在基因转录、DNA损伤修复、细胞凋亡等中起着重要的作用[17]。更为通俗的理解是PARP1参与到细胞众多的应激应答过程中,在DNA损伤修复方面,PARP1主要在DNA单链断裂修复或碱基切除修复途径中发挥作用,但PARP1活性被抑制后会因DNA单链损伤未修复而后发生DNA复制,造成DNA双链损伤。尤其在BRCA1突变的肿瘤中更加明显,因无法对DNA双链断裂进行修复,其对PARP1抑制剂更为敏感,DNA损伤程度累积及异常的DNA末端连接,而导致细胞生长阻滞及凋亡[18]。在女性恶性肿瘤前两位的乳腺癌及卵巢癌的治疗中,PARP1的抑制剂已成功用于临床[19-20]。本研究通过质谱分析并已初步验证IER5与PARP1存在相互作用,而且放射损伤后两者相互作用增强,因此,设想在宫颈癌中高表达的IER5与PARP1存在相互作用,能否在宫颈癌的治疗中有新的治疗突破点。接下来将继续验证两者相互作用方式、时效关系及功能影响等,争取有更深一层的研究突破和进展。
利益冲突 本文研究者及家属, 未因进行该研究而接受任何不正当的职务或经费利益, 在此对研究的独立性和科学性予以保证作者贡献声明 于新平执行项目的研究, 撰写论文;马腾、周平坤、吴玉梅设计研究方案,分析讨论研究结果,修改论文
| [1] | Siegel R, Ma J, Zou Z, et al. Cancer statistics, 2014[J]. CA Cancer J Clin, 2014, 64 (1): 9-29. DOI:10.3322/caac.21208. |
| [2] | Long XH, Zhao ZQ, He XP, et al. Dose-dependent expression changes of early response genes to ionizing radiation in human lymphoblastoid cells[J]. Int J Mol Med, 2007, 19 (4): 607-615. DOI:10.3892/ijmm.19.4.607. |
| [3] | Tavakoli H, Manoochehri M, Modarres MSM, et al. Dose-dependent and gender-related radiation-induced transcription alterations of Gadd45a and Ier5 inhuman lymphocytes exposed to gamma ray emitted by (60) Co[J]. Radiat Prot Dosim, 2013, 154 (1): 37-44. DOI:10.1093/rpd/ncs164. |
| [4] | Ding KK, Shang ZF, Hao C, et al. Induced expression of the IER5 gene by gamma-ray irradiation and its involvement in cell cycle checkpoint control and survival[J]. Radiat Environ Biophys, 2009, 48 (2): 205-213. DOI:10.1007/s00411-009-0213-4. |
| [5] | Ahn WS, Bae SM, Lee JM, et al. Searching for pathogenic gene functions to cervical cancer[J]. Gynecol Oncol, 2004, 93 (1): 41-48. DOI:10.1016/j.ygyno.2003.11.031. |
| [6] | Asano Y, Kawase T, Okabe A, et al. IER5 generates a novel hypo-phosphorylated active form of HSF1 and contributes to tumorigenesis[J]. Sci Rep, 2016, 6 : 19174 DOI:10.1038/srep19174. |
| [7] | Jansová E, Koutná I, Krontorád P, et al. Comparative transcriptome maps: a new approach to the diagnosis of colorectal carcinoma patients using cDNA microarrays[J]. Clin Genet, 2006, 69 (3): 218-227. DOI:10.1111/j.1399-0004.2006.00588.x. |
| [8] | Wouters J, Stas M, Govaere O, et al. Gene expression changes in melanoma metastases in response to high-dose chemotherapy during isolated limb perfusion[J]. Pigment Cell Melanoma Res, 2012, 25 (4): 454-465. DOI:10.1111/j.1755-148X.2012.01004.x. |
| [9] | Ishikawa Y, Kawabata S, Sakurai H. HSF1 transcriptional activity is modulated by IER5 and PP2A/B55[J]. FEBS Lett, 2015, 589 (10): 1150-1155. DOI:10.1016/j.febslet.2015.03.019. |
| [10] | Kasof GM, Goyal L, White E. Btf, a novel death-promoting transcriptional repressor that interacts with Bcl-2-related proteins[J]. Mol Cell Biol, 1999, 19 (6): 4390-4404. DOI:10.1128/MCB.19.6.4390. |
| [11] | Zhang S, Grosse F. Domain structure of human nuclear DNA helicase Ⅱ (RNA helicase A)[J]. J Biol Chem, 1997, 272 (17): 11487-11494. DOI:10.1074/jbc.272.17.11487. |
| [12] | Leffers H, Dejgaard K, Celis JE. Characterisation of two major cellular poly (rC)-binding human proteins, each containing three K-homologous (KH) domains[J]. Eur J Biochem, 1995, 230 (2): 447-453. DOI:10.1111/ejb.1995.230.issue-2. |
| [13] | Lee KJ, Saha J, Sun J, et al. Phosphorylation of Ku dictates DNA double-strand break (DSB) repair pathway choice in S phase[J]. Nucleic Acids Res, 2016, 44 (4): 1732-1745. DOI:10.1093/nar/gkv1499. |
| [14] | Hanauer A, Mandel JL. The glyceraldehyde 3 phosphate dehydrogenase gene family: structure of a human cDNA and of an X chromosome linked pseudogene; amazing complexity of the gene family in mouse[J]. EMBO J, 1984, 3 (11): 2627-2633. |
| [15] | Nagarajan VK, Jones CI, Newbury SF, et al. XRN 5'→3' exoribonucleases: structure, mechanisms and functions[J]. Biochim Biophys Acta, 2013, 1829 (6-7): 590-603. DOI:10.1016/j.bbagrm.2013.03.005. |
| [16] | Ederveen TH, Mandemaker IK, Logie C. The human histone H3 complement anno 2011[J]. Biochim Biophys Acta, 2011, 1809 (10): 577-586. DOI:10.1016/j.bbagrm.2011.07.002. |
| [17] | Gassman NR, Stefanick DF, Kedar PS, et al. Hyperactivation of PARP triggers nonhomologous end-joining in repair-deficient mouse fibroblasts[J]. PLoS One, 2012, 7 (11): e49301 DOI:10.1371/journal.pone.0049301. |
| [18] | Wang X, Liu L, Montagna C, et al. Haploinsufficiency of Parp1 accelerates Brca1-associated centrosome amplification, telomere shortening, genetic instability, apoptosis, and embryonic lethality[J]. Cell Death Differ, 2007, 14 (5): 924-931. DOI:10.1038/sj.cdd.4402105. |
| [19] | Gunderson CC, Moore KN. Olaparib: an oral PARP-1 and PARP-2 inhibitor with promising activity in ovarian cancer[J]. Future Oncol, 2015, 11 (5): 747-757. DOI:10.2217/fon.14.313. |
| [20] | Drew Y. The development of PARP inhibitors in ovarian cancer: from bench to bedside[J]. Br J Cancer, 2015, 113 (Suppl 1): S3-9. DOI:10.1038/bjc.2015.394. |
 2017, Vol. 37
2017, Vol. 37


