食管癌死亡率在我国所有恶性肿瘤中排第4位,大多数患者发现时已属中晚期[1]。放射治疗是食管癌主要的治疗手段之一,放射抗拒是引起食管癌放射治疗失败的重要原因[2]。如何提高食管癌放射敏感性,成为当前治疗食管癌的一个亟待解决的问题。生物信息学是近年来的热点研究工具,广泛应用于分子生物学各个领域,包括基因表达谱的分析、基因组序列信息、蛋白质序列和结构、代谢和疾病发生途径的信号通路研究等及相互作用分析[3]。
本研究利用GEO2R分析来自基因芯片公共数据库(GEO)的食管癌放射抗拒相关芯片数据,获得差异表达基因,进一步利用DAVID、String等在线分析软件对这些差异基因进行Gene Ontology分析、Kegg Pathway分析及蛋白间相互作用的网络分析,从整体上筛选食管癌放射抗拒关键基因及信号通路,有利于进一步了解食管癌放射抗拒发生机制,为食管癌治疗开辟新思路。
材料与方法1.细胞与试剂:TE13细胞系由西安交通大学第二附属医院实验室提供,具有放射抗拒性TE13R细胞系由本实验室构建并保存。RPMI 1640干粉购自美国GIBCO公司;胎牛血清购于美国Hyclone公司;TRIzol试剂购自美国Invitrogen公司。
2.基因表达谱数据获取:放射抗拒食管癌及放射敏感食管癌细胞基因表达谱来源于NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo),登录号分别为GSE61772和GSE61620,由Xiong等[4]提交。GSE61772由6个样本构成,样本编号GSM1513731~GSM1513733放射敏感细胞组,GSM1513734~GSM1513736为放射抗拒细胞组;GSE61620由6个样本构成,GSM1509454~GSM1509456为放射敏感细胞组,GSM1509457~GSM1509459为放射抗拒细胞组。
3.差异表达基因的筛选:GEO2R是利用R语言中GEOquery和limma程序包,应用t检验筛选差异表达基因[5]。本研究利用GEO2R (https://www.ncbi.nlm.nih.gov/geo/geo2r/)在线分析软件筛选GSE61772和GSE61620中放射抗拒食管癌细胞及放射敏感食管癌细胞间差异表达基因。将GSE61772和GSE61620中获取的差异基因导入韦恩图(http://bioinformatics.psb.ugent.be/webtools/Venn/)在线制作软件,获取共有的差异基因。基因表达谱数据具有高维度和小样本量的特点,需要多重假设检验(multipletest)控制,本研究中,多重假设检验控制采用Benjamin和Hochberg提出的假阳性率控制法[6]。差异基因筛选标准:adjusted P < 0.05,Fold change即FC≥2。
4.差异基因生物信息学分析:进一步运用DAVID(https://david.ncifcrf.gov/)在线分析软件对这些差异表达的基因进行GO BP分析、Kegg通路分析。采用改良Fisher精确检验的统计学方法,分析差异表达基因是否在某个功能节点上出现过,得出有显著关联的基因功能类或通路,按其P值排序后输出功能类表格,对这些基因进行生物学解释。P<0.05,计数>2具有统计学意义。进一步运用String(http://www.string-db.org/)在线分析软件对这些差异表达的基因进行Protein-Protein interaction分析。
5.细胞培养及RT-PCR方法检测CHN2基因表达变化:首先,将TE13和TE13R培养于含10%胎牛血清、250 μl青霉素、400 μl链霉素(购于上海碧云天公司)的RPMI 1640的培养基中,置于37℃、5% CO2中,每2~3天更换培养液1次。待细胞80%~90%贴壁进行传代。其次,按照试剂盒说明分别检测CHN2在TE13和TE13R表达。内参照为GAPDH。凝胶成像仪获取图片,经Quantity One软件对CHN2基因与其内参进行灰度比值半定量比较。
结果1.食管癌放射抗拒相关基因的筛选:采用GEO2R对GSE61772和GSE61620中数据进行分析,GSE61772中发现有437个差异基因,GSE61620中发现有1 196个差异基因。进一步运用韦恩图在线分析软件对GSE61772和GSE61620中共有差异基因进行分析发现:两组数据中共有49个差异基因,其中上调表达37个,下调表达12个(表 1)。其中,前12个是在放射抗拒细胞中低表达基因,后面37个为高表达基因。
|
|
表 1 两组数据库中均有的49个差异表达基因 Table 1 A total of 49 differentially expressed genes in both two databases of GSE61772 and GSE61620 |
2.共有的差异表达基因GO分析和Kegg通路分析:通过DAVID在线分析工具对49个共有差异表达基因进行生物信息学分析发现,差异基因参与生物学过程24条,主要集中在调节多细胞生物合成、离子转运、含氧化合物反应、DNA合成、代谢、调节细胞增殖、应激反应等,其中调节多细胞生物合成、离子转运、含氧化合物反应涉及基因较多(表 2)。差异基因参与主要生物学通路是Wnt信号通路,涉及基因是GPC4、PRICKLE1、CSNK1A1L。
|
|
表 2 共有的差异表达基因参与的生物学过程 Table 2 Biological processes regulated by the common differentially expressed genes |
3.共有的差异表达基因间相互作用网络分析:将49个基因导入在线分析软件String中发现12个基因间存在相互作用,分别是CPVL、CHN2、EPHA3、PRICKLE1、GPC4、COL4A3、OR6S1、OR1S2、KCNK2、KCNJ8、ZNF705D和CSNK1A1L。存在8条相互作用连接。其中CHN2与多个基因间存在相互作用(图 1)。

|
图 1 共有差异表达基因对应蛋白间相互作用 注:每个点代表对应蛋白,蛋白间每条不同线条连接代表预测的功能连接,线条颜色代表来自不同类型证据 Figure 1 Interactions of protein to protein corresponding to the common differentially expressed genes |
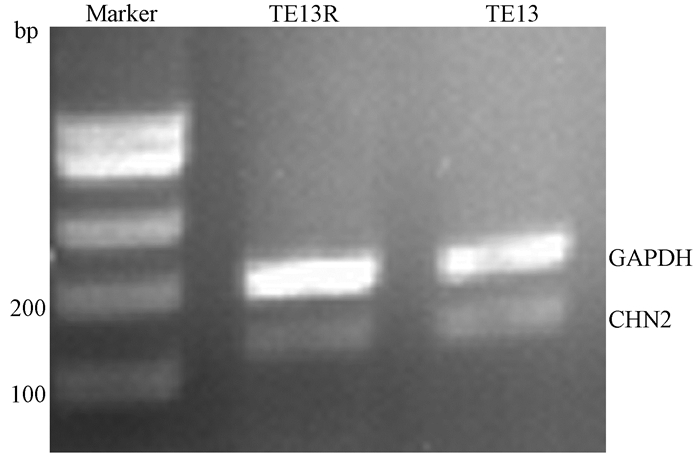
4.CHN2在不同放射敏感性食管癌细胞系中基因表达变化:半定量RT-PCR结果显示,CHN2在TE13R细胞及TE13细胞中均有表达。CHN2的mRNA在TE13R及TE13细胞中的表达分别为0.46±0.04和0.51±0.12,差异无统计学意义(P>0.05),见图 2。
|
图 2 CHN2在不同放射敏感性食管癌细胞系中基因表达变化 Figure 2 Expression of CHN2 in different esophageal carcinoma cell lines with different radiosensitivities |
讨论
食管癌是最常见胃肠道肿瘤之一。放射治疗是食管癌主要且有效治疗方法之一,然而放射治疗临床效果却并不满意。放射抗拒是影响食管癌放射敏感性最大障碍之一[7]。因此,寻找食管癌放射抗拒相关分子,探索其发生机制,针对每位患者制定个体化治疗方案显得尤为重要。随着人类基因组计划的完成及基因工程技术的发展,通过检测基因及蛋白预测,诊断疾病以及指导个体化治疗成为近来研究热点[8]。关于食管癌放射敏感性相关的分子研究已有许多报道,但由于大多数分子标志物检测手段复杂、变化大,以至于大多数研究仍没有一致结果[9]。
近年来采用生物信息学方法在分子水平上进行数据挖掘,为研究包括肿瘤在内的各种疾病的分子发病机制提供了新的思路[10]。本研究利用GEO2R分析来自基因芯片公共数据库(GEO)的食管癌放射抗拒相关芯片数据,筛选差异表达基因;通过生物学过程注释、信号通路富集分析及蛋白质相互作用分析,以挖掘与食管癌放射抗拒相关基因及信号通路,在分子水平研究食管癌放射抗拒机制,为提高食管癌放射敏感性及个体化治疗提供依据。
GEO2R是一种源于R语言程序的分析基因差异表达工具。本研究利用GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)在线分析工具分别分析GSE61772和GSE61620数据中放射抗拒食管癌细胞及放射敏感食管癌细胞间差异表达基因,进一步用韦恩作图工具选择两组芯片数据中共有差异基因,获得49个共有基因,其中12个是在放射抗拒细胞中低表达基因,37个为高表达基因。这些基因可能与食管癌放射抗拒相关,为食管癌放射抗拒发生有关基因提供理论基础。这些基因主要参与调节多细胞生物合成、离子转运、含氧化合物反应、DNA合成、代谢、调节细胞增殖、应激反应等,其中调节多细胞生物合成、离子转运、含氧化合物反应涉及基因较多,与以往研究一致[11]。
经DAVID及Kegg数据库进行通路富集分析发现,所选基因主要参与Wnt信号通路。说明该信号通路可能在食管癌放射抗拒发生中发挥一定作用。有研究采用全基因芯片对食管癌放射抗拒细胞系及其亲本细胞比较,发现Wnt通路上有19个基因出现表达差异[12]。Wnt高表达引起Wnt信号通路激活,导致β-联蛋白的积蓄,引起下游cyclin-D1和WISP1的高表达及细胞增殖加速,从而使食管癌细胞具有放射抗性。Wnt信号通路可能与食管癌放射抗拒发生相关[13]。本研究发现Wnt信号通路上GPC4、PRICKLE1、CSNK1A1L基因在食管癌放射抗拒细胞系与放射敏感细胞间表达存在差异。
本研究进一步对共有差异基因进行蛋白相互作用网络分析,发现基因CPVL、CHN2、EPHA3、PRICKLE1、GPC4、COL4A3、OR6S1、OR1S2、KCNK2、KCNJ8、ZNF705D和CSNK1A1L间存在相互作用,其中CHN2是关键节点。CHN2活化在细胞增殖和迁徙中发挥重要作用。具有正向调控GTP活性作用,CHN2基因参与代谢过程。这些功能可能与食管癌放射敏感性相关,仍需进一步证实。本研究采用RT-PCR技术检测CHN2在不同放射敏感性食管癌细胞系中基因表达变化,结果显示在TE13R细胞及TE13细胞中均有表达,其在不同放射敏感性食管癌细胞中表达差异无统计学意义。后期研究笔者将通过检测食管癌患者放疗前活检标本中CHN2表达,探讨其与食管癌放射敏感性关系,为CHN2在食管癌患者放射治疗中价值做出评估。总之,本研究进一步证实了放射抗拒发生是多基因共同参与结果。目前关于上述这些基因与放射抗拒间关系未有报道,这些基因仍需进一步研究。
综上所述,本研究通过对GEO中食管癌放射抗拒相关芯片数据进行生物信息学分析,发现Wnt信号通路可能是食管癌放射抗拒发生过程中重要的信号通路;筛选出49个差异基因可能与食管癌放射抗拒发生相关,基因CHN2可能与食管癌放射抗拒发生相关。总之,利用生物信息学方法能够能有效地筛选和再次分析基因芯片数据,挖掘食管癌放射抗拒关键分子及通路,为筛选食管癌放射抗拒相关分子及食管癌个体化治疗开辟了新的思路。后续研究中,课题组将会对本研究中筛选的基因在食管癌患者放射治疗中价值做出评估。
利益冲突 无作者贡献声明 郭亚参与该项研究设计、撰写、修改及统计学分析;马红兵参与研究设计及论文修改;王宝峰、任宏涛参与数据处理及背景信息搜索
| [1] | Wang JB, Fan JH, Liang H, et al. Attributable causes of esophageal cancer incidence and mortality in China[J]. PLoS One, 2012, 7 (8): e42281 DOI:10.1371/journal.pone.0042281. |
| [2] |
张红, 吴清明. 食管癌放射抗拒的细胞信号通路研究进展[J].
肿瘤防治研究, 2014, 41 (4): 425-427. Zhong H, Wu QM. Research progress of cell signaling pathways of esophageal cancer radioresistance[J]. Cancer Res Prev Treat, 2014, 41 (4): 425-427. DOI:10.3971/j.issn.1000-8578.2014.04.032. |
| [3] |
蔡祖艾, 王琪, 张玮, 等. 以基因通路富集法探索上皮性卵巢癌多药耐药相关生物学通路及功能性SNP位点[J].
中国癌症防治杂志, 2014 (2): 127-132. Cai ZA, Wang Q, Zhang W, et al. Using gene-set enrichment pathway analysis to identify signaling pathways and functional single nucleotide polymorphisms related to multi-drug resistance in epithelial ovarian cancer[J]. Chin J Oncol Prev Treat, 2014 (2): 127-132. DOI:10.3969/j.issn.1674-5671.2014.02.06. |
| [4] | Xiong W, Zhao J, Yu H, et al. Elevated expression of AKR1C3 increases resistance of cancer cells to ionizing radiation via modulation of oxidative stress[J]. PLoS One, 2014, 9 (11): e111911 DOI:10.1371/journal.pone.0111911.eCollection2014. |
| [5] | Huber W, Carey VJ, Gentleman R, et al. Orchestrating high-throughput genomic analysis with Bioconductor[J]. Nat Methods, 2015, 12 (2): 115-121. DOI:10.1038/nmeth.3252. |
| [6] |
刘晋, 张涛, 李康. 多重假设检验中FDR的控制与估计方法[J].
中国卫生统计, 2012, 29 (2): 305-308. Liu J, Zhang T, Li K. Control and estimation of FDR in multiple hypothesis testing[J]. Chin J Health Stat, 2012, 29 (2): 305-308. DOI:10.3969/j.issn.1002-3674.2012.02.054. |
| [7] |
胡乐林, 王俊杰. 食管癌放射敏感性标志物的研究进展[J].
癌症进展, 2015, 13 (6): 593-596. Hu LL, Wang JJ. Study on radiation sensitivity markers of esophageal carcinoma[J]. Oncol Prog, 2015, 13 (6): 593-596. DOI:10.11877/j.issn.1672-1535.2015.13.06.07. |
| [8] | Yan JY, Jiang F, Liu ZZ, et al. The enhancement of radiosensitivity in human esophageal squamous cell carcinoma cells by zoledronic acid and its potential mechanism[J]. Cytotechnology, 2014, 66 (1): 17-25. DOI:10.1007/s10616-012-9532-4. |
| [9] |
邓家营, 赵快乐. 食管癌放射敏感相关基因研究进展[J].
肿瘤, 2014, 34 (1): 97-100. Deng JY, Zhao KL. Progress in study onradiosensitivity-related genes in esophageal cancer[J]. Tumor, 2014, 34 (1): 97-100. DOI:10.3781/j.issn.1000-7431.014.01.016. |
| [10] |
蒋燕明, 李力. 宫颈上皮内瘤变进展相关基因的生物信息学分析[J].
中国肿瘤临床, 2016, 43 (19): 840-844. Jiang YM, Li L. Bioinformatics analysis of genes related to the progress of cervical intraepithelial neoplasia[J]. Chin J Clin Oncol, 2016, 43 (19): 840-844. DOI:10.3969/j.issn.1000-8179.2016.19.614. |
| [11] |
李轩. 肿瘤放射敏感相关基因的研究进展[J].
复旦学报(医学版), 2014, 41 (4): 560-564. Li X. Research development of the correlative genes in cancer radiosensitization[J]. Fudan Univ J Med Sci, 2014, 41 (4): 560-564. DOI:10.3969/j.issn.1672-8467.2014.04.026. |
| [12] |
苏华芳, 万秋燕, 邹燕, 等. 具干细胞特性食管癌耐放射细胞株基因表达分析[J].
肿瘤学杂志, 2010, 16 (1): 40-45. Su HF, Wan QY, Zou Y, et al. An analysis of the gene expression profiles of radioresistance and stem cell phenotype in the esophageal carcinoma cells[J]. J Oncol, 2010, 16 (1): 40-45. |
| [13] |
李海英, 张力, 潘欢乐, 等. Wnt信号通路在食管癌细胞放射抗拒性形成中的作用[J].
中国病理生理杂志, 2012, 28 (9): 1623-1626. Li HY, Zhang L, Pan HL, et al. Role of Wnt signaling pathway in development of radioresistance in esophageal cancer[J]. Chin J Pathophysiol, 2012, 28 (9): 1623-1626. DOI:10.3969/j.issn.1000-4718.2012.09.015. |
 2017, Vol. 37
2017, Vol. 37


