2. 215123 苏州大学医学部放射医学与防护学院
2. School of Radiation Medicine and Protection, Medical College of Soochow University, Suzhou 215123, China
作为遗传信息的载体,DNA是电离辐射影响生物体功能的主要靶点[12],而质子放疗中作用于肿瘤区域的质子能量大多处于布拉格峰附近,因此,研究该能量段的质子与DNA相互作用的特点规律对质子放疗具有重要意义。本文基于蒙特卡罗程序包Geant4及Geant4-DNA物理模型对比研究了布拉格峰附近7档能量的质子对DNA损伤的差异,这对确定质子治疗中质子能量的选择和了解肿瘤杀灭情况具有一定的指导意义。
材料与方法1. 蒙特卡罗模拟:采用欧洲核子研究中心主持开发的开源蒙卡程序包Geant4(版本96.P0.2)进行[3]。在模拟程序中,构建半径387 μm、体积24×102 μm3的球体细胞核模型,DNA占细胞核体积的16%[4-6],DNA无固定形状,随机分布于核中[4-5]。核外为球体的世界体和介质层,世界体半径为细胞核半径的11倍,介质层半径为细胞核半径的105倍。世界体、介质层、细胞核及DNA的介质均设为液态水。物理过程考虑质子和电子两种粒子,介质层外采用标准物理模型,截止范围1 μm;介质层及核内联合采用标准物理模型和Geant4DNA物理模型[7],截止范围1 nm,动能>10 MeV的电子和>99 MeV的质子采用标准物理模型,低于上述能量值时采用Geant4DNA物理模型。电子能量低于5 eV时终止模拟。模拟的质子分为20、15、10、50、10、0.8和0.6 MeV共7档能量,其随机分布于细胞核表面,并按余弦定理射向核内。记录质子及次级电子与核介质历次相互作用的位点坐标值和对应能量沉积值及质子编号,并写入EnergyDep.dat文件中。共进行9次独立的模拟,每次模拟不同能量的质子各1 000。
2. 确定损伤位点:DNA链是否断裂取决于质子及次级电子在DNA链上的能量沉积。自EnergyDep.dat文件中随机选取16%的位点数据,作为作用于DNA的位点,并根据各位点对应能量沉积值判断其是否引起DNA链的断裂。质子或次级电子导致DNA链断裂的概率(P)与能量沉积值(E,eV)的关系如式(1)所示[4, 8],而最终是否会发生断裂,则由对应P值与系统随机生成的0~1之间的随机数进行对比,若大于系统生成的随机数,则发生断裂,否则不发生断裂。将导致DNA链断裂的位点信息记录在文件BreakDot.dat文件中,这些位点对应于DNA链断裂的位置,其在DNA上形成单链断裂(SSB)。
| $\left\{ {\matrix{ {P = 0,E \le 5.0} \cr {P = \left( {E - 5.0} \right)/\left( {37.5 - 5.0} \right),5.0 < E < 37.5} \cr {P = 1,E \ge 37.5} \cr } } \right.$ | (1) |
3. 损伤集簇及损伤类型的确定:运用基于密度的空间聚类算法(DBSCAN)处理BreakDot.dat文件中数据[9-10]。放射生物学中,位于不同链上相差10个碱基对(约32 nm)内的两个SSB可形成一个双链断裂(DSB)。因此,将算法中的两个主要参数,半径r和阈值m,分别设置为32 nm和2。统计出文件中各个质子及其次级电子所形成的独立损伤位点(噪声,即距离其他位点>32 nm的位点)数、集簇(类,由相互距离在32 nm以内的位点共同构成)数及各集簇的尺寸(类的大小,即类中包含的点的数目)。由于>6的损伤集簇数目较少,将这些损伤集簇统计到尺寸为6的集簇。
本研究中将DNA链的SSB和DSB又分别分为简单单链断裂(SSSB)与复杂单链断裂(CSSB)和简单双链断裂(SDSB)与复杂双链断裂(CDSB)。SSSB指没有与其他损伤位点一起构成损伤集簇的损伤,即独立损伤位点所对应的损伤;CSSB指所有损伤位点均位于DNA同一链上的集簇对应的损伤;SDSB指尺寸为2,且损伤位点分别位于DNA不同链上的集簇对应的损伤;CDSB指尺寸>2,且损伤位点不位于DNA同一链上的集簇对应的损伤。以上4种损伤在生物学上修复的难易程度依次为SSSB<CSSB<SDSB<CDSB。对于集簇中的各个损伤位点,假设其以相同的概率分布于DNA的两条链上,则不同尺寸(ni)的集簇形成后3种损伤的概率分别为P(CSSB)=0.5ni-1、P(SDSB)=0.5(ni=2)和P(CDSB)=1-0.5n i-1(ni>2)。
4. 统计及绘图方法:数据以x±s表示。采用Excel 2013计算数据均值及标准差,采用OriginPro 8绘图。
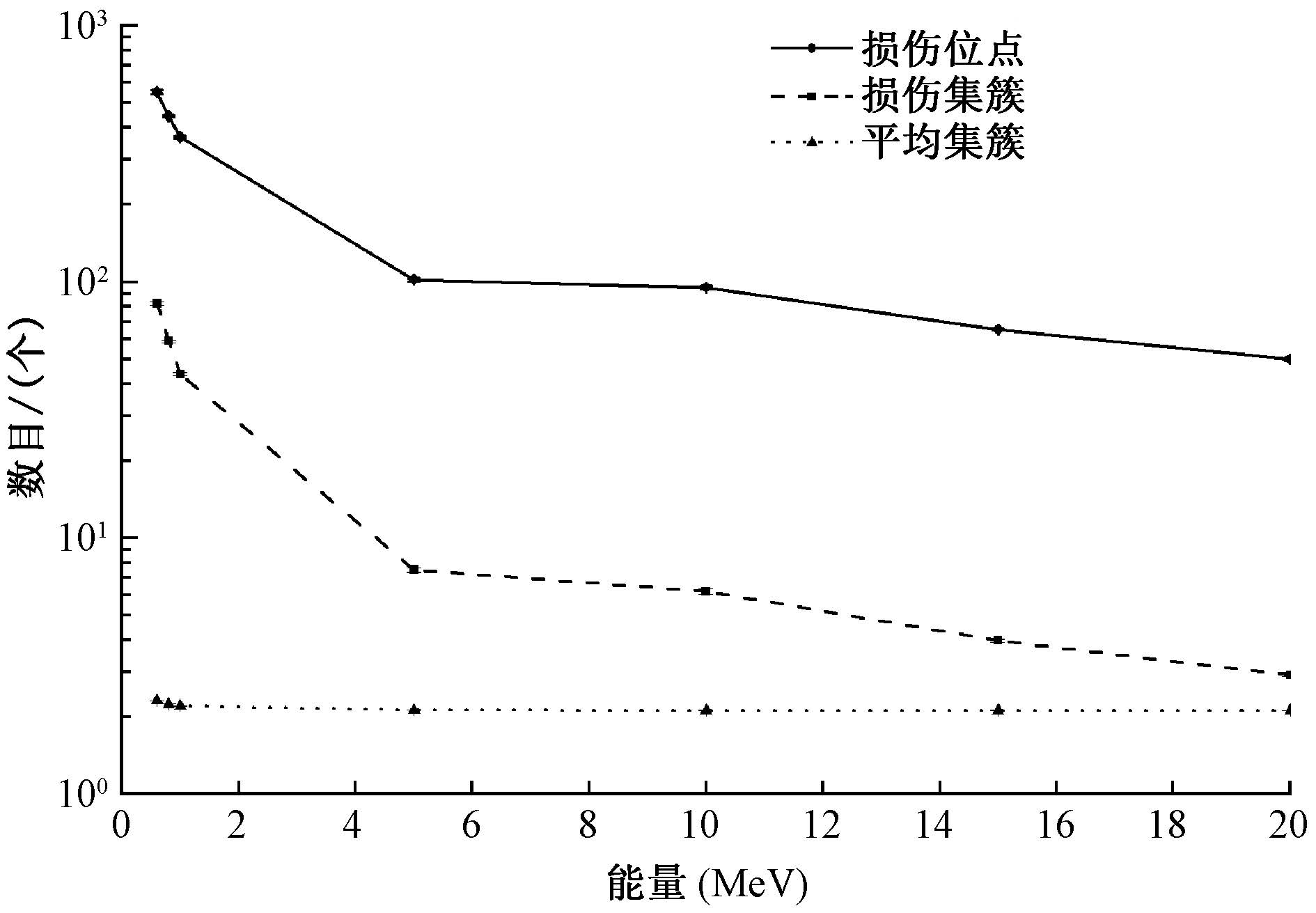
结果1. DNA损伤概况:不同能量单个质子致DNA损伤的损伤位点数、损伤集簇数及平均集簇尺寸的情况如图 1所示。可知,随质子能量的降低,损伤位点数及损伤集簇数均逐渐增加,在20 ~ 5.0 MeV时增加比较平缓,分别由(49.86±0.61)和(2.92±0.05)增加至(102.11±1.39)和(7.51±0.17),在5.0 ~ 0.6 MeV时快速增加,分别达到了(549.88±9.00)和(82.46±1.40);平均集簇尺寸也随质子能量的降低而略呈增加趋势,由(2.12±0.01)增加至(2.32±0.00)。
|
图 1 不同能量单个质子致DNA损伤概况 Figure 1 Variation in DNA damages with proton energies |
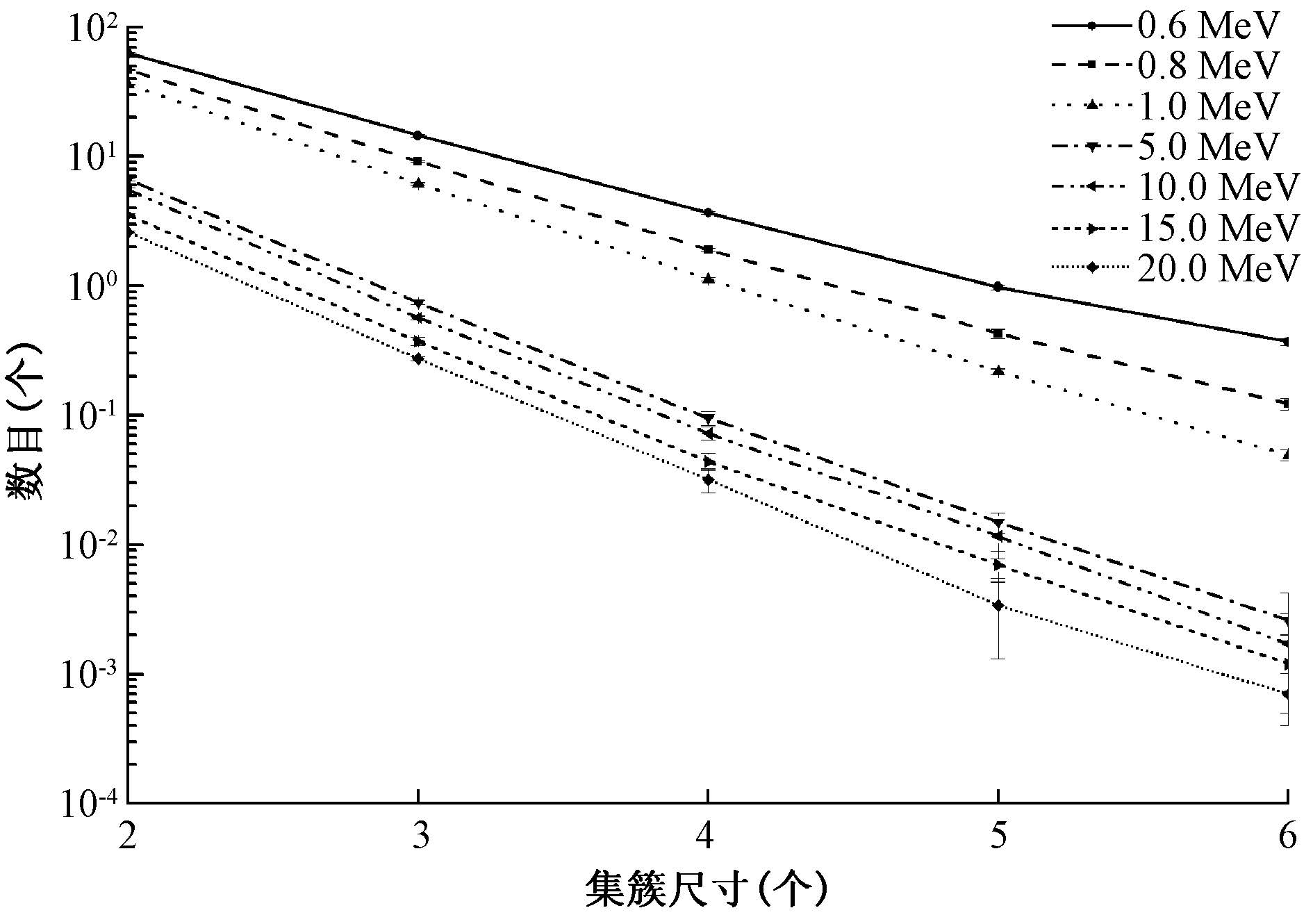
2. DNA损伤集簇分布:不同能量单个质子在细胞核模型中致DNA损伤的集簇尺寸分布如图 2所示。可知,随质子能量的降低,相同集簇尺寸的损伤均逐渐增加,但在20 ~ 50 MeV之间时增加缓慢,在50 ~ 0.6 MeV时增加显著,且随着集簇尺寸的增加,该趋势更加明显,与20 MeV的质子相比,0.6 MeV的质子在尺寸为2时,集簇数约为其24倍,而尺寸≥5时约为329倍;且能量越低,最大集簇尺寸也有增加的趋势,0.6 MeV质子可产生大小为10以上的集簇,而20 MeV的质子产生大小为6的集簇也很少。
|
图 2 不同能量单个质子致DNA损伤集簇尺寸分布 Figure 2 Variation in DNA damage with cluster size caused by protons with different energies |
3. 单个质子致DNA不同损伤分布:表 1所示为各能量的单个质子在模型中所致DNA不同损伤类型的分布。可知,在讨论能量范围内,质子的能量越低,单个质子平均导致DNA受到的4类不同损伤的个数越多,且难以修复的损伤增加更加明显,相对于20 MeV的质子,0.6 MeV质子SSSB增加约7倍,而CDSB增加了约63倍。
|
|
表 1 不同能量单个质子致DNA各类型断裂分布(x±s) Table 1 Distribution of DNA damage types caused by protons with different energies (x±s) |
4. DNA不同损伤类型概率分布:对应能量的质子致DNA各类损伤的概率分布如表 2所示。可知,在所研究能量范围内,随质子能量的降低,难以修复的DSB,尤其是CDSB所占比例越大,相对于20 MeV的质子,0.6 MeV的质子这两个概率分别增加了约3和7倍,而比较容易修复的SSB,尤其是SSSB则所占比例越小,但即使对于0.6 MeV的质子,它们依然是主要的损伤类型,发生概率分别达到约0.89和0.81。
|
|
表 2 不同能量质子致DNA各类型断裂概率分布(%) Table 2 Probability distribution of DNA damage types caused by protons with different energies (%) |
讨论
与X射线和电子相比,质子的侧向散射更少,在坪区的能量沉积及放射生物效应类似,布拉格峰处则骤然沉积大部分能量,这些特点使得质子在更加有效杀灭肿瘤的同时能很好地保护周围正常组织。国外及我国上海质子重离子医院的临床试验表明其适用于多种肿瘤,且在治疗很多肿瘤方面要明显优于传统的X射线和电子。
本研究基于Geant4DNA物理模型和所构建细胞核模型,研究了质子在布拉格峰附近7档能量时在DNA上所致损伤位点及集簇的平均分布情况和相应的DNA各类型损伤数目和相对分布。由结果可知,在研究能量范围内,随着能量的降低,单个质子所致DNA的损伤位点、损伤集簇、各尺寸的集簇数目均增加,且在能量<5 MeV时增加尤其明显,而在20 ~ 5 MeV时,则增加相对平缓,在Francis等[4]的研究中虽未给出具体数值,但通过频率对比可知本研究结果与其基本相符,所存差异可能源自细胞核模型大小不同所致;DNA的SSSB、CSSB、SDSB和CDSB数量均增加,在能量<5 MeV时增加显著,而在20 ~ 5 MeV时,增加较慢;在各类损伤中,SSSB所占比例减小,而CSSB、SDSB和CDSB均增加,且同样是能量<5 MeV时该变化明显,而在20~5 MeV时则变化较缓慢,SSB与DSB结果与Francis等[4]研究结果基本一致,但与Santos等[11]所发表结果存在较大差异,这可能是由于模型、质子入射方式及损伤判断等多重差异所致。虽然随着能量降低,DSB发生概率不断增加,但即使对于0.6 MeV的质子,其SSB在发生的所有损伤中依然占据近90%,SSSB也要达到81%以上,因此质子照射中以SSB损伤为主,这与徐辉辉等[12]的研究结果基本一致,而这些损伤很容易被修复,因此,本研究也从物理学方面解释了质子在坪区与布拉格峰区的相对生物效应基本相同,仅略高于X射线与电子。质子放疗中,假定相同的剂量沉积对应相同数目的DNA断裂点,可以预测,使布拉格峰处于靶区内,除了可以增大靶区与周围正常组织的剂量差外,对肿瘤的杀灭作用也略有提高;而对于靶区周围正常组织,在评估其剂量时,则应考虑到相对生物效应提高引起的过性损伤。
本研究讨论了质子在物理作用过程阶段所致DNA损伤的情况,虽然实际照射中还包括了化学过程和生物过程,而且研究表明,化学过程,即自由基的产生,对DNA损伤的影响更大,但物理作用过程在一定程度上可以反映不同粒子在造成生物损伤方面的差异;至于照射后的反应,由于生物体是一个非常复杂的系统,无论是肿瘤还是正常组织,均有一定的自我修复能力,并且不同组织或肿瘤的修复能力也不一样,因此在不同能量的质子照射不同生物体或同一生物体的不同部位时,有可能产生与上述结果相反的结论。本文模拟的物理作用过程结果只在一定范围内说明问题,具体情况还有待进一步生物学实验数据。
利益冲突 作者无利益冲突,排名无争议,作者的配偶、工作伙伴或子女不存在影响研究结果的财务关系,在此对研究的独立性和科学性予以保证作者贡献声明 孔栋负责本文模型构建及实验设计,论文撰写;孙亮参与实验研究,负责在论文写作过程中给予指导;崔甜甜、李向阳参与实验并负责数据整理及处理
| [1] | Goodhead DT. Initial events in the cellular effects of ionizing radiations:clustered damage in DNA[J]. Int J Radiat Biol , 1994, 65 (1) : 7-17 DOI:10.1080/09553009414550021 |
| [2] | Olive PL. The role of DNA single-and double-strand breaks in cell killing by ionizing radiation[J]. Radiat Res , 1998, 150 (5 Suppl) : S42-S51 DOI:10.2307/3579807 |
| [3] | Agostinelli S, Allison J, Amako K, et al. Gent4-a simulation toolkit[J]. Nucl Instr Sect A , 2003, 506 (3) : 250-303 DOI:10.1016/S0168-9002(03)01368-8 |
| [4] | Francis Z, Villagrasa C, Clairand I. Simulation of DNA damage clustering after proton irradiation using an adapted DBSCAN algorithm[J]. Comput Meth Prog Bio , 2011, 101 (3) : 265-270 DOI:10.1016/j.cmpb.2010.12.012 |
| [5] | Coghill MT.Radiobiological modeling using track structure analysis[D].Georgia:Georgia Institute of Technology,2012. |
| [6] | Bewley DK. A comparison of the response of mammalian cells to fast neutrons and charged particle beams[J]. Radiat Res , 1968, 34 (2) : 446-458 DOI:10.2307/3572569 |
| [7] | Incerti S, Baldacchino G, Bernal M, et al. The geant4-DNA project[J]. IJMSSC , 2010, 1 (2) : 157-164 DOI:10.1142/S1793962310000122 |
| [8] | Incerti S, Champion C, Tran HN, et al. Energy deposition in small-scale targets of liquid water using the very low energy electromagnetic physics processes of the Geant4 toolkit[J]. Nucl Instr Sect B , 2013, 306 (4) : 158-164 DOI:10.1016/j.nimb.2012.12.054 |
| [9] | 孔栋.DNA纳剂量学模型影响因素研究[D].苏州:苏州大学,2014. Kong D. Research of impact factors of nanodosimetric DNA model[D]. Suzhou:Soochow University, 2014. |
| [10] | 陈京民. 数据仓库与数据挖掘技术[M]. 北京:电子工业出版社[M]. 2003 . Chen JM. Data warehouse and data mining techniques[M]. Beijing:Publishing House of Electronics Industry[M]. 2003 . |
| [11] | Santos MD, Villagrasa C, Clairand I, et al. Influence of the DNA density on the number of clustered damages created by protons of different energies[J]. Nucl Instru Sect B , 2013, 298 : 47-54 DOI:10.1016/j.nimb.2013.01.009 |
| [12] | 徐辉辉, 隋丽, 刘晓丹, 等. 不同LET辐射致DNA集簇性损伤的研究[J]. 原子核物理评论 , 2010, 27 (4) : 469-473 Xu HH, Sui L, Liu XD, et al. Comparative studies on clustered DNA damage induced by different LET ionizing radiation[J]. Nucl Phys Rev , 2010, 27 (4) : 469-473 |
 2016, Vol. 36
2016, Vol. 36


