2. 215006 苏州大学系统生物学研究中心
2. Center for Systems Biology, Soochow University, Suzhou 215006, China
近年来,术前同期放化疗已成为局部晚期直肠癌的标准治疗模式。术前放化疗不仅提高了患者的局部控制率,同时还提高了中低位患者保肛的几率[1]。然而,并非所有患者均能从术前同期放化疗中获益,临床上迫切需要寻找能早期预测直肠癌放化疗疗效的方法。微小RNA(microRNA/miRNA)是一类大小约22个核苷酸(nts)的内源性非编码RNA,能够在转录后层次上调控基因的表达。它们在细胞分化、增殖、凋亡、生物生长发育及人类疾病发生、发展等过程中发挥着不可忽视的作用[2]。研究表明,microRNA以及相关基因的表达水平与局部晚期直肠癌术前放化疗效果密切相关[3]。本研究旨在寻找与直肠癌放化疗反应相关的microRNA及其靶基因,并通过生物信息学分析,从生物机制层面上解释直肠癌放化疗疗效的差异,寻找作为预测其疗效的肿瘤标志物,为直肠癌放化疗研究提供新的依据。
材料与方法1. MicroRNA数据:在PubMed数据库中挖掘直肠癌放化疗疗效相关的microRNA,检索关键词为:(miRNA OR microRNA) AND (colorectal cancer OR rectal cancer OR rectum cancer OR colorectal carcinoma OR rectal carcinoma) AND (chemoradiotherapy OR radiotherapy OR radiation)。选取标准包括:实验验证的可以作为预测直肠癌放化疗疗效的microRNA;通过计算模型和数据分析得到的与直肠癌放化疗疗效潜在相关的microRNA。
2. MicroRNA靶基因的鉴定:基于公开的microRNA-mRNA调控关系识别直肠癌放化疗反应相关microRNA的靶基因。其中,microRNA-mRNA调控关系来源于7个主流的microRNA靶基因数据库,包括4个实验验证的数据库:miRecords[4]、Tarbase[5]、miR2Disease[6]和miRTarbase[7],以及3个计算机预测的数据库:HOCTAR[8]、ExprTargetDB[9]和starBase[10]。
3.直肠癌相关的基因芯片数据:直肠癌相关的基因芯片数据来源于Gene Expression Omnibus (GEO, http://www.ncbi.nlm.nih.gov/geo/),数据集选择GSE35452[11],该数据集包含24个放疗敏感和22个放疗不敏感的直肠癌患者新鲜冰冻组织标本的样本数据,实验平台为Affymetrix Human Genome U133 Plus 2.0 arrays。本文下载了标准化的表达数据进行统计分析,寻找差异表达的基因。
4.基因功能及通路分析:利用DAVID[12]工具对靶基因进行基因本体论(gene ontology, GO)和KEGG信号通路(pathway)富集分析。其中,GO分析包括3个方面:即分子功能、生物过程和细胞组成。同时,本研究使用IPA(ingenuity pathway anaylsis)软件分析靶基因影响的信号代谢通路、分子网络和生物学过程。
5.统计学处理:在R平台下使用Limma软件包中的Student′s t检验方法筛选差异表达的基因。在GO及KEGG通路分析中,数据间差异比较采用ANOVA检验。KEGG和IPA通路中选择差异具有统计学意义的(P < 0.05)信号转导通路进行研究。具体的研究流程见图 1。
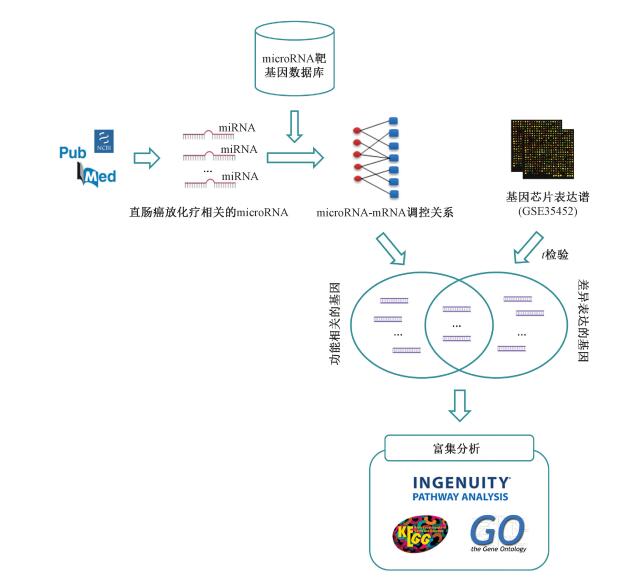
|
图 1 本研究的研究流程 Figure 1 The schematic workflow of this study |
结果
1.直肠癌放化疗疗效相关的microRNA:通过在PubMed数据库中使用相应的关键词进行高级嵌套检索,人工筛选共找到38个与直肠癌放化疗潜在相关的microRNA,见表 1。
|
|
表 1 与直肠癌放化疗疗效相关的miRNA总结 Table 1 Summary of the microRNAs associated with the chemoradiotherapy efficacy of RC |
2.直肠癌放化疗疗效相关microRNA的靶基因:基于microRNA-mRNA调控关系,筛选得到上述38个microRNA的靶基因,共计3 545个。结合直肠癌患者的基因芯片数据以及统计分析,共得到911个差异表达基因。取交集后,最终得到131个与直肠癌放化疗特异相关的基因。通过将这些基因与文献搜集的microRNA关联分析,得到了如图 2所示的调控网络。该网络中包含123个差异表达基因以及26个文献报道的microRNA。由图 2可见,约有93.9%(123/131)的差异表达基因能够被相关microRNA调控,这在一定程度上揭示了microRNA参与直肠癌放化疗过程的作用方式。除此之外,有8个差异基因不在该网络中,分别是:NBEA、KIAA0895、HECA、SCPEP1、HSD17B11、IRF8、VAPB和PCF11。从调控的角度,网络中有些基因被多个microRNA共同调控,有些基因则被某一microRNA独立调控,这种共同和独立的调控关系也分别体现出microRNA作用的协同性和特异性。
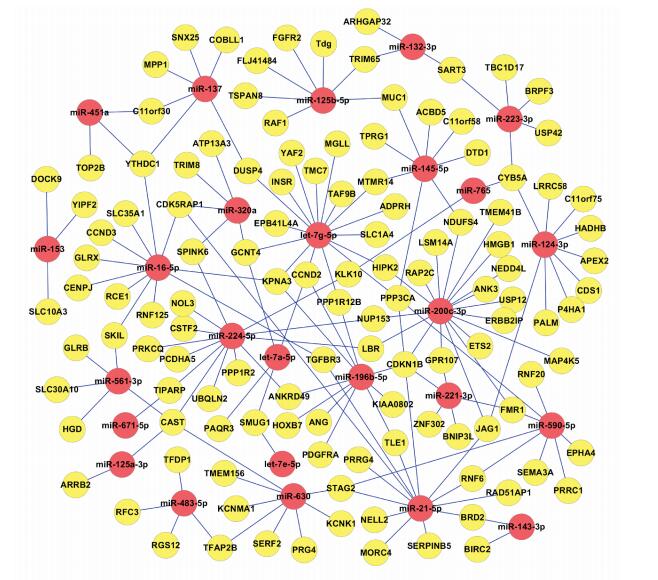
|
图 2 直肠癌放化疗相关的microRNA及其靶基因 Figure 2 MicroRNAs and their tagret genes associated with the chemoradiotherapy efficacy of RC |
3.直肠癌放化疗疗效相关的基因功能:将上述131个基因进行GO功能富集分析。结果共得到16类细胞组分注释信息,其中差异有统计学意义(P < 0.05)的有12类,前10条细胞组分注释信息主要集中于核质、核腔、细胞内细胞器内腔、细胞器的内腔、膜封闭腔、核质部分、PML小体、核小体、细胞器膜、被膜等;共得到103类生物学过程注释信息,其中有统计学意义(P < 0.05)的有51类,前10条生物学过程注释信息主要集中于成人的行走行为、酶联蛋白受体蛋白信号转导途径、细胞运动规律、氧含量的响应、细胞大小调节、跨膜受体蛋白酪氨酸激酶信号途径、蛋白质异质齐聚、有机物反应、细胞周期蛋白依赖性蛋白激酶活性的调节、成人运动行为;共得到16类分子功能注释信息,其中有统计学意义(P < 0.05)的有12类,前10条分子功能注释信息主要集中于蛋白激酶结合、水解酶活性,水解N-糖基化合物、激酶结合、跨膜受体蛋白酪氨酸激酶活性、蛋白激酶活性、酶结合、转录辅因子、羧酸结合、转录辅阻遏物的活性、核纤层蛋白结合。对每个层次前10条目进行PubMed数据库检索验证,发现细胞组分层次的基因功能有90%的条目(9/10)参与放射反应,如通过影响核质、核腔、被膜等细胞核的关键部位影响放射敏感性。生物学过程方面有60%的条目(6/10)参与放射反应,如通过调节细胞周期蛋白依赖性蛋白激酶以及相关信号转导途径影响放射敏感性。而在分子功能方面,同样有90%(9/10)的条目参与放射反应,通过影响蛋白激酶、水解酶、转录因子等影响放射敏感性,如抑制DNA依赖性蛋白激酶,可以增强细胞的放射敏感性等。以上结果在很大程度上表明这些microRNA及其调控的相关基因参与影响放射敏感性。
4.直肠癌放化疗疗效相关的信号通路:将上述131个基因进行KEGG及IPA信号转导通路富集分析。其中,KEGG共富集得到7条相关信号通路(表 2),包括碱基切除修复、细胞周期、MAPK信号通路、p53信号通路等,其中,具有统计学意义(P < 0.05)的有5条。IPA共得到245条相关信号通路,包括STAT3通路、PTEN信号通路、细胞周期蛋白和细胞周期调控、细胞周期G1/S检查点调控、NF-κB信号通路等,其中,前10条显著富集的(P < 0.05)通路如表 3所示。文献检索表明,约有80%显著富集的KEGG信号通路(4/5)以及IPA信号通路(8/10)已经被报道与放射敏感性有关。例如,Vens和Begg[13]发现,碱基切除修复通过影响DNA修复影响细胞放射敏感性。而细胞周期与放射敏感性已经被多篇文献报道,主要通过DNA损伤识别、细胞周期调控点以及DNA损伤修复参与影响放射敏感性[14]。Dent等[15]研究表明,MAPK通过多种生长因子受体,自分泌因子和Ras突变的表达参与应答放射反应。Spitzner等[16]总结了近些年的研究成果,发现STAT3通路在多种癌症中参与影响放射敏感性。Lee等[17]报道PTEN基因通过影响细胞周期调控影响放射敏感性,可以作为潜在的治疗靶点。Liu等[18]发现NF-κB与癌症进展有关,抑制其可以增加细胞放射敏感性。而p53信号通路[19]、癌症的分子机制[20]、IL-8[21]通路等通路与癌症的发生发展密切相关,也参与影响细胞的放射敏感性。虽然其余的通路目前尚未有文献报道与之相关,但仍具有一定的研究价值,需要进一步的分析论证。综上表明,这些microRNA及其调控的差异表达基因与直肠放化疗疗效高度相关,可以作为预测直肠癌放化疗疗效的潜在分子标志物。
|
|
表 2 KEGG信号通路富集结果 Table 2 KEGG pathway analysis for the target genes |
|
|
表 3 IPA信号通路富集结果 Table 3 IPA pathway analysis for the target genes |
讨论
近年来,关于直肠癌放化疗疗效的研究是一大热点,发现了很多预测放化疗疗效的基因及分子标志物。截止目前,p53、表皮生长因子、胸苷酸合成酶、Ki-67、p21、Bcl-2、bax已经被报道与放疗疗效相关[22],然而这些结果充满争议。以DNA芯片为基础的基因表达谱技术可以同时分析大量的基因数据,系统性寻找生物标志物,在预测疗效上具有巨大潜力。然而,综合国外研究发现,不同研究间的异质性较大。Ghadimi等[23]使用两个芯片平台分析了30例参与Ⅲ期临床试验的结直肠患者的组织,发现了54个在放疗敏感与不敏感的患者中显著差异表达的基因;Watanabe等[11]通过基因芯片研究了52例没有接受过化疗的术前放疗的直肠癌患者的组织,发现了33个在反应与不反应患者中显著差异表达的基因;Kim等[24]在46个结直肠癌患者组织中找到了95个在敏感与不敏感患者中显著差异表达的基因;Rimkus等[25]在43个反应与不反应的结直肠癌患者组织中发现了42个差异表达的基因;Nishioka等[26]在反应者与不反应者中找到了17个差异表达的基因。然而,Nishioka等[26]与Kim等[24]的研究结果中只有一个基因(MMP14)是相同的,Watanabe等[11]与Kim等[24]的研究也只有一个相同基因(FLNA),其余的研究并没有发现相同基因。
本研究采用整合的生物信息学分析方法,通过搜集已报道的与直肠癌密切相关的microRNA,结合microRNA-mRNA调控关系以及基因芯片表达谱数据,筛选得到与直肠癌放化疗特异相关的靶基因。通过总结国内外相关的研究成果,本研究挖掘出38个与直肠癌放化疗疗效潜在相关的microRNA,结合7个主流的microRNA靶基因数据库,得到直肠癌放化疗疗效的microRNA-mRNA调控关系,其中包括38个microRNA和3 545个靶基因。通过统计分析直肠癌术前放化疗患者手术标本的基因芯片表达谱数据,结合microRNA-mRNA调控关系,得到131个与直肠放化疗疗效特异相关的基因。
本研究认为,microRNA通过调控关键基因参与影响直肠癌放化疗疗效的生物过程。为了证实这一假设,本研究通过GO分析及信号通路富集分析探究相关基因与放化疗疗效的关系。GO分析的结果表明,这些基因在影响直肠癌放化疗疗效中发挥重要作用。同时本研究进行了更深入的探索,通过KEGG及IPA通路富集分析寻找到这些基因主要的作用途径,得到了一些关键信号通路。通过文献检索,证实了这些信号转导通路与放射敏感性高度相关,进一步解释了这些基因的作用机制,揭示了结直肠癌放化疗疗效差异的原因。
当前,临床上对直肠癌患者放化疗的选择并没有很好地考虑患者的个性化差异, 也缺乏良好的评估方法。研究表明,microRNA及其靶基因能够为疾病的临床诊断、预后以及治疗提供帮助,通过检测microRNA及基因表达谱数据,整合数据分析,建立预测模型,筛选出放化疗不敏感的患者,建议其尽早手术,在一定程度上可以对患者的预后产生积极的作用。在治疗上,目前人工合成的microRNA类似物以及microRNA阻滞剂等技术已经相对成熟,可以通过对病人体内或者瘤内注射,提高放射敏感性,让更多的患者因为放化疗获益。同时,随着基因敲除技术的不断发展,可以通过敲除某些参与调控放化疗疗效的关键基因从而影响其作用的信号通路,进一步提高直肠癌放化疗疗效的药物作用水平。当然,这方面的应用仍然需要深入的理论分析以及临床验证。因此,本研究搜集的microRNA、筛选出的基因以及分析得到的信号转导通路可以为相关研究提供一定的帮助。
从系统生物学角度,单独的分子标志物很难反映疾病在系统层次上的演化过程。癌症是一种典型的复杂疾病,因此,基于组合或网络的生物标志物将更有利于剖析和解释癌症发生发展的内在机制和外在影响。同样,直肠癌放化疗疗效也需要结合多个microRNA、基因进行整合预测。目前,本研究发现的基因以及相关的信号转导通路仅基于理论分析,虽有相关的文献报道,但仍有待进一步的实验验证它们与直肠癌放化疗疗效的相关性。
综上所述,本研究搜集了与直肠癌放化疗疗效相关的microRNA,通过对它们靶基因的生物信息学分析,发现这些基因的功能注释以及所富集的信号转导通路与直肠癌放化疗疗效密切相关,从生物机制层面上解释了直肠癌放化疗疗效的差异。同时,这些microRNA以及筛选出的差异表达基因也为预测直肠癌放化疗疗效的研究提供了基础依据。
利益冲突 本研究接受苏州市科技发展计划项目(SYS201342),苏州大学附属第二医院优势临床学科群(XKQ2015005)资助,本人与本人家属、其他研究者,未接受其他机构提供的不当利益,在此对研究的独立性和科学性予以保证作者贡献声明 彭啟亮负责文献检索、数据处理、文章撰写;林宇鑫、袁徐烨对本研究的生物信息学分析技术给予指导;朱雅群负责本研究的选题以及整体指导
| [1] | Bosset JF, Collette L, Calais G, et al. Chemotherapy with preoperative radiotherapy in rectal cancer[J]. N Engl J Med , 2006, 355 (11) : 1114-1123 DOI:10.1056/NEJMoa060829 |
| [2] | He L, Hannon GJ. MicroRNAs:small RNAs with a big role in gene regulation[J]. Nat Rev Genet , 2004, 5 (7) : 522-531 DOI:10.1038/nrg1379 |
| [3] | Svoboda M, Sana J, Fabian P, et al. MicroRNA expression profile associated with response to neoadjuvant chemoradiotherapy in locally advanced rectal cancer patients[J]. Radiat Oncol , 2012, 7 : 195 DOI:10.1186/1748-717X-7-195 |
| [4] | Xiao F, Zuo Z, Cai G, et al. miRecords:an integrated resource for microRNA-target interactions[J]. Nucleic Acids Res , 2009, 37 (Database issue) : D105-110 DOI:10.1093/nar/gkn851 |
| [5] | Sethupathy P, Corda B, Hatzigeorgiou AG. TarBase:A comprehensive database of experimentally supported animal microRNA targets[J]. RNA , 2006, 12 (2) : 192-197 DOI:10.1261/rna.2239606 |
| [6] | Jiang Q, Wang Y, Hao Y, et al. miR2Disease:a manually curated database for microRNA deregulation in human disease[J]. Nucleic Acids Res , 2009, 37 (Database issue) : D98-104 DOI:10.1093/nar/gkn714 |
| [7] | Hsu SD, Lin FM, Wu WY, et al. miRTarBase:a database curates experimentally validated microRNA-target interactions[J]. Nucleic Acids Res , 2011, 39 (Database issue) : D163-169 DOI:10.1093/nar/gkq1107 |
| [8] | Gennarino VA, Sardiello M, Avellino R, et al. MicroRNA target prediction by expression analysis of host genes[J]. Genome Res , 2009, 19 (3) : 481-490 DOI:10.1101/gr.084129.108 |
| [9] | Gamazon ER, Im HK, Duan S, et al. Exprtarget:an integrative approach to predicting human microRNA targets[J]. PLoS One , 2010, 5 (10) : e13534 DOI:10.1371/journal.pone.0013534 |
| [10] | Yang JH, Li JH, Shao P, et al. starBase:a database for exploring microRNA-mRNA interaction maps from Argonaute CLIP-Seq and Degradome-Seq data[J]. Nucleic Acids Res , 2011, 39 (Database issue) : D202-209 DOI:10.1093/nar/gkq1056 |
| [11] | Watanabe T, Kobunai T, Akiyoshi T, et al. Prediction of response to preoperative chemoradiotherapy in rectal cancer by using reverse transcriptase polymerase chain reaction analysis of four genes[J]. Dis Colon Rectum , 2014, 57 (1) : 23-31 DOI:10.1097/01.dcr.0000437688.33795.9d |
| [12] | Huang dW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc , 2009, 4 (1) : 44-57 DOI:10.1038/nprot.2008.211 |
| [13] | Vens C, Begg AC. Targeting base excision repair as a sensitization strategy in radiotherapy[J]. Semin Radiat Oncol , 2010, 20 (4) : 241-249 DOI:10.1016/j.semradonc.2010.05.005 |
| [14] | Wilson GD. Radiation and the cell cycle, revisited[J]. Cancer Metastasis Rev , 2004, 23 (3-4) : 209-225 DOI:10.1023/B.CANC.0000031762.91306.b4 |
| [15] | Dent P, Yacoub A, Fisher PB, et al. MAPK pathways in radiation responses[J]. Oncogene , 2003, 22 (37) : 5885-5896 DOI:10.1038/sj.onc.1206701 |
| [16] | Spitzner M, Ebner R, Wolff HA, et al. STAT3:A novel molecular mediator of resistance to chemoradiotherapy[J]. Cancers (Basel) , 2014, 6 (4) : 1986-2011 DOI:10.3390/cancers6041986 |
| [17] | Lee C, Kim JS, Waldman T. PTEN gene targeting reveals a radiation-induced size checkpoint in human cancer cells[J]. Cancer Res , 2004, 64 (19) : 6906-6914 DOI:10.1158/0008-5472.CAN-04-1767 |
| [18] | Liu YC, Chiang IT, Hsu FT, et al. Using NF-κB as a molecular target for theranostics in radiation oncology research[J]. Expert Rev Mol Diagn , 2012, 12 (2) : 139-146 DOI:10.1586/erm.12.2 |
| [19] | Chiu SJ, Hsu TS, Chao JI. Expression of securin promotes colorectal cancer cell death via a p53-independent pathway after radiation[J]. Chem Biol Interact , 2007, 170 (3) : 153-161 DOI:10.1016/j.cbi.2007.07.015 |
| [20] | Vanhoefer U. Molecular mechanisms and targeting of colorectal cancer[J]. Semin Oncol , 2005, 32 (6 Suppl 8) : 7-10 DOI:10.1053/j.seminoncol.2005.07.018 |
| [21] | Meeren AV, Bertho JM, Vandamme M, et al. Ionizing radiation enhances IL-6 and IL-8 production by human endothelial cells[J]. Mediators Inflamm , 1997, 6 (3) : 185-193 DOI:10.1080/09629359791677 |
| [22] | Akiyoshi T, Kobunai T, Watanabe T. Predicting the response to preoperative radiation or chemoradiation by a microarray analysis of the gene expression profiles in rectal cancer[J]. Surg Today , 2012, 42 (8) : 713-719 DOI:10.1007/s00595-012-0223-8 |
| [23] | Ghadimi BM, Grade M, Difilippantonio MJ, et al. Effectiveness of gene expression profiling for response prediction of rectal adenocarcinomas to preoperative chemoradiotherapy[J]. J Clin Oncol , 2005, 23 (9) : 1826-1838 DOI:10.1200/JCO.2005.00.406 |
| [24] | Kim IJ, Lim SB, Kang HC, et al. Microarray gene expression profiling for predicting complete response to preoperative chemoradiotherapy in patients with advanced rectal cancer[J]. Dis Colon Rectum , 2007, 50 (9) : 1342-1353 DOI:10.1007/s10350-007-277-7 |
| [25] | Rimkus C, Friederichs J, Boulesteix AL, et al. Microarray-based prediction of tumor response to neoadjuvant radiochemotherapy of patients with locally advanced rectal cancer[J]. Clin Gastroenterol Hepatol , 2008, 6 (1) : 53-61 DOI:10.1016/j.cgh.2007.10.022 |
| [26] | Nishioka M, Shimada M, Kurita N, et al. Gene expression profile can predict pathological response to preoperative chemoradio-therapy in rectal cancer[J]. Cancer Genom Proteom , 2011, 8 (2) : 87-92 |
 2016, Vol. 36
2016, Vol. 36


